Introducción
En la actualidad existen varias enfermedades altamente prevalentes en felinos domésticos, como sida, enfermedad producida por el virus de la inmunodeficiencia felina (VIF), un lentivirus que presenta afinidad por las células del sistema inmune especialmente la población CD4+ de linfocitos T14, otra afección es la leucemia causada por el virus de la leucemia felina (VLFe), un retrovirus que afecta las células del sistema inmune15. Los virus discutidos anteriormente atacan el sistema inmune del hospedero por lo cual la sintomatología en ambos casos es similar y puede llevar a un diagnóstico confuso, por lo tanto se debe corroborar con pruebas de laboratorio16, las más comunes son las basadas en la tecnología de ensayo por inmunoabsorción ligado a enzimas (ELISA), una metodología sencilla que obtiene resultados rápidos; sin embargo, estos métodos de diagnóstico presentan desventajas como la aparición de falsos positivos, por razones como reacciones cruzadas, anticuerpos maternos o vacunas37, y falsos negativos por falta de anticuerpos debido al corto tiempo de exposición al patógeno o por concentraciones bajas del antígeno evaluado17 , 19, además, no es posible detectar infecciones mixtas en la mayoría de los casos15 , 3. Por las desventajas de estas pruebas, se han realizado diferentes estudios para diagnosticar estos patógenos por medio de herramientas moleculares como la reacción en cadena de la polimerasa (PCR) o PCR en tiempo real para detectar un ADN copia a partir del ARN viral, permitiendo la diferenciación entre los virus31, detectar infecciones mixtas y cuantificar la carga viral7 , 24 , 25 , 31.
Una razón por la cual estas enfermedades son altamente prevalentes es debido a su trasmisión, ya que no involucra un vector, es causada por mordidas, contacto de mucosa, lactancia y mutuo acicalamiento3, por consiguiente, es necesario conocer el estado de los gatos para poder determinar un mejor tratamiento, definir métodos de limpieza y manejo de los individuos infectados para evitar la diseminación de estos virus. Dadas estas necesidades se han desarrollado pruebas diagnósticas con tecnologías moleculares, como una PCR anidada amplificando el gen pol en retrovirus y en comparación con pruebas serológicas encontraron una concordancia de 0,69 y 0,87 para leucemia y sida, respectivamente2, así mismo, Westman ME et al. realizaron un estudio de comparación entre tres pruebas serológicas para la detección de leucemia, debido a los resultados poco confiables y a una compleja relación huésped-patógeno utilizando como prueba de referencia una qPCR38. Por características como la detección directa del agente etiológico de la enfermedad3 , 7 , 16, la posibilidad de diagnosticar varios agentes en una única reacción24 y la viabilidad de cuantificar la carga viral16, hacen que la PCR múltiple en tiempo real sea conveniente para desarrollar un primer acercamiento en la estandarización de una metodología de diagnóstico que permita identificar los virus causantes de sida y leucemia en gatos domésticos en Colombia y suplir las desventajas presentadas por las pruebas de diagnóstico actuales15.
Materiales y Métodos
Se diseñó una PCR múltiple con sondas Taqman para diferenciar individuos sanos y aquellos que presenten infección por VIF, VLFe, o infecciones mixtas, además se incluyó la amplificación de un gen constitutivo del genoma del gato doméstico, como control positivo interno, con el propósito de verificar que los resultados son verdaderos y no producto de una incorrecta ejecución del experimento12.
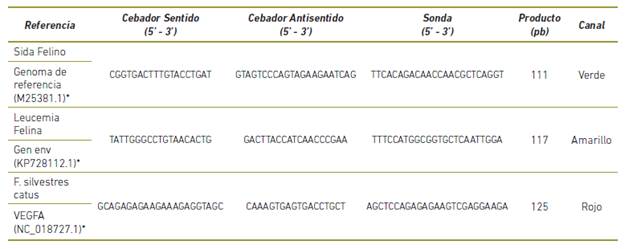
Los cebadores y sondas se crearon con el programa PrimerQuest de Integrated DNA Technologies26 con secuencias de referencia de la literatura (Tabla 1)10 , 32 y se verificó su especificidad en el programa Basic Local Alignment Search Tool (BLAST)1. Adicionalmente para el control positivo interno se realizó una simulación de PCR en el programa UCSC Genome Browser de la Universidad de California Santa Cruz13, que permite evaluar los cebadores y las sondas en el genoma completo de F. silvestris catus.
La toma de muestra fue realizada en la ciudad de Medellín y Envigado, avalada por el Comité Institucional para el Cuidado y Uso de los Animales de la Universidad CES (Cicua) con riesgo mínimo (Acta: 04, código del proyecto: N.32, fecha: 16 julio 2013). Un consentimiento informado de los tenedores fue utilizado antes de incluir los gatos en el estudio. Se formaron dos grupos cada uno con 50 individuos, grupo 1: casos, fueron establecidos previamente por signos clínicos compatibles con las patologías, exámenes hematológicos irregulares y prueba inmunológica positiva para sida o leucemia; grupo 2: controles, fueron establecidos previamente por signos clínicos incompatibles con las patologías, exámenes hematológicos normales y motivo de consulta ajeno a las enfermedades analizadas. Se tomaron entre dos a cuatro mL de sangre periférica en tubos con EDTA y se almacenaron a -20 ºC.
La obtención de ARN de las muestras se realizó con el kit GeneJET RNA Purification de (Thermo Scientific, Inc. USA) siguiendo las recomendaciones de la casa comercial, se tomaron 200µL de sangre periférica, se sometieron a un proceso de lisis celular suplementando con β-mercaptoethanol, purificación por medio de membranas de sílice y buffer de lavado, por último, una elución, finalmente se recupera el ARN en solución y se almacenaba a -20 ºC. El ARN se cuantificó por medio de espectrofotómetro usando el NanoDrop 2000 (Thermo Scientific, Inc. USA) y se evaluó su pureza por medio de la relación de absorbancia 260/280 nm. Adicionalmente se utilizó el kit First strand cDNA synthesis (Thermo Scientific, Inc. USA) para la conversión de ARN a ADN copia con cebadores no específicos incluidos en el kit, para el desarrollo del protocolo se siguieron las recomendaciones de la casa comercial. El ADN copia se cuantificó por medio de espectrofotómetro usando el NanoDrop 2000 (Thermo Scientific, Inc. USA) y se evaluó su pureza por medio de la relación de absorbancia 260/280 nm. El kit utilizado para la PCR fue QuantiTect Múltiple PCR NoROX (QIAGEN, Alemania) y la detección fue por medio de sondas Taqman en el termociclador en tiempo real Rotor Gene 6000 (Corbett Robotics, Inc. USA). Se incluyeron controles negativos (sin muestra de ADN copia) que fueron utilizados para calcular el umbral de detección para cada canal de amplificación, promediando los valores de la fluorescencia basal cada cinco ciclos de cinco PCRs diferentes, al determinar este valor, se ajustaba con un intervalo de confianza del 95% y basado en esta cantidad se determinaba que muestras eran positivas o negativas33.
La PCR múltiple se estandarizó para la detección de VIF y VLFe de forma simultánea bajo las siguientes condiciones 12,5 µL de 2x QuantiTect Multiplex PCR NoROX Master Mix, 0,67 µL de cada cebador y sonda a 10mM, ADN copia en una concentración mayor o igual a 10.000 ng en la reacción y agua libre de RNAsas hasta alcanzar un volumen de 25 µL. Las condiciones del ciclo fueron 94 ºC por 15 min (desnaturalización) seguido por 50 ciclos de 94 ºC por 60s (alineamiento cebadores), 59 ºC por 60 s (alineamiento sondas), 68 ºC por 35 s (elongación).
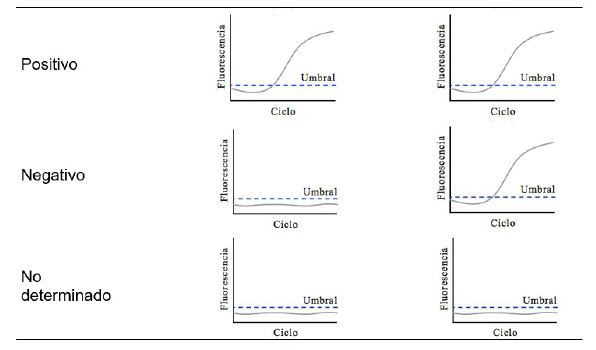
Para el análisis de la PCR múltiple se consideró como positiva una muestra que sobrepasara el umbral de detección en alguno de los canales de lectura de los virus y el control positivo interno, negativa una muestra que sobrepasara el umbral de detección únicamente en el canal del control positivo interno y no determinado una muestra que no sobrepasara el umbral de detección en ninguno de los canales (Figura 1). La concordancia entre los resultados de la PCR y la prueba serológica fue medida usando el coeficiente Kappa5 el cual se calculó mediante el paquete epiR23 con el software R28. Se estimaron la sensibilidad y la especificidad de la PCR usando la aproximación bayesiana descrita, entre otros, por Zhou39 y Broemeling4, la cual considera que la prueba de referencia contra la cual se compara no es un verdadero referente (Gold standard), como es el caso de la serología en este estudio. El análisis bayesiano fue ejecutado en el software WinBugs 1,4 empleando el código Bugs 7,2 de Broemeling4 considerando prioris no informativas. El análisis fue realizado con una cadena de 310.000 iteraciones descartándose las primeras 10.000. Se muestran la mediana e intervalos de credibilidad del 95% desde la distribución posteriori obtenida.
Resultados
De las muestras analizadas, once presentaron hemolisis o coagulación y luego de realizar diez ensayos de PCR múltiple se determinó que muestras con una concentración de ARN menor a 0,1 ng/µL no generaban resultados en los ensayos posteriores, por esta razón se descartaron cuatro muestras adicionales y se analizaron 44 muestras positivas de las cuales siete habían sido previamente diagnosticadas con sida, 37 con leucemia, una de estas presentaba infección mixta (Grupo 1: casos) y 41 muestras negativas (Grupo 2: controles).
Según los resultados de la PCR múltiple para determinar el VIF, cinco muestras superaron el umbral de detección calculado para este canal, por lo tanto, fueron consideradas positivas para este virus luego de verificar con PCR individual y electroforesis en gel de agarosa al 3% que la banda correspondía al tamaño del amplificado (111 pb) esperando (datos no mostrados).
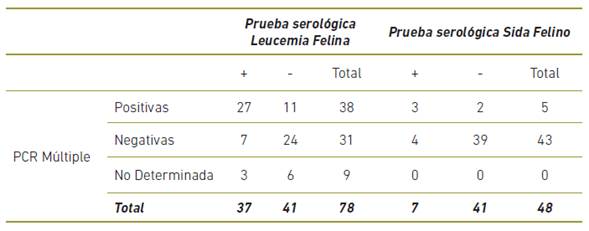
De las 78 muestras analizadas con PCR múltiple se obtuvieron 38 positivas, 31 negativas y nueve no determinadas para leucemia, y de las 48 muestras analizadas para sida se obtuvieron 5 positivas y 43 negativas. Dos muestras presentaban una infección mixta.
Según los resultados para sida, tres muestras fueron positivas y 39 negativas con ambas pruebas, seis presentaron discrepancia en el resultado, es decir, muestra determinada positiva con prueba serológica dieron negativas con la PCR múltiple o viceversa. Para leucemia 27 muestras fueron positivas y 24 negativas con ambas pruebas y 18 presentaron discrepancia en el resultado (Tabla 2). Los controles negativos no presentaron amplificación en ningún canal y la fluorescencia siempre estuvo por debajo del umbral de detección.
Al considerar estos datos y analizarlos teniendo como punto de referencia una prueba serológica de inmunoensayo para la detección de respuesta inmunológica del animal, la proporción de concordancia entre la prueba serológica y la PCR múltiple con respecto a leucemia, luego de excluir el azar es de 0,5 (95% CI 0,24 a 0,72) y de sida es de 0,43 (95% CI -0,15 a 0,70) esto representa un acuerdo bajo, lo cual significa que no existe una relación entre los resultados de las pruebas; sin embargo, al ser calculado con una prueba de referencia imperfecta se presenta un sesgo, por lo tanto los valores de sensibilidad (0,53/0,26) y la especificidad (0,46/0,74) para leucemia y sida respectivamente se determinaron con la metodología de Broemeling4 ) (Tabla 3).
Tabla 3 Resultados de sensibilidad y especificidad para la PCR múltiple y la prueba serológica de leucemia y sida felino obtenidos de acuerdo a análisis bayesiano.
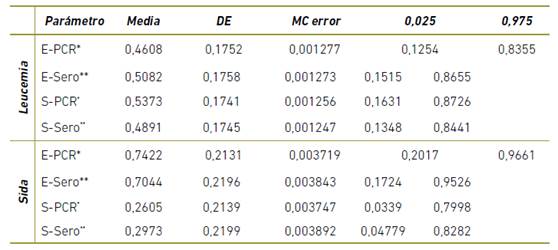
*Especificidad para la PCR múltiple.
**Especificidad para la prueba serológica.
*Sensibilidad para la PCR múltiple (S-PCR).
**Sensibilidad para la prueba serológica.
DE: Desviación estándar.
Discusión
Durante la estandarización de la técnica el paso de ARN a ADN copia generaba altas concentraciones; sin embargo, las muestras con una cantidad menor a 0,1 ng/µL de ARN no cumplían la relación de absorbancia 260/280 nm presentando inconvenientes en el desarrollo de la PCR múltiple, por lo cual fueron descartadas. Las bajas concentraciones de ARN se atribuyen a errores en la fase pre analítica y se considera que la metodología de toma de muestra debe ser modificada, debido a errores encontrados durante el proceso, principalmente hemolisis y coagulación34, por esta razón 11 muestras fueron descartadas antes de iniciar el proceso de extracción; sin embargo, este problema en el procedimiento también puede ser el causante de los resultados no determinados con la PCR múltiple, ya que la concentración y calidad de ARN y posteriormente de ADN copia se ve directamente afectada por la condición de la muestra29.
Los resultados de la PCR múltiple fueron determinados basados en el umbral de detección calculado para cada canal, debido a que no es posible detectar el límite de detección de la prueba, ya que, se analizaron muestras clínicas y no se conocía la proporción de ARN de felino y de los virus en las muestras22, por esta razón es necesario analizar controles de laboratorio o muestras simuladas para incluir este parámetro de validación de la PCR múltiple27) y poder estimar la carga viral en las muestras clínicas. A pesar de que no fue posible calcular esta variable, la PCR múltiple en tiempo real es más sensible y posibilita el diagnóstico de varias enfermedades en una única prueba7.
En los resultados obtenidos no fue posible observar una curva exponencial de amplificación para las muestras positivas para sida con la PCR múltiple, lo cual puede deberse a que la cantidad de ADN copia presente en la muestra correspondiente al virus, es insuficiente para alcanzar el crecimiento exponencial y verse reflejado en la curva (Figura 2)30; no obstante, cinco muestras superaron el umbral de detección y se determinaron como positivas luego de corroborar este resultado con PCR individual (Datos no mostrados).
La cantidad de muestras positivas para sida es baja; sin embargo, según la referencia más cercana existente, reportado por Salim Mattar Velilla et. al en un estudio en Montería, Cordoba36, se presenta una mayor prevalencia para leucemia, tanto por diagnóstico serológico3 , 17 como molecular25, lo cual es consistente con lo reportado en esta investigación. Es importante resaltar que el paso de ARN a ADN copia, no se realiza con cebadores específicos de los virus, sino con cebadores aleatorios incluidos en el kit First strand cDNA synthesis (Thermo Scientific, Inc. USA), que sintetizan ADN copia de todos los ARN presentes en la muestra, igualmente por esta razón es necesario usar grandes cantidades de ADN copia en la reacción de la PCR múltiple, por lo cual desarrollar cebadores específicos para la síntesis de ADN copia podría mejorar la eficiencia29 , 30. Además, se debe considerar que la prueba de referencia detecta la respuesta inmunológica del animal sugiriendo que este puede presentar una infección por el virus causante de sida, no que el agente etiológico se encuentre en el hospedero11, lo cual está relacionado con la baja concordancia entre las pruebas de detección de los virus.
De 13 muestras con resultado serológico negativo, dos fueron positivas para sida y 11 para leucemia con la PCR múltiple (Figura 3), esto se relaciona con la respuesta inmune del hospedero que puede tomar hasta 60 días o más para sida y 30 días para leucemia, generando un resultado negativo ya que no supera el límite de detección determinado para la prueba serológica17 , 21, adicionalmente seis de los individuos, convivían con gatos que estaban infectados (corroborado con prueba serológica y PCR múltiple), lo cual aumenta la probabilidad de ser positivos, ya que la transmisión de estos virus no involucra un vector, sino el contacto con animales portadores3 , 16 y en ninguno de los casos los gatos domésticos se encontraban aislados, según información de los propietarios; además, los animales portadores se incluyeron en este estudio y certificaron su estado positivo tanto con la prueba serológica como con la PCR múltiple. Siete muestras con resultado serológico positivo para leucemia, fueron negativas con análisis molecular, lo cual puede ser atribuido a tratamientos que impiden la replicación de virus en el hospedero, disminuyendo la carga viral y no la producción del antígeno detectado por la prueba serológica en el individuo infectado8 , 20.
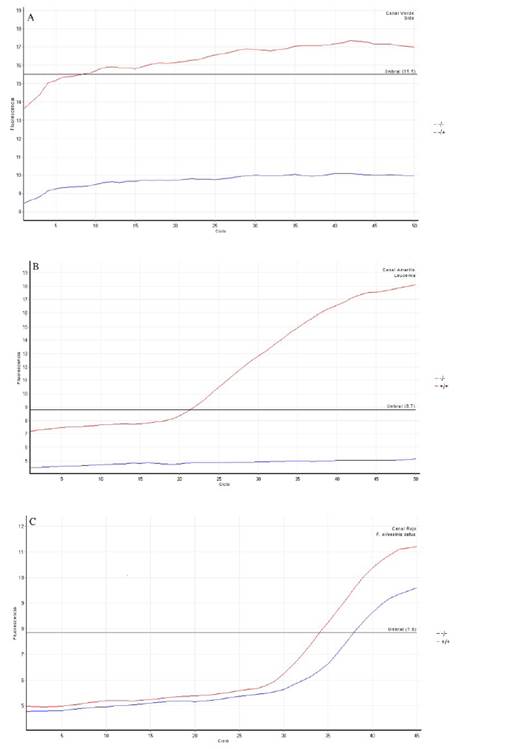
Figura 2 Resultados PCR múltiple, donde se observa la fluorescencia (Eje Y, dF/dT) en relación con el ciclo (Eje X); se presentan dos muestras (línea roja y azul) en los canales correspondientes a la amplificación de sida (A), leucemia (B) y de F. silvestris catus (C), al lado derecho se presenta la relación de los resultados con los dos mé todos de diagnóstico (Prueba serológica/PCR múltiple)
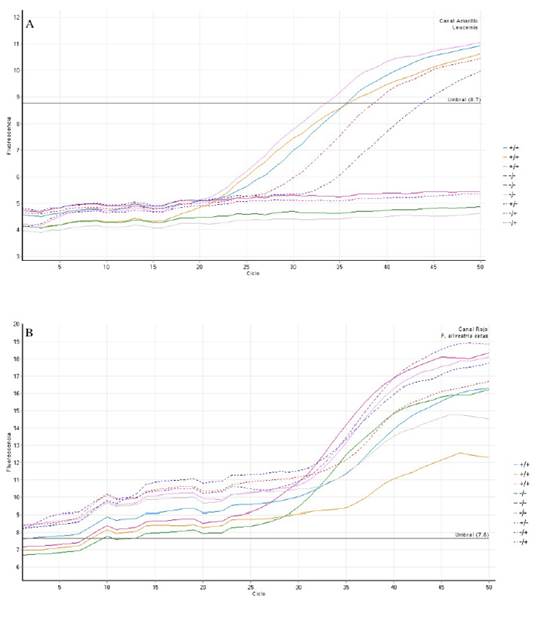
Figura 3 Resultados PCR múltiple para leucemia, donde se observa la fluorescencia (Eje Y, dF/dT) en relación con el ciclo (Eje X); se presentan nueve muestras en los canales correspondientes a la amplificación de leucemia (A) y de F. silvestris catus (B), al lado derecho se presenta la relación de los resultados con los dos métodos de diagnóstico (Prueba serológica/PCR múltiple)
Según los resultados obtenidos con la estimación de Broemeling4, los datos presentan una alta incertidumbre como se puede observar por la amplitud de los intervalos de credibilidad, lo cual resulta de un tamaño muestral bajo, y de no contar con información anterior que mejore la certeza de los datos, estos valores no son concluyentes para definir la veracidad de la PCR múltiple. Un caso similar lo reportó Mathevon18 obteniendo resultados de sensibilidad y especificidad bajos al evaluar una PCR cuantitativa utilizando el método bayesiano, sin embargo, al presentar un tamaño muestral significativo obtuvieron unos intervalos de confianza más exactos, pero de igual forma concluyeron que el método molecular era más apto para realizar diagnóstico. Adicionalmente, diferentes estudios han demostrado que las técnicas moleculares son más sensibles y específicas que las pruebas serológicas2 , 6 , 7 , 9 , 18 , 35 ya que son pruebas directas, detectan ADN, ARN, proteínas estructurales y no estructurales propias del organismo que se está identificando, por el contrario, las metodologías serológicas son indirectas ya que detectan la respuesta inmune del hospedero3 , 17.
Conclusiones
La PCR múltiple desarrollada en este estudio fue validada de forma in silico y presentó resultados congruentes al realizarla in vivo, es una herramienta para la detección de sida y leucemia en el gato doméstico; los cebadores y sondas desarrollados en esta investigación fueron estandarizados y fue posible la detección de los virus causantes de leucemia y sida felina en muestras de sangre periférica, sin embargo, se requieren estudios adicionales para determinar el límite de detección de la PCR múltiple. Adicionalmente, es necesario aumentar el número de muestras, determinar información que mejore la certeza de los datos o cambiar la prueba de referencia para tener un mejor estimativo de la sensibilidad y especificidad de la PCR múltiple.