Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Producción + Limpia
Print version ISSN 1909-0455
Rev. P+L vol.10 no.2 Caldas July/Dec. 2015
Bacterias resistentes a antibióticos en ecosistemas acuáticos*
Antibiotic resistant bacteria in aqueous ecosystems
Bactérias resistentes a antibióticos em ecossistemas aquáticos
Rosa Leonor Acevedo Barrios**, Carlos Alberto Severiche Sierra***, José Del Carmen Jaimes Morales****
* Artículo de revisión derivado del proyecto de investigación:"Aislamiento de bacterias resistentes a antibióticos en playas y suelos salinos¨, realizada en la Región Caribe de Colombia, durante el segundo semestre del año 2014.
** Doctoranda en Toxicología Ambiental, Magíster en Microbiología, Bióloga. Docente de Tiempo Completo de la Universidad Tecnológica de Bolívar. Cartagena, Colombia. rosautb@gmail.com
*** Magíster en Desarrollo Sostenible y Medio Ambiente, Especialista en Ingeniería Sanitaria y Ambiental, Químico, Doctorante en Ciencias. Docente Investigador de la Universidad de Cartagena, Docente Investigador de la Universidad Tecnológica de Bolívar. Cartagena, Colombia. cseveriches@gmail.com
**** Magister en Ingeniería, Magister en Ciencia y Tecnología de Alimentos, Especialista en Ciencia y Tecnología de Alimentos, Ingeniero de Alimentos, Licenciado en Biología y Química, Doctorante en Ciencias. Docente Investigador de la Universidad de Cartagena. Cartagena de Indias, Colombia. jjaimesmor@yahoo.es
Autor correspondencia: Rosa Leonor Acevedo Barrios, email: rosautb@gmail.com
Artículo recibido: 19/03/2015; Artículo aprobado: 11/12/2015
Resumen
En la presente revisión, se detalla la temática de bacterias resistentes a antibióticos en ecosistemas acuáticos, caracterizando el panorama de la resistencia bacteriana a los antibióticos, se hace la discusión de la difusión de los antibióticos en el medio ambiente, estudiando las fuentes de antibióticos en los ecosistemas acuáticos y por último se analizan los efectos ambientes de la contaminación por bacterias resistentes. Los antibióticos son probablemente la familia más exitosa de fármacos desarrollados hasta fecha para mejorar la salud humana, para prevenir y tratar infecciones. Se encontró, que el uso y la venta de ellos en todo el mundo reveló que en general, existe una falta sistemática de datos, que permita una intervención eficaz y la propuesta de estrategias de mitigación a la problemática ambiental de las bacterias resistentes a antibióticos; existe una marcada relación entre los residuos de antibióticos en ecosistemas y el aumento de bacterias resistentes a ellos en el medio ambiente; pero en la actualidad, no hay información suficiente para llegar a una conclusión definitiva sobre la importancia y el impacto de la presencia de estas bacterias que permita la evaluación de los riesgos potenciales en los ecosistemas y la salud humana. Se propone la hipótesis que la utilización de antibióticos para fines distintos de la terapia antibiótica puede enriquecer la población de bacterias resistentes capaces de infectar a los seres humanos.
Palabras clave: contaminación, fármacos, salud, toxicología.
Abstract
In this revision the subject of antibiotic resistant bacteria in aqueous ecosystems is analyzed in detail, characterizing the outlook of bacterial resistance to antibiotics. A discussion on the dissemination of antibiotics in the environment is made, studying the sources of antibiotics in aqueous ecosystems and, finally, the environmental effects of antibiotic resistant bacteria contamination are analyzed. Antibiotics are probably the most successful family of drugs developed to improve human health by preventing and treating infections, but their worldwide use and sale reveal that, in general, there is a systematic failure in data to achieve an effective intervention and the proposition of strategies against the environmental problems brought by antibiotic resistant bacteria. Besides, there is a close relationship between the remains of antibiotics in ecosystems and the increase of antibiotic resistant bacteria, but at the moment the information available is not enough to achieve a definite conclusion concerning the importance and the impact of the presence of such bacteria in aqueous environments that allow the assessment of their potential hazardousness in the ecosystems and for human health. The hypothesis that the use of antibiotics for purposes different than antibiotic therapy can increase the population of antibiotic resistant bacteria able to affect human beings and the biota in general, is also proposed.
Key words: contamination, drugs, health, toxicology.
Resumo
Na presente revisão, se detalha a temática de bactérias resistentes a antibióticos em ecossistemas aquáticos, caracterizando o panorama da resistência bacteriana aos antibióticos; se faz a discussão da difusão dos antibióticos no meio ambiente, estudando as fontes de antibióticos nos ecossistemas aquáticos e, por último, se analizam os efeitos ambientes da contaminação por bactérias resistentes. Os antibióticos são provavelmente a família mais exitosa de fármacos desenvolvidos até a data para melhorar a saúde humana, para prevenir e tratar infecções. Se encontrou que o uso e a venda deles em todo o mundo revela que, em geral, existe uma falta sistemática de dados, que permita uma intervenção eficaz e a proposta de estratégias de mitigação à problemática ambiental das bactérias resistentes a antibióticos; ademais, há uma marcada relação entre os resíduos de antibióticos em ecossistemas e o aumento de bactérias resistentes a eles no meio ambiente; mas na atualidade, não existe informação suficiente para chegar a uma conclusão definitiva sobre a importância e o impacto da presença destas bactérias no meio ambiente aquático que permita a avaliação dos seus riscos potenciais nos ecossistemas e na saúde humana. Se propõe a hipótese que a utilização de antibióticos para fins distintos da terapia antibiótica pode enriquecer a população de bactérias resistentes capazes de infectar aos seres humanos e a biota em geral.
Palavras chave: contaminação, fármacos, saúde, toxicologia.
Introducción
Desde el descubrimiento de los antibióticos por Flemming en 1928, estos son probablemente la familia más exitosa de fármacos desarrollados hasta fecha para mejorar la salud humana (antimicrobianos en general); para prevenir y tratar infecciones de animales y plantas, en la ganadería se usan como promotores del crecimiento y suplemento alimenticio (Ayokunle & Ahmad, 2013). Pero el mal uso de los antimicrobianos en la clínica, en medicina veterinaria y en la agricultura han dado lugar a fuertes presiones sobre las comunidades de bacterias ambientales y a la difusión de sus genes de resistencia; así como de agentes selectivos (antibióticos, biocidas, contaminantes ambientales como metales pesados, entre otros) que se mueven entre los diferentes ecosistemas y ejercen sus efectos (Acevedo & Severiche, 2013; Mostafa et al., 2011).
Uno de los principales efectos incluyen la toxicidad aguda o crónica de especies de plantas y animales expuestos a estos fármacos, debido principalmente a que la mitad de los antibióticos administrados a los seres humanos y los animales se excreta en forma activa sin cambios principalmente a través de la orina y heces fecales. La literatura reporta una absorción incompleta hasta del 95 % de los antibióticos administrados y la producción de sus metabolitos,estos últimos pueden ser más tóxicos que el compuesto original y pueden llegar a acumularse en todas las matrices ambientales, principalmente las aguas superficiales, subterráneas, suelos y sedimentos (Ajit et al., 2006; Durso et al,. 2013; Acevedo & Severiche, 2013).
Otro efecto es el aumento significativo de la aparición de "superbacterias", resistentes a los antibióticos y la tolerancia humana o de animales a estos medicamentos, sumado a esto, estos fármacos puede llegar afectar la salud de los seres vivos, por esto son considerados contaminantes emergentes y grandes amenazas potenciales para los ecosistemas; lo que ha llevado a la comunidad científica a interesarse por el estudio sobre el mal uso de los antibióticos en la identificación de cómo sus propiedades fisicoquímicas y toxicológicas pueden llegar a producir graves efectos en los ecosistemas acuáticos (Baquero et al. 2008; Storteboom et al., 2010; Ayokunle & Ahmad, 2013). Pero poco se conoce sobre los efectos globales de estos fármacos en la dinámica poblacional de las bacterias presentes en los cuerpos de aguas naturales (Mostafa et al., 2011; Moore et al. 2010); debido a que la mayoría de la información reportada es para entornos clínicos (Graham et al. 2011; Levin et al. 2011; Zheng et al. 2011; Martínez, 2010; Moore et al. 2010; Bécares et al., 2011).
El aumento mundial de las bacterias resistentes a los antibióticos en el medio ambiente hace que sea imperativo la vigilancia de la presencia y distribución de las bacterias y de genes de resistencia; procedentes de la clínica al medio ambiente, problemática que no se limita a las aguas superficiales sino también a las aguas subterráneas; donde también se han aislado bacterias multirresistentes (Graham et al., 2011; Levin et al., 2011; Wright 2010); es por esto que se debe profundizar en el conocimiento sobre la ocurrencia, el destino y el transporte de antibióticos y sus metabolitos en los ecosistemas acuáticos, para comprender la relación existente entre los residuos de antibióticos después de su excreción, sus metabolitos y las poblaciones de bacterias resistentes a ellos (Peng et al., 2013; Riszzo et al., 2013).
La resistencia bacteriana es un proceso inevitable y la comprensión de sus orígenes, su evolución y la difusión de elementos de resistencia a antibióticos proporciona una información vital para el descubrimiento de nuevos medicamentos, ayudando a guiar las investigaciones de estos compuestos, para la identificación y la selección de nuevos candidatos importantes en los ensayos clínicos, así como búsqueda de alternativas de mitigación de los efectos de estos fármacos en las poblaciones bacterianas presentes en el medio ambiente acuático (Parisien et al., 2008). Por lo anterior, se hace una revisión bibliográfica sobre la temática, bacterias resistentes a antibióticos en ecosistemas acuáticos, detallando el panorama de la resistencia bacteriana a los antibióticos, se trata la difusión de los antibióticos en el medio ambiente, seguidamente se estudia las fuentes de antibióticos en los ecosistemas acuáticos y por último se analizan los efectos ambientales de la contaminación por bacterias resistentes a antibióticos.
Panorama de la resistencia bacteriana a los antibióticos
Las bacterias potencialmente patógenas se liberan constantemente en las aguas, muchas de ellas albergan genes de resistencia a antibióticos que se insertan en plataformas genéticas móviles (plásmidos, transposones e integrones) capaces de propagarse entre las comunidades bacterianas que viven en el agua y en el suelo (Venegas et al., 2009). El agua constituye la ruta principal por la cual se introducen genes de resistencia bacterianos en los ecosistemas naturales (Storteboom et al., 2011), en donde las bacterias no patógenas pueden servir como reservorio de genes de resistencia. Esto acompañado de la introducción y la acumulación progresiva en el agua de agentes antimicrobianos como detergentes, desinfectantes, residuos de contaminación industrial (metales pesados) contribuyen a una mayor propagación y evolución de mecanismos de resistencia de las bacterias autóctonas presentes en el medio ambiente acuático (Baquero et al., 2008).
La problemática de la resistencia a antibioticos se asemeja en algunos aspectos a la contaminación por metales pesados. Al igual que los antibióticos, los metales pesados son componentes naturales presentes en los diferentes ecosistemas. Sin embargo, su utilización por los seres humanos ha aumentado su biodisponibilidad, lo que conduce a cambios dramáticos en las poblaciones bacterianas presentes en los ecosistemas contaminados (Acevedo & Severiche, 2013; Volles & Branan, 2008). A diferencia de los metales pesados que afectan todas las formas de vida, los antibióticos alteran fundamentalmente a los microorganismos, probablemente debido a esto, las consecuencias de la contaminación de estos farmacos en la biodiversidad han recibido menos atención que la contaminación causada por los metales pesados (Martínez et al., 2010; Martinez, 2009).
Comprender la resistencia a metales pesados en los ecosistemas naturales puede ayudar también a entender la resistencia a antibióticos en el medio ambiente (Acevedo & Severiche, 2013). Los elementos que intervienen en la resistencia a los metales pesados están codificados en los cromosomas de bacterias que pueden estar bien adaptados para sobrevivir en los hábitats ricos en metales pesados naturalmente (por ejemplo, suelos volcánicos). Sin embargo, una fuerte presión selectiva debido a la contaminación antropogénica, conlleva a la transferencia de genes que pueden propagar la resistencia de forma eficiente entre las poblaciones bacterianas. (Wellington et al., 2013). Del mismo modo, los genes de resistencia a antibióticos que estaban presentes de forma natural en los cromosomas de las bacterias del medio ambiente ahora están presentes en plásmidos que se pueden transferir a los patógenos humanos. Se ha puesto de manifiesto que el contacto de las bacterias de la microbiota humana asociada con microorganismos ambientales en plantas de aguas residuales o en los ecosistemas naturales es una característica importante para entender la aparición de nuevos mecanismos de resistencia en patógenos humanos (Martinez, 2009;Akiyama & Savin, 2010).
Las bacterias y los genes de resistencia a antibióticos se encuentran en todo el mundo, sin embargo la amplia difusión de ellos indica que su presencia en ecosistemas naturales puede ser alta (Zheng et al., 2011). Por esta razón, los genes de resistencia a antibióticos están siendo considerados como los propios contaminantes. Puesto que ellos se encuentran naturalmente en los cromosomas de las bacterias del medio ambiente. La contaminación por antibióticos es relevante, porque la liberación de los antibióticos, junto con las bacterias resistentes puede afectar también la microbiota ambiental (Chang et al., 2012; Muñoz et al., 2007).
La resistencia es un proceso natural conservado evolutivamente. Un factor que contribuye a la capacidad de adaptación es que las bacterias tienen acceso a un gran grupo de genes que se mueven por la transferencia horizontal y vertical y se extienden a través de las poblaciones bacterianas en elementos genéticos móviles como los plásmidos y trasposones (Argemi et al., 2005). La resistencia a los antibióticos se cuantifica por lo general como la concentración mínima necesaria para producir un efecto definible. Los diferentes mecanismos de acción y los métodos utilizados para evaluar la susceptibilidad son cruciales para los resultados de las pruebas de sensibilidad y la evaluación de la resistencia (Kümmerer, 2009).
Las biopelículas son consideradas una barrera taxonómica para la transferencia horizontal de material genético y una fuente importante en la aparición de bacterias resistentes en el medio ambiente, la concentración de antibióticos, la alta densidad bacteriana y una larga exposición; es suficiente para promover la resistencia en bacterias (Farkas et al., 2013).
La concentración de los antibióticos puede ser mucho mayor si los compuestos activos son persistentes y se acumulan por adsorción en las superficies sólidas como los lodos, sedimentos o suelos. No se conoce totalmente bajo qué circunstancias los antibioticos están biológicamente activos después de la sorción (Feng et al.,2006).Los que indica que la entrada de bacterias resistentes en el medio ambiente, pueden ser aun más importantes que la presencia de los compuestos activos de antibióticos en el medio ambiente. Muy poco se ha reportado para aclarar el papel de las biopelículas bacterianas en ambientes acuáticos y su papel en la resistencia a los antibióticos (Fenton, 2006; Baquero et al., 2008).
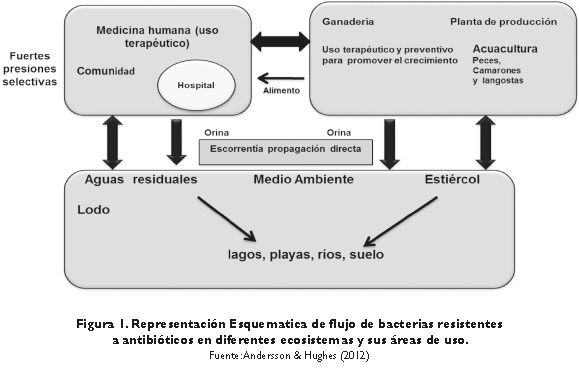
Las bacterias resistentes a los antibióticos se liberan en las aguas a través de la orina, heces, eventualmente cadáveres, estiércol, que usan como ruta principal las aguas residuales de los hospitales, efluentes municipales, de la industria ganadera y piscícola se que constituyen probablemente en las mayores fuentes de bacterias y de genes de resistencia a antibióticos que se liberan en el medio ambiente. Por tanto es prioritario investigar sobre cuales son las barreras adecuadas que impidan su incorporación en el medio ambiente (Baquero et al., 2008). El uso intensivo de compuestos químicos persistentes, aumenta la presión selectiva en bacterias, facilitado la transferencia de la resistencia a antibióticos entre las comunidades de bacterias en ambientes acuáticos que producen efectos adversos en peces, invertebrados, tortugas, sedimentos marinos, entre otros (Figura 1) (Anderson & Hughes, 2012; Ozaktas et al. 2012).
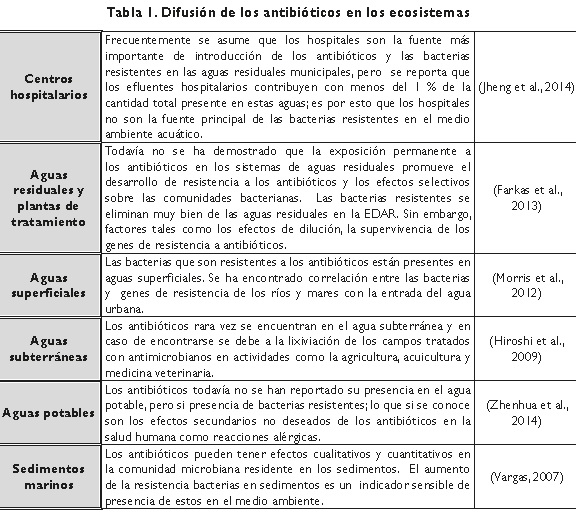
Estudios recientes indican que más del 90 % de las cepas bacterianas marinas son resistentes a más de un antibiótico, y 20 % son resistentes al menos a cinco. Lo más preocupante es que se ha identificado resistencia a antibióticos y a metales en lugares remotos como los glaciales; en donde la acción antropogénica es poca (Iannacone & Alvariño, 2009). La asociación de resistencia a los antibióticos y la resistencia a los metales pesados es muy frecuente en el mismo organismo (también en el mismo plásmido, transposón, o integrón). Dado que los estudios han demostrado que estos compuestos son transportados en el agua superficial y el agua subterránea de fuentes urbanas y agrícolas, es por esto que los investigadores han comenzado a realizar estudios de sus efectos. Sin embargo, hay una escasez de datos sobre el destino y el comportamiento de los compuestos en el suelo-agua (Kümmerer, 2009). En la tabla 1, se hace una caracterización de ecosistemas para la difusión de bacterias resistentes a antibióticos.
Fuentes de antibióticos en el medio ambiente acuático: La exposición a los antibióticos en las aguas superficiales puede tener efectos adversos en la reproducción en las primeras etapas de vida de los diferentes organismos (Vargas, 2007). En la tabla 2, se cita el origen de bacterias resistentes a antibioticos, algunos autores describen la posibilidad de un daño considerable al equilibrio natural, ya que los organismos constituyen el alimento para otros animales acuáticos, y por lo tanto su desaparición afecta a otros organismos, así la presencia de los antibióticos en el medio ambiente también pueden afectar el comportamiento de los organismos acuáticos. Tal como el caso de peces como deformaciones del esqueleto, tortugas con deformidades en sus caparazones que han estado expuestos a altas concentraciones de antibióticos en el medio ambiente.
Efectos ambientales de la contaminación por bacterias resistentes a antibióticos: Si una sustancia que no se elimina de ninguna manera se puede alcanzar el medio ambiente con el potencial de afectar adversamente a los organismos acuáticos y terrestres (Kümmerer, 2009; Yangali et al. 2010).
La contaminación por los genes de resistencia a antibióticos puede aumentar las posibilidades de patógenos humanos para la adquisición de la resistencia. Se ha propuesto que la liberación de los residuos de los hospitales que contienen comensal humano y bacterias infectivas (resistentes y susceptibles), así como antibióticos, se debe reducir a un mínimo para evitar el intercambio de material genético (incluso en plantas de tratamiento de aguas residuales. Según lo declarado por Baquero y Colaboradores en 2008, el contacto de la microbiota humana con otro tipo de microbiota en diferentes ecosistemas aumentará la posibilidad de la variación genética y la posible de aparición de nuevos mecanismos de resistencia que se vuelven a introducir en el entorno humano (Torreblanca & López, 2005; Baquero et al., 2008).
La propagación de genes de resistencia en los ecosistemas naturales puede desafiar la dinámica poblacional y la fisiología de las poblaciones microbianas naturales (Parisien et al. 2008). Varios informes indican que los genes de resistencia presentan actualmente en la microbiota asociada humano o animal se encuentran en entornos sin contaminación de antibióticos. En varias ocasiones, los genes se encuentran en bacterias con origen humano/animal (Wellintong et al. 2013). Sin embargo, aunque no se ha analizado en detalle, también es posible que esos genes se extiendan entre las poblaciones bacterianas ambientales. La pregunta aquí es si la incorporación de genes de resistencia a antibióticos puede producir cambios relevantes en los organismos receptores. Algunos trabajos indican que este puede ser el caso (Roman & Alfaro 2005; Martinez, 2009; Acuña et al., 2011).
Por lo tanto, es de suma importancia que se implementen estrategias para reducir la tasa de aparición y la propagación de bacterias resistentes a permitir descubrir nuevos fármacos para evitar el aumento de la resistencia bacteriana.
¿Cuál es el impacto esperado de una mayor tasa de mutación en la tasa de desarrollo de resistencia? Esta pregunta es parte del problema más general de la determinación de las condiciones en que un aumento de la tasa de mutación podría acelerar la adaptación (es decir, cuando no va en beneficio de una más rápida generación de mutaciones beneficiosas son mayores que el costo de la generación de mutaciones deletéreas) (Santos et al., 2010). La respuesta es compleja y depende de varios factores: el tamaño de la población bacteriana, la estabilidad y la estructura del medio ambiente, lo cerca que la población es máxima a la adaptación y si uno considera el destino a corto o largo plazo de las bacterias con el aumento de las tasas de mutación.
Estas consideraciones sugieren que si en una comunidad de bacterias resistentes a los antibióticos se han subido a una alta frecuencia que es probable que permanezcan allí por mucho tiempo. Estos resultados subrayan la necesidad de estrategias para reducir el desarrollo de resistencia (por ejemplo, un uso más restringido y prudente de los antibióticos, la vigilancia de la intensificación, mejorado y sostenido control de infecciones de trasmisión) para prolongar la vida útil de nuestros antibióticos disponibles. Además, hay una necesidad de utilizar nuevos antibióticos con objetivos y modos de acción nuevos. Dado que el costo biológico de la resistencia es un parámetro importante en la determinación de la velocidad y la estabilidad de desarrollo de resistencia, que valdría la pena dejar máxima compensación económica mínima sea un principio rector en la búsqueda de posibles nuevos objetivos farmacológicos.
Los estudios han demostrado que los cambios generalizados a la microbiota humana han resultado de tratamiento con antibióticos y que las cepas resistentes pueden persistir durante años. La eficacia de estos compuestos contra la vida se deriva de su capacidad de bloquear los procesos celulares bacterianas críticos. Dado que estos objetivos celulares son a menudo genética y estructuralmente conservados a través de diversos miembros del reino bacteriano, el despliegue de un compuesto antibiótico contra una bacteria específica, es bastante probable para impartir los daños colaterales a la comunidad bacteriana que comparte el medio ambiente del destinado organismo objetivo (Ozaktas et al., 2012). El espectro de efectos no deseados de la terapia con antibióticos es probable que sea considerablemente mayor que la inhibición del crecimiento sencilla de cepas sensibles, especialmente teniendo en cuenta el papel de estos compuestos orgánicos bioactivos como multi-actividad de las moléculas de señalización (Yangali et al., 2010; Brown et al., 2006; Igbinosa & Okoh, 2012).
La difusión de los genes de resistencia a antibióticos requiere la transferencia física o adquisición de la resistencia a los antibióticos como elemento de codificación genética. Los mecanismos responsables de esta transferencia son conjugación, transformación, transducción o, todos los cuales son completamente capaces de transferencia de determinantes de resistencia a antibióticos in vitro a partir de organismos donantes resistentes a los organismos receptores susceptibles (Vaneechoutte et al., 2004; Román & Alfaro, 2005 ).
En primer lugar la presión selectiva, los antibióticos en los ecosistemas naturales pueden seleccionar la integración y la difusión de los genes de resistencia a antibióticos en las unidades de transferencia de genes, que se pueden entonces considerarse contaminantes (Yangali et al. 2010).
En segundo lugar, los residuos de hospitales, casas y granjas contienen bacterias que pueden llevar a los determinantes de resistencia a antibióticos (Hiroshi et al., 2009). El hallazgo de genes de resistencia a antibióticos específicos, que ya se difunden entre los humanos, animales o plantas o agentes patógenos bacterianos (comensales) será un buen indicador de la historia de contaminación. A diferencia de la situación con antibióticos, esta contaminación no es necesariamente local, no depende de la liberación constante de los residuos, ya que una vez que esos genes se encuentran en el medio ambiente, pueden difundirse entre diferentes especies bacterianas y hábitats distintos. Se ha demostrado que los genes de resistencia a antibióticos pueden migrar entre los sistemas acuáticos conectados. No está claro si la presencia de genes de resistencia a antibióticos es el resultado de la migración de las bacterias resistentes a los antibióticos o la transmisión de genes de resistencia (Zenhua et al., 2014). Las actividades de transporte y comerciales en todo el mundo están ayudando así a la difusión de las bacterias, incluso entre distintos océanos y continentes. Por esta razón, los genes de resistencia a antibióticos en primer lugar reportados en los patógenos humanos, se encuentran así en varios hábitats diferentes, incluyendo aquellos en los que la contaminación con antibióticos es muy baja o incluso nula.
El hecho de que las poblaciones humanas remotas con la exposición a antibióticos mínima llevan bacterias comensales resistentes a los antibióticos apoyar aún más la difusión a escala mundial de genes de resistencia (Pollard & Woodley, 2007; Martinez, 2009; Segura et al., 2011).
Una cuestión clave para predecir el destino de los genes de resistencia a antibióticos en los ambientes naturales es la comprensión de sus efectos sobre la fisiología bacteriana. Se ha supuesto que la resistencia a antibióticos confiere una carga metabólica de las bacterias resistentes para que puedan estar fuera de competencia por sus homólogos de tipo salvaje en ausencia de presión selectiva antibiótico. Aunque en algunas ocasiones esto es probable que así sea, algunos ejemplos demuestran que la resistencia a los antibióticos puede permanecer en ausencia de selección de antibióticos. Entre los diferentes mecanismos implicados, algunas preocupaciones bacterias fisiología y algunos otros de la estructura de las plataformas genéticas implicadas en la transferencia de los determinantes de resistencia a antibióticos (Martinez, 2009).
Genes de resistencia a antibióticos pueden ser agrupados en la misma unidad con otros elementos que pueden proporcionar una ventaja ecológica. Los integrones son estructuras modulares con un papel importante en el desarrollo de resistencia a los antibióticos. Sus estructuras permiten la formación de organiza de genes de resistencia a antibióticos que se transfieren simultáneamente de tal manera que la selección por un co-selecciona otros genes de resistencia a los antibióticos presentes en el integrón. Los mismos tipos de integrones se encuentran en muestras de humanos, animales y hábitats naturales, lo que indica que los elementos gene-reclutamiento pueden recorrer a través de los diferentes ecosistemas (Aguilera et al., 2004; Brown et al., 2006; De la Torre et al., 2012).
La utilización de antibióticos con fines clínicos o la agricultura selecciona microorganismos resistentes. Por tanto, es predecible que los residuos de hospitales o granjas contendrán ambos tipos de contaminantes: antibióticos y genes de resistencia. Sin embargo, el destino de ambos tipos de contaminantes es probablemente diferente. Varios antibióticos son compuestos naturales que han estado en contacto con la microbiota ambiental durante millones de años por lo que son biodegradables. Sin embargo, todavía son degradados a diferentes velocidades en entornos naturales. Se ha demostrado que la ciprofloxacina presente en muestras de agua del río es completamente degradada después de 3 meses, mientras que sólo el 20 % de ácido oxolínico presente en estas muestras se degrada después de cinco meses.
El hecho de que los antibióticos son degradados en ecosistemas naturales no significa que no sean contaminantes persistentes. Algunos ecosistemas sufren una constante liberación de los antibióticos (por ejemplo, efluentes hospitalarios, residuos de granjas), de modo que sean siempre contaminadas con independencia de la degradación de los antibióticos (Martinez, 2009).
Para mitigar el efecto que la liberación de los antibióticos para el uso no humano puede tener para la selección de resistencia en los patógenos humanos, se prohibió la alimentación del ganado con antibióticos y su uso como factor de crecimiento, muchos países han restringido el uso de antibióticos en la acuicultura, incluyendo fuertes restricciones en el uso de la profilaxis con antibióticos y la automedicación en el tratamiento de las infecciones humanas.
Se puede concluir que, los datos sobre el uso y la venta de antibióticos en todo el mundo revelaron que en general, existe una falta sistemática de datos, que permita una intervención eficaz y la propuesta de estrategias de mitigación a la problemática ambiental de las bacterias resistentes a antibióticos. Si existe una relación marcada entre los residuos de antibióticos y el aumento de bacterias resistentes a ellos en el medio ambiente; pero en la actualidad, no hay información suficiente para llegar a una conclusión definitiva sobre la importancia y el impacto de la presencia de estas bacterias que permita la evaluación de los riesgos potenciales en los ecosistemas y la salud humana. La propagación de bacterias y sus genes de resistencia a los antibióticos en los ecosistemas naturales puede afectar la dinámica poblacional y la fisiología de las poblaciones microbianas nativas llegando a producir cambios relevantes en los organismos receptores.
Conflicto de intereses: El manuscrito fue preparado y revisado con la participación de todos los autores, quienes declaramos que no existe ningún conflicto de intereses, que ponga en riesgo la validez de los resultados presentados.
Referencias bibliográficas
Acevedo, R.; Severiche, C. (2013). Identificación de Bacterias Resistentes a Di-Bromo-Mercurio aisladas de Sedimentos en Playas de Cartagena de Indias, Caribe Colombiano. Rev. Avan. Inv. Ing. 10 (2): 73-79. [ Links ]
Acuña, A.; Torres, C.; Pucci, G.; Pucci, O. (2011). Evaluación del tiempo de vida de bacterias potencialmente patógenas en sedimentos marinos. Rev.Soc. Ven. Microbiol. 31:124-129. [ Links ]
Aguilera, S.; Aguilar, M.E.; Chávez, M.P.; López J.E.; Pedraza, M.; Campos, J.; Cervantes, C. (2004). Essential residues in the chromate transporter Chra of Pseudomonas aeruginosa. Fems Microb. Let. 232: 107-112. [ Links ]
Ajit, S.; Meyer, M.; Boxall,A. (2006). A global perspective on the use, sales, exposure pathways, occurrence, fate and effects of veterinary antibiotics (VAs) in the environment. Chemosphere. 65: 725-759. [ Links ]
Akiyama, T.; Savin, M. (2010). Populations of antibioticresistant coliform bacteria change rapidly in a wastewater effluent dominated stream. Sci.Tot. Environ. 408: 6192-6201. [ Links ]
Andersson, D.; Hughes, D. (2012). Evolution of antibiotic resistance at non-lethal drug concentrations. Drug. Resist. Upd. 15: 162- 172. [ Links ]
Argemi, F.; Cianni, N.; Porta, A. (2005). Disrupción endócrina: perspectivas ambientales y salud pública. Act. Bioq. Clin. Lat. 39: 291-300. [ Links ]
Ayokunle, C.; Ahmad, A. (2013). Speciation and antimicrobial resistance of Enterococci isolated from recreational beaches in Malaysia. Env. Mon. Ass. 185: 1583-1599. [ Links ]
Baquero, F.; Martinez, J.; Canto, R. (2008). Antibiotics and antibiotic resistance in water environments. Curr. Opi. Biotech. 19: 260-265. [ Links ]
Bécares; E.; Villacorta, J.; Hijosa, M.; Cardona, R. (2011). Bacterias resistentes a antibióticos en el medio ambiente acuático. Seguir. Med.Amb. 124: 25-40. [ Links ]
Brown, K.; Kulis, J.; Thomson, B.; Chapman, T.; Mawhinney, D. (2006). Occurrence of antibiotics in hospital, residential, and dairy effluent, municipal wastewater, and the Rio Grande in New México. Sci. Tot. Environ. 366: 772-783. [ Links ]
Cabello,F.(2006).Heavy use of prophylactic antibiotics in aquaculture: a growing problem for human and animal health and for the environment. Environ.Microb. 7: 1137-1144. [ Links ]
Castañeda, Y.; López, P.; Figueroa, R.; Fuentes, J. (2009). Susceptibilidad a antibióticos de bacterias indicadoras de contaminación fecal aisladas de aguas y sedimentos marinos de playas de la Isla de Margarita,Venezuela. Saber. 21: 12-19. [ Links ]
Chang, H.; Ran, T.; Rui, Z., Jian Z.; You, L. (2012). Class 1 and 2 integrons, sul resistance genes and antibiotic resistance in Escherichia coli isolated from Dongjiang River, South China. Env. Pollut. 169: 42-49. [ Links ]
De la Torre, A.; Iglesias, I.; Carballo, M.; Ramírez, P.; Muñoz, M. (2012). An approach for mapping the vulnerability of European Union soils to antibiotic contamination. Sci. Tot. Env. 414: 672-679. [ Links ]
Durso, L.; Gregory, P.; Harhay, G.; Bono J.; Smith, T. (2011). Virulence-associated and antibiotic resistance genes of microbial populations in cattle feces analyzed using a metagenomic approach. J. Microb. Meth. 84: 278-282. [ Links ]
Farkas, A.; Butiuc, A.; Ciatarâş, D.; Neamţu, C.; Crăciunaş, C.; Drăgan M. (2013). Microbiological contamination and resistance genes in biofilms occurring during the drinking water treatment process. Sci. Tot. Env. 443: 932-938. [ Links ]
Fenton, K.; Weston, A.; Caminada, D. (2006). Ecotoxicology of human pharmaceuticals. Aquat.Toxic. 76: 122-159. [ Links ]
Graham, D.; Olivares, S.; Knapp, C.; Lima L.; Werner, D.; Bowen, E. (2011). Antibiotic Resistance Gene Abundances Associated With Waste Discharges To The Almendares River Near Havana, Cuba. Environ. Sci. Technol. 45: 418-424. [ Links ]
Harada, K., Okada, E.; Shimizu, T.; Kataoka, Y.; Sawada, T.; Takahashi, T. (2012). Antimicrobial resistance, virulence profiles, and phylogenetic groups of fecal Escherichia coli isolates: A comparative analysis between dogs and their owners in Japan. Comp. Imm. Microb. Infec. Dis. 35: 139- 144. [ Links ]
Hiroshi, Y.; Nakamura, Y.; Moriguchi, S.; Sekizawa, J. (2009). Persistence and partitioning of eight selected pharmaceuticals in the aquatic environment: Laboratory photolysis, biodegradation, and sorption experiments. Wat. Res. 43: 351-362. [ Links ]
Lannacone, J.; Alvariño, L. (2009). Evaluación del riesgo acuático de siete productos farmacéuticos sobre daphnia magna. Ecol.Aplic. 8: 71-80. [ Links ]
Igbinosa, I.; Okoh. A. (2012). Antibiotic Susceptibility Profile of Aeromonas Species Isolated from Wastewater Treatment Plant. Sci. World J. 6: 34-43. [ Links ]
Jiang, J.; Lee, C.; Fang, M. (2014). Emerging organic contaminants in coastal waters: Anthropogenic impact, environmental release and ecological risk. Mar. Pol. Bul. 23: 78-82. [ Links ]
Jiang, W.; Wang, X.; Ying, F.; Hu, G.; Wang, X.; Li, D.; Yu, H.; Han, X. (2011). In vitro assessment of reproductive toxicity on rats induced by organic contaminants of source water. Ecotox. Env. Saf. 74: 1756-1764. [ Links ]
Kümmerer, K. (2009). Antibiotics in the aquatic environment - A review - Part I, Chemosphere. 75: 417-434. [ Links ]
Levin, E.; Bonilla, N.; Meschke, S.; Roberts, M. (2011). Survival of environmental and clinical strains of methicillin-resistant Staphylococcus aureus [MRSA] in marine and fresh waters. Water. Res. 45: 5681-5686. [ Links ]
Li, D.; Qi, R.; Yang, M.; Zhang, Y.; Yu T. (2011). Bacterial community characteristics under long-term antibiotic selection pressures. Water. Res. 45: 29-39. [ Links ]
López, M.; Ferrer, A. (2005). Interés de la analítica toxicológica y no toxicológica en la atención del intoxicado. Rev.Toxic. 22:71-72. [ Links ]
Martínez, A.; Cruz, M.; Veranes, O.; Carballo, M. (2010). Antibiotic and metals resistance in bacteria isolates from Almendares River. Rev. Cenic. 41: 1-10. [ Links ]
Martinez, J. (2009). Environmental pollution by antibiotics and by antibiotic resistance determinants. Env. Pol. 157: 2893-2902. [ Links ]
Moore, J.; Juñuri, R.; Peter, J.; Cherie, M.; Colin, G.; Anne, L.; Paul, R. (2010). Antibiotic resistant bacteria be an indicator of ecological change? Aquat. Ecol. 44: 349-358. [ Links ]
Morris, D.; Galvin, S.; Boyle, F.; Hickey, P.; Mulligan, M.; Cormican, M. (2012). Enterococcus faecium of the van A Genotype in Rural Drinking Water, Effluent, and the Aqueous Environment. Env. Microb. 78: 596-611. [ Links ]
Mostafa, E.; Seif, M.; Ashour, D. (2011). Microbiological Environmental Monitoring in Pharmaceutical Facility. Egypt. Acad. J. Biolog. Sci. 3: 63- 74. [ Links ]
Moura, A.; Pereira, C.; Henriques, I.; Correia, A. (2012). Novel gene cassettes and integrons in antibiotic-resistant bacteria isolated from urban wastewaters. Res. Microb. 163: 92-100. [ Links ]
Muñoz, D.; Castañeda, Y.; Graü, C. (2012). Prevalence and Susceptibility to Antibiotics of Motile Strains of Aeromonas Isolated from the Mangrove Oyster (Crassostrea rhizophorae). Rev. Cient. FCV-LUZ. 22: 565 - 573. [ Links ]
Muñoz, J.; Lang, K.; Lapara, T.; Gonzalez, G.; Singe, R. (2007). Evaluating the Effects of Chlortetracycline on the Proliferation of Antibiotic-Resistant Bacteria in a Simulated River Water Ecosystem. App. Env. Microb. 73: 5421-5425. [ Links ]
Ozaktas, T.; Taskin, B.; Gozen, A. (2012). High level multiple antibiotic resistances among fish surface associated bacterial populations in non-aquaculture freshwater environment. Water Res. 46, 6382-6390. [ Links ]
Parisien, A.; Alain, B.; Zhang, J. (2008). Novel Altermativas to antibioticts: bacteriphages, bacterial cell wall hydrolases and antimicrobial peptides. J.Appl. Microbiol. 104: 1-13. [ Links ]
Peng, S.; Jia, S.; Zhang, X.; Zhang, T.; Cheng, S.; Li, A. (2013). Metagenomic insights into chlorination effects on microbial antibiotic resistance in drinking water. Water. Res. 47: 111-120. [ Links ]
Pollard, D.; Woodley, J. (2007). Biocatalysis for pharmaceutical intermediates: the future is now. Trends. Biotechnol. 25: 66-73. [ Links ]
Riszzo, L.; Manaia, C.; Fatta, D. (2013). Urban wastewater treatment plants as hotsports for antibiotic resistant bacteria and genes spread into environment: A review. Sci. Tot. Env. 447: 345-360. [ Links ]
Román, A.; Alfaro, J. (2005). Nuevos disruptores endocrinos: su importancia en la población pediátrica. Latreia. 18: 446-45. [ Links ]
Rudolph, A.; Medina, P.; Ahumada, R.; Novoa, V. (2011). Calidad ecotoxicológica de los sedimentos en fiordos del sur de Chile. Rev. Bio. Mar. Ocea. 46, 79-84. [ Links ]
Santos, L.; Araújo, A.; Fachini A.; Pena, A.; Delerue, C.; Montenegro, M. (2010). Ecotoxicological aspects related to the presence of pharmaceuticals in the aquatic environment. J. Hazard. Mat. 175: 45-95. [ Links ]
Segura, P.; Macleod, S.; Lemoine, P.; Sauvé, S.; Gagnon, C. (2011). Quantification of carbamazepine and atrazine and screening of suspect organic contaminants in surface and drinking waters. Chemosphere. 84: 1085-1094. [ Links ]
Storteboom, H.; Davi, S.; Crimi, B.; Pruden, A. (2010). Identification of Antibiotic-Resistance-Gene Molecular Signatures Suitable as Tracers of Pristine River, Urban, and Agricultural Sources. Environ. Sci. Technol. 44: 1947-1953. [ Links ]
Torreblanca, A.; López, J. (2005). Proteómica: conceptos, desarrollo actual y aplicación en monitorización ambiental. Rev. Toxic. 22: 72-73. [ Links ]
Vaneechoutte, M.; Kampfer, P.; Baere, T.; Falsen, E.; Verschraegen, G. (2004). Wautersia gen. nov., a novel genus accommodating the phylogenetic lineage including Ralstonia eutropha and related species, and proposal of Ralsonia [Pseudomonas] syzygii comb. nov. Int. J. Syst. Evol. Microb. 54: 317-327. [ Links ]
Vargas, E.; Ruiz, L. (2007). Química Verde en el siglo XXI: Química Verde, Una Química Limpia. Rev. Cub. Quím. 14: 29-32. [ Links ]
Venegas, M.; Correa, N.; Jiménez, F. (2009). Resistencia a antibióticos de bacterias aisladas de biopelículas en una planta de alimentos. Rev. MVZ. 14: 1677-1683. [ Links ]
Volles, D.; Branan,T. (2008). Antibiotics in the intensive care unit: focus on agents for resistant pathogens. Emerg. Med. Clin. 26: 813-834. [ Links ]
Wellington, E.; Boxall, B.; Cross, P.; Williams, P. (2013). The role of the natural environment in the emergence of antibiotic resistance in Gramnegative bacteria. T.Lan.Infec.Dis.13:155 - 165. [ Links ]
Yangali, V.; Maeng, S.; Fujioka, T.; Kennedy, M.; Amy, G. (2010). Proposing nanofiltration as acceptable barrier for organic contaminants in water reuse. J. Mem. Sci. 362: 334-345. [ Links ]
Zheng, S.; Qiu, X.; Chen, B.; Yu, X. (2011). Freestone Antibiotics pollution in Jiulong River estuary: Source, distribution and bacterial resistance. Chemosphere. 84:1677-1685. [ Links ]
Zhenhua, Y.; Yang, X.; Lu, G.; Liu, J.; Xie, Z.; Wu, D. (2014). Potential environmental implications of emerging organic contaminants in Taihu Lake, China: Comparison of two ecotoxicological assessment approaches. Sci. Tot. Env. 470: 171-179. [ Links ]