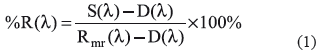Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Revista Ingeniería Biomédica
versão impressa ISSN 1909-9762
Rev. ing. biomed. vol.4 no.8 Medellín jul./dez. 2010
MÉTODOS DE CLASIFICACIÓN PARA IDENTIFICAR LESIONES EN PIEL A PARTIR DE ESPECTROS DE REFLEXIÓN DIFUSA
CLASSIFICATION METHODS TO IDENTIFY LESIONS IN SKIN STARTING FROM SPECTRA OF DIFFUSE REFLECTANCE
Eber Enrique Orozco Guillén1, Guillermo Iruretagoyena Garcia2, Sergio Vazquez y Montiel3, José Alberto Delgado-Atencio3, Jorge Castro Ramos3, Francisco Gutierrez Delgado4
1 Facultad Experimental de Ciencia y Tecnología, Departamento de Física, Universidad de Carabobo, Valencia, Venezuela. eeorozco@uc.edu.ve.
2 Facultad Experimental de Ciencia y Tecnología, Departamento de Computación, Universidad de Carabobo, Valencia, Venezuela.
3 Instituto Nacional de Astrofísica Óptica y Electrónica, Puebla, México.
4 Centro de Estudios y Prevención del Cáncer, Oaxaca.
Recibido 3 de Octubre de 2010. Aceptado 16 de Diciembre de 2010
RESUMEN
Con el objetivo de discriminar lesiones benignas y malignas en la piel humana a partir de espectros de reflexión difusa, se han analizado diferentes algoritmos de clasificación usando el software de aprendizaje automático y reconocimiento de patrones WEKA. Además, dada la alta dimensionalidad de la señal espectral, fue empleada una técnica selección de atributos para determinar las variables que aporten la mayor cantidad de información. Se probó la clasificación de la señal usando los algoritmos de máquinas de vectores de soporte, redes neuronales y bosques aleatorios, el desempeño fue evaluado usando el promedio de la k-fold cross-validation tomando en cuenta los porcentajes de instancias clasificadas correctamente, el índice kappa, el área bajo la curva ROC, la sensibilidad, y la especifidad. Finalmente se demuestra que el algoritmo de redes neuronales con los parámetros momentum y learning rate en 0,6 y 0,3 respectivamente, es el que mejor se adapta al problema de reconocimiento de patrones ya que clasifica correctamente al 89,89% de los casos.
PALABRAS CLAVE: Cáncer, Espectroscopia de reflexión difusa, Óptica de tejidos, Reconocimiento de patrones.
ABSTRACT
In order to differentiate between benign and malignant lesions in the human skin using diffuse reflection spectra, different classification algorithms were tested using the WEKA data mining software. In addition, due to the high dimensionality of the spectral signal, an attribute selection technique was applied to determine the variables that contribute with more information. The spectral signal classification was tested using support vector machines, neural networks and random forests, their performance was measured using the k-fold cross-validation percentages of the Kappa statistic, area under the ROC curve, specificity and sensitivity. Finally it is shown that the one layer neural network with 6 neurons and the parameters momentum and learning rate in 0.6 and 0.3 respectively, is best suited to the problem of pattern recognition, achieving correctly classify 89.89% of the cases.
KEY WORDS: Cancer, Diffuse reflectance spectroscopy, Tissue optics, Pattern recognition.
I. INTRODUCCIÓN
El diagnóstico de lesiones pigmentadas en piel se hace generalmente mediante la apreciación visual empleando el método ABCD [1,2] (asimetría, borde, color, dimensión), principalmente del color y el borde. La biopsia aún se mantiene como la regla de oro para obtener un diagnóstico fiable, sin embargo estas son costosas, invasivas y los resultados no se obtienen inmediatamente. Además los especialistas fallan en realizar diagnósticos rápidos al no poder identificar lesiones pequeñas como malignas si no hasta poder estudiar su evolución [3], lo que trae consigo retrasos en la intervención. La detección temprana de ciertos tipos de lesiones en la piel humana como los melanomas es importante, dado que el éxito en la intervención de este tipo de lesiones disminuye en etapas avanzadas, produciendo el mayor porcentaje de muertes relacionadas con el cáncer de piel. Por esta razón en los últimos años se han desarrollado investigaciones empleando técnicas ópticas no invasivas que permiten diagnosticar lesiones y extraer parámetros ópticos en tejidos biológicos [4-6].
La espectroscopia de reflexión difusa es una técnica óptica no invasiva que ha sido ampliamente usada para la caracterización de tejidos biológicos [7-10]. Esta técnica ha sido aplicada al estudio de lesiones de piel, ya que permite obtener información suficiente para la clasificación de una lesión como maligna o benigna. Sin embargo una vez obtenida la señal espectral, no existe un criterio único para la categorización de los espectros, por ello para llevar a cabo la tarea de clasificación es necesaria la utilización de técnicas de reconocimiento de patrones que aprendan las posibles diferencias entre patrones de tejido sano y lesionado, y logren con un alto porcentaje de éxito, diagnosticar a cual grupo pertenece un espectro. Para llevar a cabo tareas de reconocimiento de patrones es necesario contar con un conjunto de datos certificados, en nuestro caso los datos han sido valorados por médicos especialistas en oncología y dermatología.
El principio fundamental para diagnosticar enfermedades con técnicas espectroscópicas consiste en construir algoritmos robustos que permitan extraer las características más importantes de la señal espectral y correlacionarlas con su respectiva patología [11]. Los algoritmos de clasificación se han aplicado para la solución de problemas de diversa índole, una de las áreas más importantes es la solución de problemas de naturaleza médica. Para detectar lesiones cancerígenas en el cuello uterino, se han probado usando WEKA, algoritmos como el ensemble AdaBoostM1-Reptree, para diferenciar entre imágenes (obtenidas mediante un colposcopio) de cuello uterino sano y cuello uterino con lesiones precancerosas, logrando un porcentaje de acierto del 89,4737% [12]. La detección de lesiones en la piel mediante espectrometría de reflexión difusa y algoritmos de clasificación fue probada por Wallace et al. [7], usando una red neuronal formada por siete neuronas; en la capa oculta fue posible obtener un 83,3% de sensibilidad y 88,9% de especificidad en la clasificación de la señal espectral. Alternativamente para detectar lesiones en la piel, también se ha empleado el análisis de imágenes obtenidas mediante epiluminescencia [13], en conjunto con algoritmos como el clasificador k-nearest neighbors (KNN) se evaluaron atributos en la imagen como: tamaño de la lesión, media del borde de la lesión. Otra aplicación médica a los algoritmos de reconocimiento de patrones, es la detección de diferentes tipos de lesiones en el tejido oral mediante el uso de redes neuronales, con un 93% de exactitud fueron capaces de diferenciar entre tejido sano y tumores cancerosos, pero no pudieron lograr discernir con entre tejido sano y tejido pre-canceroso [14].
En este trabajo se ha evaluado el desempeño de diferentes técnicas computacionales para clasificar espectros de reflexión difusa entre muestras de tejido sano y maligno, usando el software WEKA de investigación en aprendizaje automático y minería de datos, las técnicas implementadas son máquinas de vectores de soporte, redes neuronales y bosques aleatorios.
Después de aplicar estas técnicas de clasificación, se ha demostrado que el algoritmo correspondiente a las redes neuronales es el que tiene mejor desempeño, alcanzando 89,89% de aciertos en la clasificación de los casos estudiados. Así, la espectroscopia de reflexión difusa, con el soporte de las técnicas de reconocimiento de patrones y la respectiva correlación con la regla de oro (la biopsia) en el diagnóstico de enfermedades de la piel, es una técnica prometedora y no invasiva, de respuesta rápida y que puede ser empleada en jornadas masivas de detección de cáncer de piel por médicos no expertos en el área dermatológica.
II. MATERIALES Y MÉTODOS
La colección de los espectros de reflexión difusa en piel humana fue obtenida de pacientes voluntarios que asistieron a una jornada de prevención de cáncer de piel promovida por el Centro de Prevención de Cáncer (CEPREC) en la ciudad de Juchitán de Zaragoza en el estado de Oaxaca (México), en total fueron empleados en este trabajo 2000 espectros, correspondientes a 285 lesiones de 91 pacientes, diagnosticadas por un médico especialista.
2.1 Sistema experimental
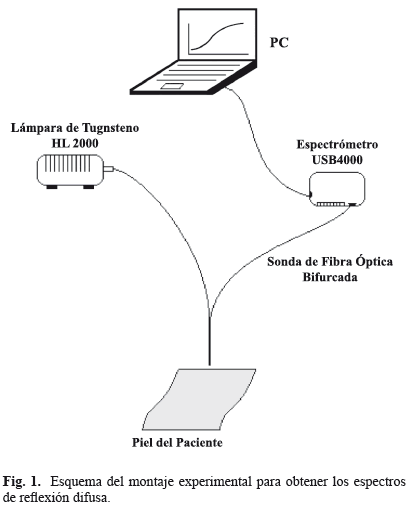
En la Fig. 1 se presenta un esquema del arreglo experimental empleado para capturar los espectros, el cual consiste de un espectrómetro USB4000 fabricado por la empresa Ocean Optics que está equipado con un detector CCD Toshiba de 3648 elementos, una fuente de luz HL2000 optimizada para el VIS-NIR (360 nm-2000 nm), una sonda de fibra óptica bifurcada (R600\7\VIS\125F) de la misma firma comercial, un patrón de reflexión (Teflón) y un computador con el software SpectraSuite (Ocean Optics) que calcula automáticamente el porcentaje de luz reflejada mediante la siguiente expresión matemática [15].
Donde S(λ) es la señal del medio analizado (Piel) para cada longitud de onda (λ), D(λ) es la señal de oscuridad y Rmr(λ) es la muestra de referencia.
En la Fig. 2 es mostrada una fotografía con una lesión de tipo carcinoma Basocelular y dos curvas espectrales una correspondiente a la lesión y otra a un tejido sano o piel normal.
2.2 Algoritmos computacionales
Una vez obtenidos los espectros mediante el sistema experimental (Fig.1), es necesario acondicionarlos para las siguientes etapas de la clasificación. Este proceso consiste en normalizar los datos para evitar que la función de decisión sea influenciada por variables de magnitudes considerablemente mayores que otras; y posteriormente aplicar una técnica de selección de atributos para analizar la influencia de los atributos de los patrones de entrenamiento en el proceso de su propia categorización y así poder determinar un subconjunto óptimo de atributos para evitar redundancias de información. La clasificación de los espectros fue realizada mediante las máquinas de vectores de soporte, árboles aleatorios y redes neuronales artificiales.
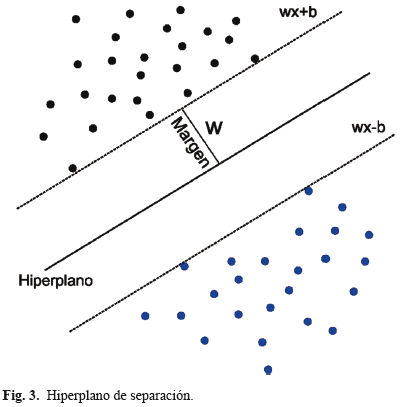
Las máquinas de soporte vectorial (MSV) han sido desarrollados por Vapnik [16] y han ganado mucha popularidad debido a sus características, rendimiento, a que permiten enfrentar problemas de clasificación en dominios complejos y pueden ser usadas para extraer información relevante a partir de conjuntos de datos y construir algoritmos de clasificación eficientes y rápidos para datos masivos. Son un modelo de clasificación cuyo funcionamiento se basa en la búsqueda de un margen de separación máximo entre un hiperplano y los patrones de las diferentes clases que comprenden el conjunto de entrenamiento, el modelo de optimización busca establecer la frontera de decisión mediante los patrones que más resaltan las distribución de clases, estos son los llamados vectores de soporte. En la Fig. 3 es mostrado el hiperplano de separación que es calculado maximizando la distancia de los patrones más cercanos.
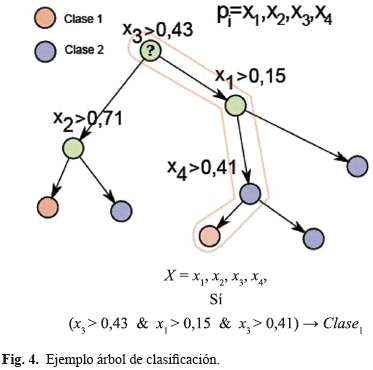
Los árboles de decisión (AD), son árboles dirigidos en los que en cada nodo se realiza una consulta a una de las características de un patrón con el objetivo de asignarle una categoría. Partiendo desde el nodo raíz hasta algún nodo hoja se van considerando las posibilidades de que el patrón pertenezca a una u otra clase, la decisión final depende del nodo hoja en el que se termine, ya que cada uno de estos nodos tiene asociada una categoría (Fig. 4). El uso de un árbol de clasificación como discriminador se puede interpretar mediante la conjunción lógica de las decisiones tomadas en cada nodo. Entre los métodos empleados con esta técnica se tiene el Random Forest (bosques aleatorios) que es un algoritmo compuesto por numerosos árboles de clasificación, en el se definen una cantidad de árboles a desarrollar y una cantidad de atributos m tal que sea menor a la cantidad total de atributos. Entre los árboles se reparten k patrones con reemplazo y se desarrollan los árboles, el resto de los patrones son usados para la prueba. Al desarrollar cada nodo se eligen m atributos y se determina el mejor atributo para desarrollar el nodo. Para el entrenamiento los patrones son repartidos aleatoriamente con repetición entre cada árbol.
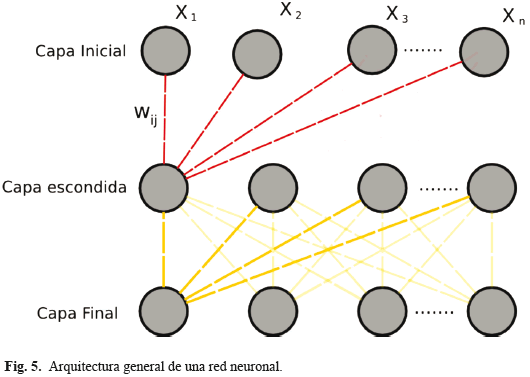
Las redes neuronales (RN) consisten en un sistema de procesamiento de información planteado inicialmente por inspiración biológica, está compuesta por unidades de procesamiento llamadas neuronas que están separadas en capas donde se recibe, procesa y transmite la información. Las unidades de procesamiento reciben como entrada los elementos del patrón con el que es alimentada la red neuronal y estas transmiten los elementos como señales a la siguiente capa, el enlace entre cada capa está afectado por un peso que biológicamente representa el nivel de sinapsis en la conexión. Las salidas de las neuronas de la última capa representan la respuesta de la red neuronal al estimulo inicial. En la Fig. 5 es mostrada la arquitectura de una red neuronal de flujo hacia delante.
Para realizar las tareas de clasificación con las técnica mencionadas se ha utilizado el software WEKA (Waikato Environment for Knowledge Analysis), que es una aplicación ampliamente usada para la experimentación e investigación en áreas como el reconocimiento de patrones y la minería de datos, ya que incluye una gran cantidad de algoritmos de preprocesado, clasificación y clustering. Es posible realizar experimentos de reconocimiento de patrones usando alguno de los módulos gráficos que ofrece WEKA o mediante el uso de los algoritmos de clasificación que poseen aplicaciones escritas en el lenguaje JAVA [17]. En este trabajo se probó la segunda alternativa ya que de esta manera es posible variar automáticamente los parámetros de los algoritmos empleados.
III. RESULTADOS Y DISCUSIÓN
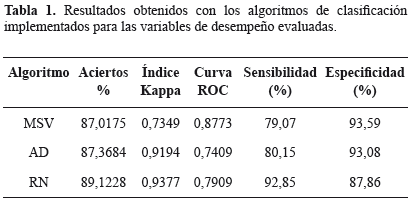
En la prueba de cada algoritmo se variaron los parámetros de los mismos en diferentes intervalos, cada prueba se realizó mediante validación cruzada con k=10 pliegues. Es decir, el conjunto de instancias disponibles se divide en diez partes iguales usando una para la validación del modelo y el resto para su entrenamiento, el resultado final de la prueba se obtiene al promediar la métricas arrojadas en cada una de las pruebas. La validez de los modelos fue evaluada con las mediciones del porcentaje de predicciones correctas, el área bajo la curva ROC, el índice Kappa y el promedio de la sensibilidad y especificidad de los resultados (Tabla 1).
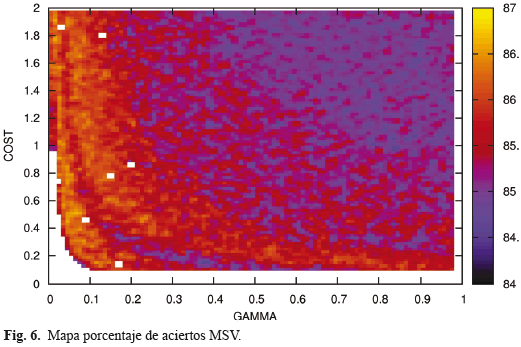
Las máquinas de soporte vectorial fueron implementadas con un núcleo RBF (Función de Base Radial) variando los parámetros γ entre 0 y 1 con pasos de 0,1 y C entre 0,1 y 2 con pasos de 0,2; en total fueron realizadas 100 pruebas. Al realizar los experimentos se evidencia la relación entre los parámetros γ y C y los resultados. En la Fig. 6 se puede apreciar que la relación entre estos parámetros no es lineal, pues se forma una curva en la que los valores más altos para las variables de medición del desempeño se dan por la combinación de valores altos de γ y bajos de C y viceversa.
El estudio con los árboles de decisión fue llevado a cabo mediante el algoritmo Random Forest, variando los parámetros NumTrees (número máximo de árboles) entre 1 y 110 y NumFeatures (número de características) entre 1 y 11. En total se realizaron 1210 pruebas. Este algoritmo logra clasificar correctamente al 88,071% de los espectros sí se le permite desarrollar 44 árboles y evaluar cuatro atributos en cada uno.
En la Fig. 7 no se observa a simple vista una relación entre la variación de los parámetros y las variables de medición, aunque la documentación del Random Forest señala que este es considerablemente sensible a la variación del número de características.
La red neuronal implementada en WEKA corresponde a la clase MultilayerPerceptron, que es un perceptron multicapa entrenado usando backpropagation. Se utilizó una capa oculta y se varió la cantidad de neuronas entre 3 y 12 y en cada caso, los parámetros momentum y learnig-rate fueron variados en el intervalo 0-1. Aunque no se percibe que el sistema sea sensible a la variación de los parámetros, al validar el modelo con las medidas de desempeño, se ha obtenido un índice kappa que indica que existe un alto acuerdo entre las predicciones y las clases originales. En cuanto a la sensibilidad y especificidad, los parámetros obtenidos indican que la red neuronal tiene una alta capacidad de detectar correctamente a pacientes sanos y enfermos. La Fig. 8 corresponde a los mapas del porcentaje de aciertos, variando el número de neuronas.
IV. CONCLUSIÓN
La técnica de espectroscopia de reflexión difusa en conjunto con un sistema de reconocimiento de patrones, entrenado con una amplia base de datos certificada por la regla de oro en el diagnóstico de lesiones en piel como es la biopsia, y el criterio expertos en oncología y dermatología permitiría implementar un sistema automático para llevar a cabo jornadas masivas de diagnóstico de cáncer de piel a muy bajo costo, ya que personal médico no especialista con entrenamiento en el manejo del equipo y la adquisición de datos podrían llevar a cabo esta actividad.
En cuanto a los resultados obtenidos, una vez evaluados cada uno de los modelos de clasificación elegidos, se concluye que una red neuronal de una capa oculta de seis neuronas con los parámetros momentum y learnig-rate en 0,6 y 0,3 respectivamente, poseen la mayor capacidad de distinguir entre patrones correspondientes a lesiones sanas y malignas. Es decir que existe una alta probabilidad que el modelo clasifique como positivas las instancias positivas; además el alto promedio de sensibilidad y especificidad indican que el modelo tiene un excelente desempeño para diferenciar entre lesiones y tejido sano. De forma general todos los algoritmos implementados tienen un buen desempeño. Es importante resaltar la necesidad de extender la experimentación usando un mayor número de espectros de entrenamiento que estén etiquetados como muestras de tejido sano o maligno mediante el diagnóstico realizado con biopsia por parte de un patólogo, y además el probar nuevas técnicas de clasificación con el objetivo de obtener mayores porcentajes de acierto.
REFERENCIAS
[1]. Wolf K., Johnson R.A. Color atlas and synopsis of clinical dermatology. McGraw Hill, Sección 9, 178-191, 2009. [ Links ]
[2]. Friedman R.J., Rigel D.S., Kopf A.W. Early detection of malignant melanoma: the role of physician examination and selfexamination of the skin. American Cancer Journal for Clinicians, 35, 130-151, 1985. [ Links ]
[3]. Abbasi N.R. Early diagnosis of cutaneous melanoma: revisiting the ABCD criteria. Journal of the American Medical Association, 292, 22, 2771-2776, 2004. [ Links ]
[4]. Mirabal Y.N., Chang S.K., Atkinson E.N., Malpica A., Follen M., Richards-Kortum R.R. Reflectance spectroscopy for in vivo detection of cervical precancer, Journal of Biomedical Optics., 7, 587-594, 2002. [ Links ]
[5]. Koenig F., Larne R., Enquist H., McGovern F.J., Schomacker K.T., Kollias N. Deutsch T.F. Spectroscopic measurement of diffuse reflectance for enhanced detection of bladder carcinoma. Urology 51, 342-345, 1998. [ Links ]
[6]. Skala M.C., Palmer G.M., Vrotsos K.M., Gendron-Fitzpatrick A., Ramanujam N. Comparison of a physical model and principal component analysis for the diagnosis of epithelial neoplasias in vivo using diffuse reflectance spectroscopy. Optics. Express 15, 7863-7875, 2007. [ Links ]
[7]. Wallace P., Crawford D.C., Mortimer P.S., Ott R. J., Bamber J.C. Spectrophotometric assessment of pigmented skin lesions: methods and feature selection for evaluation of diagnostic performance. Physics in Medicine and Biology. 45, 735-751, 2000. [ Links ]
[8]. Scarisbrick J. J., Pickard C.D.O., Lee A.C., Brigs G.M., Kristie J., Bown S.G., Novelli M., Keshtgar M.R.S., Bigio I.J., Yu R. Elastic scattering spectroscopy in the diagnosis of pigmented lesions: comparison with clinical and histopathological diagnosis. Proceedings of SPIE, 5141, 147-156, 2003. [ Links ]
[9]. Bigio J., Mourant J.R. Ultraviolet and visible spectroscopies for tissue diagnostics: fluorescence spectroscopy and elasticscattering spectroscopy. Physics in Medicine and Biology. 42, 803-813, 1997. [ Links ]
[10]. Muller M.G., Valdez T.A., Georgakoudi I., Backman V., Fuentes C., Kabani S., Laver N., Wang Z., Boone C.W., Dasari R., Shapsay S.M., Feld M.S. Spectroscopic detection and evaluation of morphologic and biochemical changes in early human oral carcinoma. Cancer, 97, 1681-1692, 2003. [ Links ]
[11]. Lin W., Yuan X., Yuen P., Wei W., Sham J., Shi P. Classification of in vivo autofluorescence spectra using support vector machines. Journal of Biomedical. Optics, 9, 180-186, 2004. [ Links ]
[12]. Palma. Sistema de apoyo al diagnóstico médico en detección de lesiones precancerosas cérvico uterinas, Master's thesis, Universidad de Carabobo, 2010. [ Links ]
[13]. Burroni M. Melanoma computer-aided diagnosis: reliability and feasibility study. Clinical Cancer Research, 10, 1881-1886, 2004. [ Links ]
[14]. De Veld D.C.G., Skurichina M., Witjes M.J.H., Duin R.P.W., Sterenborg H.J.C., Roodenburg J. L.N. Clinical study for classification of benign, dysplastic, and malignant oral lesions using autouorescence spectroscopy, Journal of Biomedical Optics, 9, 940-950, 2004. [ Links ]
[15]. Oceans Optics. SpectraSuite Installation and Operation Manual, Document Number 000-20000-300-02-0607. Appendix A, 125-128, 2006. [ Links ]
[16]. Vapnik. The nature of statistical learning theory. Springer-Verlag, Chapter 5, 138-141, 2000. [ Links ]
[17]. Ian H., Eibe F., Len T., Holmes M.H., Cunningham S.J. Weka: Practical machine learning tools and techniques with java implementations. Department of Computer Science. University of Waikato. New Zealand. Consultado el 10 Mayo de 2010 en: http://www.cs.waikato.ac.nz/~ml/publications/1999/99IHW-EFLT-MH-GH-SJC-Tools-Java.pdf. [ Links ]