Serviços Personalizados
Journal
Artigo
Indicadores
-
 Citado por SciELO
Citado por SciELO -
 Acessos
Acessos
Links relacionados
-
 Citado por Google
Citado por Google -
 Similares em
SciELO
Similares em
SciELO -
 Similares em Google
Similares em Google
Compartilhar
Revista Ingeniería Biomédica
versão impressa ISSN 1909-9762
Rev. ing. biomed. vol.8 no.15 Medellín jan./jun. 2014
CLASIFICACIÓN DE LATIDOS DE CONTRACCIÓN VENTRICULAR PREMATURA BASADOS EN MÉTODOS DE APRENDIZAJE NO SUPERVISADO
CLASSIFICATION OF PREMATURE VENTRICULAR CONTRACTION BEATS BASED ON UNSUPERVISED LEARNING METHODS
CLASSIFICAÇÃO DE PREMATUROS BATIDAS CONTRAÇÃO VENTRICULAR BASEADO SUPERVISIONADO MÉTODOS DE APRENDIZAGEM
C.A. Rodriguez1,2, J.H. Gallego1, I.D. Mora1, A. Orozco-Duque3, J. Bustamante3
1 Semillero de Investigación en Automática y Diseño.
2 Dirección para correspondencia: carlos.rodriguez91815@gmail.com.
3 Centro de Bioingeniería Universidad Pontificia Bolivariana, Medellín, Colombia.
Recibido 4 de septiembre de 2013. Aprobado 4 de junio de 2014.
RESUMEN
Las enfermedades cardiovasculares son la principal causa de mortalidad en el mundo, por lo que el desarrollo de algoritmos que detecten arritmias cardíacas en tiempo real se ha convertido en un campo de investigación importante. El desarrollo de estos algoritmos ha conllevado a la mejora de dispositivos cardiacos portátiles. Este artículo presenta el desempeño de dos algoritmos basados en aprendizaje de máquina no supervisado para la detección de latidos de contracción ventricular prematura en la señal ECG. Los latidos se extraen de las bases de datos del MIT-BIH, los cuales fueron pre-procesados y segmentados por el grupo de investigación de Dinámica Cardiovascular de la UPB. La Transformada Wavelet Discreta, el Análisis de Componentes Principales y un método híbrido propuesto son implementados para la extracción de características y reducción de dimensiones, a partir de los cuales se generan 8 espacios de características para la evaluación de los algoritmos. Kmeans y Mapas auto-organizados son desarrollados y comparados en términos de precisión y costo computacional. Se logró una especificidad del 96.22 % y una sensibilidad del 95.04 % con un tiempo de ejecución de 79.41µs por latido. Los resultados permiten concluir que estos métodos pueden implementarse en aplicaciones de detección de arritmias en tiempo real debido a su bajo costo computacional.
PALABRAS CLAVES: Arritmia; Latido ventricular prematuro; Transformada Wavelet Discreta; Análisis de Componentes Principales; Kmeans; SOM.
ABSTRACT
Cardiovascular diseases are the principal cause of mortality in the world, so that the development of algorithms that detect cardiac arrhythmias in real time has become an important field of research. The development of these algorithms has led to the improvement of wearable cardiac devices. This paper presents the performance of two algorithms based in unsupervised learning methods for the detection of Premature Ventricular Contraction in the ECG signal. The beats are extracted from MIT-BIH databases, which were preprocessed and segmented by the UPB’s Dynamic Cardiovascular research group. The Discrete Wavelet Transform (DWT), Principal Component Analysis (PCA) and a proposed hybrid method are implemented for the feature extraction and dimension reduction, from which 8 feature spaces are generated and tested. Kmeans and Self Organizing Maps are developed and compared in terms of accuracy and computational cost. Specificity of 96.22 % and sensitivity of 95.94% with 79.41µs per beat are accomplished. The results show that these methods can be implemented in applications of real time arrhythmia detection because of their low computational cost.
KEYWORDS: Arrhythmia; Premature Ventricular Beat; Discrete Wavelet Transform; Principal Component Analysis; Kmeans; SOM.
RESUMO
A doença cardiovascular é a principal causa de morte em todo o mundo, de modo que o desenvolvimento de algoritmos para detectar arritmias cardíacas, em tempo real, tornou-se um importante campo de pesquisa. O desenvolvimento desses algoritmos tem levado a melhores dispositivos cardíacos portáteis. Este artigo apresenta o desempenho dos dois com base na aprendizagem de máquina sem supervisão para detecção de batidas de contração ventriculares prematuras nos algoritmos de sinais de ECG. As batidas são extraídos das bases de dados do MIT-BIH, que foram pré-processados e segmentado pelo grupo da UPB Cardiovasculares Dynamics pesquisa. A Transformada Wavelet Discreta, Análise de Componentes Principais e uma abordagem híbrida proposta são implementadas para extração de características e redução de dimensão, a partir do qual 8 espaços de recursos para a avaliação dos algoritmos são gerados. Kmeans e mapas de auto-organização são desenvolvidos e comparados em termos de precisão e custo computacional. A especificidade de 96,22% e uma sensibilidade de 95,04% com um tempo de execução de 79.41µs por batida foi alcançado. Os resultados mostram que estes métodos podem ser implementados em aplicações de detecção de arritmia em tempo real, devido ao seu baixo custo computacional.
PALAVRAS-CHAVE: Arritmia; Ventricular batida prematura; Transformada Wavelet Discreta; Análise de Componentes Principais; Kmeans; SOM.
I. INTRODUCCIÓN
El electrocardiograma (ECG) ha llegado a ser una herramienta clínica indispensable para el diagnóstico de enfermedades cardiovasculares. Hoy en día es registrado en diversas aplicaciones clínicas, donde los algoritmos de procesamiento de señales son esenciales para la extracción de información confiable [1]. Entre estas enfermedades cardíacas, se encuentran las arritmias, muchas de las cuales son fatales sino se tratan inmediatamente. Por esta razón, ha sido de gran importancia el desarrollo y mejoramiento de dispositivos portátiles, tales como los monitores tipo holters, los cuales pueden registrar y almacenar la actividad eléctrica del corazón [2]; los más comunes son los de 24 y 48 horas, aunque también existen holters de una semana.
Para un cardiólogo, analizar 24 horas o más de la señal eléctrica del corazón registrada sería demasiado tedioso, por lo que se han desarrollado algoritmos que agrupen los latidos con patrones similares de la señal registrada, con el fin de facilitar al especialista la lectura del registro. Los tipos de algoritmos que pueden implementarse en esta clase de dispositivos, son los no supervisados basados en clustering (agrupamiento) [3].
Por esta razón, como objetivo de este trabajo, se implementaron dos algoritmos basados en aprendizaje no supervisado: Kmeans y mapas auto-organizados (en inglés SOM, Self organized maps). Antes de implementar los algoritmos de clustering, se pre-procesa la señal, se segmentan los latidos y se realiza la extracción de características a partir de métodos como la transformada wavelet discreta (en inglés DWT, Discrete Wavelet Transform) [4] y el análisis de componentes principales (en inglés PCA, Principal Component Analysis) [5].
El algoritmo de Kmeans es uno de los métodos de clustering más conocidos. Este término fue utilizado por primera vez por MacQueen en 1967 [6], aunque Steinhaus en 1956 [7] propuso un algoritmo que realiza la división de un sólido n-dimensional en k partes con el fin de minimizar la suma de las inercias de cada clase específica. En 2007, Rodríguez, Cuesta y Castellanos en [8] desarrollan un algoritmo de agrupamiento, donde usaron la DWT de cuarto orden para la extracción de características de la señal ECG. Con estas características se agrupan los datos en 2 etapas, con el fin de reducir el costo computacional: el algoritmo Dynamic Time Warping (DTW) como preclustering, y luego un algoritmo Kmeans modificado llamado Kmedians. En 2010, Mehta, Shete, Lingayat y Chouhanc en [9] implementan el algoritmo Kmeans para la detección de complejos QRS a partir de la señal ECG, formando dos grupos: un grupo con los latidos que tienen complejos QRS y otro grupo con los latidos que no tienen.
El algoritmo SOM es un método basado en aprendizaje competitivo y ha sido ampliamente usado. En 2006, Kinoshita y colaboradores [10] desarrollaron un algoritmo para la detección de arritmias cardíacas fatales con un desfibrilador automático implantable, extrayendo las características de la señal ECG y realizando pruebas en un modelo canino. Las neuronas fueron etiquetadas con el método supervisado KNN [11], lo que conllevó a que la precisión del mismo fuera mejor que otros métodos competitivos. Entre otros, se han hecho modificaciones sobre el algoritmo SOM, como lo hicieron Wen y colaboradores en 2008 [12], quienes combinan dos métodos de redes neuronales, el SOM y el controlador de la articulación del modelo cerebelosa [13], con el fin de clasificar arritmias basándose en las características del complejo QRS.
II. MATERIALES Y MÉTODOS
2.1 Bases de datos
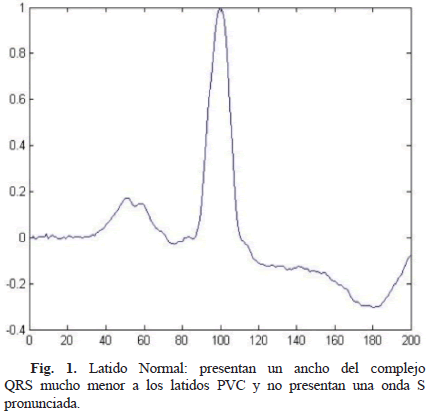
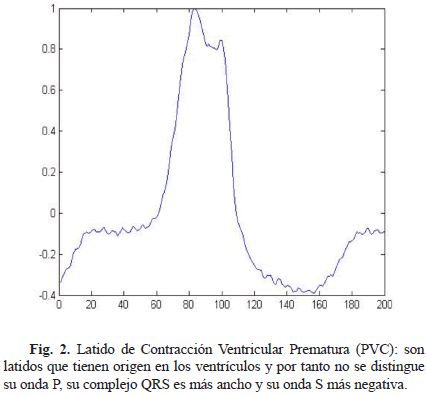
Se usaron las bases de datos del MIT-BIH Arrhythmia [14]. Solo se consideraron latidos normales y los latidos de contracción ventricular prematura (en inglés PVC, Premature Ventricular Contraction), los cuales son el principal enfoque de este trabajo. Se extrajeron en total 4234 latidos, de los cuales se tomaron 2538 latidos (60 %) para el conjunto de entrenamiento, 850 latidos para el conjunto del test (20 %) y 846 latidos para el conjunto de la validación cruzada (20 %). Cada conjunto se construye con igual número de latidos normales (0's) y PVC (1's) seleccionados aleatoriamente para evitar un desbalance en las clases. En las Fig. 1 y 2 se ilustran las morfologías de los latidos.
2.2 Transformada Wavelet Discreta
La transformada wavelet representa una señal en una secuencia de coeficientes basados en bases ortogonales (ψ) de ondas finitas (wavelet madre, trasladada y escalada en el tiempo). Se define la transformada wavelet para señales continuas como la correlación entre la señal y la función ψ escalada y trasladada en el tiempo. Para aplicaciones prácticas se definió la DWT la cual es un muestreo discreto de la transformada wavelet. El muestreo se realiza con el fin de reducir la redundancia [3].
2.3 Análisis de Componentes Principales
También conocido como un método de proyección, el objetivo es reducir un conjunto de datos n-dimensional en un espacio k-dimensional, siendo k<n, con la mínima pérdida de información y maximizando la varianza [4].
2.4 Pre-Procesamiento y segmentación
El pre-procesamiento de las señales extraídas de las bases de datos y la segmentación de los latidos fueron desarrollados por Bustamante C., Duque S., Orozco A. y Bustamante J. en [16], quienes usan la FDWT (Fast Discrete Wavelet Transform) para la reducción de ruido, remover la línea base y reducir la interferencia de línea de potencia. Se implementó la FDWT con 8 niveles de descomposición con las wavelet madre Daubechies y Symlets de diferentes órdenes, de forma que se descompone la señal en escalas de frecuencia, colocando un umbral conforme al nivel de relación señal a ruido. El umbral se aplica a los coeficientes de detalle cD1-cD8. Se elimina el ruido de la línea base removiendo los coeficientes de aproximación cA8, correspondientes a las bajas frecuencias.
Para la segmentación, se implementó un algoritmo basado en la transformada wavelet para detectar el pico R conforme a [17]; la suma de los coeficientes de detalle reconstruidos cD2, cD3, cD4 y cD5 son usados para detectar el complejo QRS. Los picos R son detectados usando un umbral adaptativo, y luego se extraen 200 puntos alrededor del pico R identificado.
2.5 Extracción de características
A partir de los latidos segmentados se crea un espacio de características con cada punto que conforma el latido, es decir, con 200 dimensiones. A este espacio se le denomina el espacio temporal, ya que representa la señal en el tiempo de cada latido.
Se realiza una descomposición wavelet de 8 niveles con las madres wavelet Daubechies y Symlets de orden 9 (Db9 y Sym9). A partir de cada descomposición, se extraen los coeficientes wavelet pertenecientes a las sub-bandas cA8, cD8, cD7...cD1 para crear dos espacios de características. Los coeficientes de detalle cD1 y cD2 se remueven debido a que corresponden a las altas frecuencias y solo representan el 2 % de la energía total. Como resultado, cada espacio generado contiene 160 dimensiones.
A partir de los coeficientes seleccionados se extraen los porcentajes de la energía contribuida por cada sub-banda y se almacenan como características para la creación de un nuevo espacio. Como resultado se genera un espacio de 7 dimensiones para cada madre wavelet.
Se aplica PCA a los coeficientes wavelet del método Sym9 (ya que obtuvo mayor desempeño que Db9 en la etapa de pre-procesamiento [16]) para crear otro espacio donde se reducen 160 dimensiones a 10, preservando así el 81,55 % de la varianza. También se crea un espacio con los componentes principales de las energías de las sub-bandas wavelet sym9.
De esta forma se tienen 7 espacios de características diferentes, con los cuales se evalúan los algoritmos, aunque se encontró la necesidad de crear un espacio de características adicional con el objetivo de mejorar el desempeño de los métodos no supervisados. En este tipo de métodos, que no tienen etiquetas, la eficiencia radica en las características del espacio de entrada. Por esta razón, se almacenaron características como el valor absoluto del área bajo la curva del complejo QRS y de la onda S, los cuales son los patrones más dominantes de la señal ECG, como se observa en las Fig. 1 y 2. También se encontró que 3 características de las componentes principales de los coeficientes wavelet sym9 ayudan a mejorar la caracterización de la señal ECG. De esta forma se crea el espacio con las 5 mejores características, al que se le denomina espacio híbrido.
2.6 Clustering
Los algoritmos Kmeans y SOM son usados en la etapa de clustering.
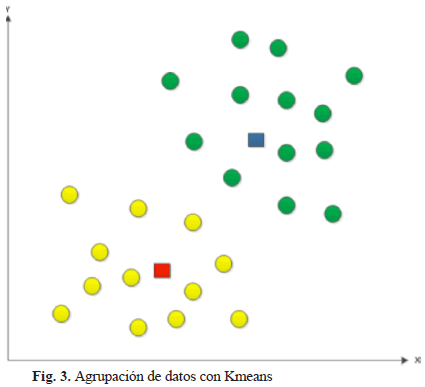
El objetivo de Kmeans es particionar un espacio de entrada m-dimensional en k grupos de tal manera que los puntos de un grupo sean similares entre sí. En este método se busca encontrar los centroides y los vectores indicadores que minimicen la función costo. Los centroides o vectores prototipo se definen como µ1, µ2 ,… µk y los vectores indicadores se definen como rij [18]. En la Fig. 3 se ilustra un ejemplo donde se agrupan los datos (círculos) alrededor del centroide más cercano (cuadrados). Para este artículo, se implementa Kmeans usando las Statistics Toolbox de MATLAB para obtener el resultado para cada latido.
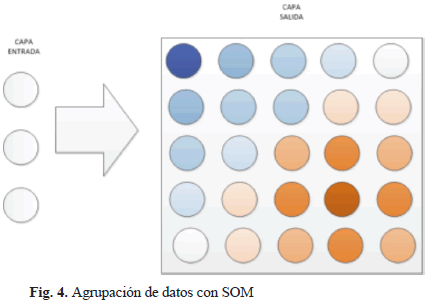
SOM es un método que distribuye los datos de un espacio de entrada en una rejilla, usualmente bidimensional, de tal forma que los datos con patrones similares se agrupan en regiones. El modelo SOM se compone de 2 capas de neuronas: la capa de entrada, el cual recibe los datos del espacio de entrada y las transmite a la capa de salida, donde esta última se encarga de procesar la información y formar las regiones en el mapa. Cada neurona de entrada i está conectada con cada neurona de salida j mediante un peso wij, el cual se almacena en un vector de pesos wj llamado vector de referencia o codebook [19]. La Fig. 4 ilustra un ejemplo de agrupación de datos (neuronas) en un mapa bidimensional, donde la intensidad de color de cada vector de peso (círculos) representa la cantidad de neuronas que compiten, y las clases se representan con los colores.
Para este artículo, el algoritmo Batch se implementa como una variante del entrenamiento de SOM, debido a que presenta menor costo computacional y resultados similares. SOM es implementado en MATLAB usando la SOM Toolbox 2.0 [20], con el fin de obtener el resultado de cada latido y visualizar su distribución en el mapa de pesos.
2.7 Validación
Para elegir los mejores parámetros de los algoritmos, se evalúa cada uno de estos con el conjunto de validación cruzada; luego se asignan las clases a los latidos para calcular las medidas de especificidad, sensibilidad y F1 score [5] para estimar el desempeño de los algoritmos. La sensibilidad es una medida relacionada con el porcentaje de pacientes que se detectaron con latidos PVC, mientras que la especificidad es una medida relacionada con el porcentaje de pacientes sin afección que se detectaron; donde un algoritmo debe ser capaz de detectar latidos PVC y diferenciarlos de latidos normales. Por lo tanto se usa la medida F1 score, el cual es un promedio ponderado entre la precisión y la sensibilidad, dando información del rendimiento general del algoritmo [15]. Este proceso se realiza gracias a que las bases de datos del MIT-BIH contienen las etiquetas de los latidos.
2.8 Medida del costo computacional
La medición del tiempo de ejecución se toma como indicador del costo computacional. Para ello se mide el tiempo del algoritmo de predicción, ya que éste es el que se lleva a las aplicaciones, y no el entrenamiento. Se usa el comando tic-toc de MATLAB para realizar esta medida, usando una CPU con un procesador de 1.65 GHz AMD E-450 APU con 3GB de RAM y sistema operativo Windows 8 de 32 bits.
III. RESULTADOS Y DISCUSIÓN
3.1 Resultados
En las Tablas I y II se muestra el desempeño de los algoritmos Kmeans y SOM respectivamente. El parámetro de Kmeans es la función distancia, la cual calcula la distancia métrica entre los centroides y cada uno de los datos; los parámetros de SOM están relacionados a la forma en que se entrena el mapa, como la duración del entrenamiento y el tamaño del mapa, además de los vectores más cercanos (en inglés BMU, Best matching Unit) entre el espacio de entrada y los codebooks. Por defecto, el algoritmo ajusta una malla de 20x20, BMU=1 y con un entrenamiento no muy extenso.
Para Kmeans, el espacio híbrido obtuvo el mejor resultado con una especificidad del 97,41 %, una sensibilidad del 92,94 %, un F1 score de 0,9507 y un tiempo de ejecución de 18,71 µs por latido. Al igual que Kmeans, SOM obtuvo el mejor resultado con el espacio híbrido, con una especificidad del 96,22 %, una sensibilidad del 95,04 %, un F1 score del 0,956 y un tiempo de ejecución de 79,41 µs por latido.
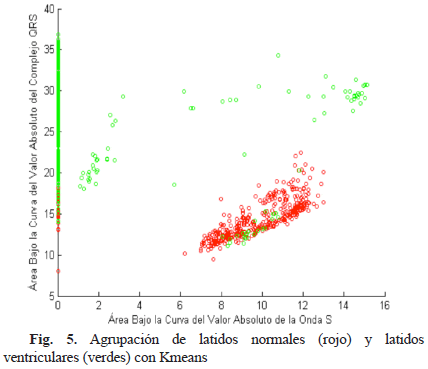
Mientras que los parámetros de desempeño muestran los resultados de forma cuantitativa, facilitando la comparación detallada entre los algoritmos, las interfaces muestran de forma cualitativa como quedan agrupadas las clases de latidos en un plano determinado. Por esta razón, en la Fig. 5 se ilustra la agrupación de latidos luego de implementar Kmeans, comparando las dimensiones del espacio híbrido correspondientes al área del complejo QRS y el área de la onda S de la señal ECG; mientras que en la Fig. 6 se ilustra la agrupación de los latidos luego de implementar SOM con el espacio híbrido, en un mapa bidimensional con celdas hexagonales.
3.2 Discusión
Un espacio al tener mayor número de dimensiones, suele tener características redundantes, además su costo computacional aumenta. Por esta razón, con el espacio híbrido, el cual tiene características muy distintivas, obtiene un costo computacional reducido y resultados mayores al 90 % para ambos algoritmos. Es importante que ambos parámetros de especificidad y sensibilidad arrojen valores correspondientes, ya que no es eficiente que uno de los dos sea mucho menor al otro, como ocurre en la Tabla 1 con el espacio temporal, el cual arroja una especificidad del 99,52 % pero una sensibilidad del 48,94 %; por esta razón, el F1 score es el parámetro a destacar, ya que es el promedio ponderado de éstos parámetros.
En la Tabla 2 se observa que SOM arroja resultados menores al 90 % en espacios con dimensiones mayores a 7; esto se debe a que cada característica aporta una distribución de pesos en el mapa, dando indicio de la celda en la que debe competir el latido; por ende si se tiene mayor número de características, cada latido tiene más celdas en las que compite y por lo tanto el BMU no consigue el mayor rendimiento del algoritmo. También ocurre que con el espacio temporal, la especificidad es mucho mayor a la sensibilidad.
En la Fig. 5 se observa que con el método de kmeans los latidos normales (rojos) tienen valores similares, forman un grupo más compacto, mientras que los latidos PVC (verdes) están más dispersos, aunque se observa claramente una separación de clases. Los datos que no fueron separados indican el porcentaje de error del algoritmo.
En la Fig. 6 se observa que con el método de SOM, los latidos PVC tienen más pesos en el mapa, aunque están separados de los pesos con los latidos normales.
El resultado obtenido en las Fig. 5 y Fig. 6 concuerda con los resultados cuantitativos registrados en las Tablas 1 y 2, donde el porcentaje de error se ve reflejado en los latidos agrupados en clases erróneas.
IV. CONCLUSIÓN
La ventaja de este tipo de métodos presentados es que no se ven afectados por el sobre-entrenamiento, ya que son algoritmos enfocados en encontrar patrones dentro del espacio de entrada, es decir que para pacientes externos al grupo entrenado, la eficiencia en la detección no disminuye radicalmente como puede ocurrir en algoritmos supervisados. Por lo tanto son apropiados para encontrar anomalías en la señal cardiaca, razón por la cual son muy implementados en aplicaciones como holters.
Kmeans obtuvo una especificidad mayor y un costo computacional menor que el algoritmo SOM, aunque este último tiene una sensitividad y F1 score mayor, además SOM obtuvo resultados mayores al 90% con otros espacios de características, a diferencia de Kmeans, lo que lo hace un algoritmo más adaptativo.
Debido a que el tiempo de ejecución de estos algoritmos es mucho menor a la duración de cada latido (mayor a 100 ms), se propone para trabajos futuros la implementación del algoritmo SOM en sistemas embebidos para aplicaciones de dispositivos portátiles como los monitores holters, permitiendo registrar más horas de la señal cardíaca del paciente sin que implique más trabajo para el médico especialista que realiza el estudio. Este tipo de tecnologías conlleva que los largos registros se puedan realizar fuera del hospital, y en equipos muy portables y cómodos para los pacientes.
Para trabajos posteriores se propone adicionar otras clases de latidos a considerar y métodos con una caracterización diferente, ya que la morfología de la señal puede variar entre múltiples tipos de latidos patológicos, con variaciones con respecto a los latidos normales y PVC, que es donde el espacio hibrido se enfoca.
REFERENCIAS
[1]. Sormo L., Laguna P. Bioelectrical Signal Processing in Cardiac and Neurological Applications. Elsevier Academic Press, 2005. [ Links ]
[2]. Carmen E., About.com. Enfermedades del corazón. ¿Qué es un Holter? (15 mar. 2013). Consultado el 25 de julio de 2013 en: http://enfermedadescorazon.about.com/od/Diagnostico/a/que-Es-Un-Holter.htm. [ Links ]
[3]. Dayan P. Unsupervised Learning. p. 1-7. Consultado el 26 de julio de 2013 en: http://www.gatsby.ucl.ac.uk/~dayan/papers/dun99b.pdf. [ Links ]
[4]. Nieto N., Orozco M. Señales Senosoidales. 38, 381-386, 2008. Consultado el 15 de mayo de 2013 en: http://revistas.utp.edu.co/index.php/revistaciencia/article/view/3809/1975. [ Links ]
[5]. Alpaydin E. Introduction to Machine Learning, 2nd ed. Massachusetts Institute of Technology, 2010. [ Links ]
[6]. Bock H.-h., Aachen R., A history of the k-means algorithm. p. 12, 2007. Consultado el 15 de mayo de 2013 en: http://www.modulad.fr/dac/Slides/Bock/Ahistoryofthek-means.pdf. [ Links ]
[7]. Bock H.-h., Aachen R. A history of the k-means algorithm. p. 9, 2007. Consultado el 15 de mayo de 2013 en: http://www.modulad.fr/dac/Slides/Bock/Ahistoryofthek-means.pdf. [ Links ]
[8]. Rodríguez J. L., Cuesta D., Castellanos G., Valencia U. P. D. Dynamic Time Warping y k-means modificado. 4to Congreso Latinoamericano de ingeniería biomédica: soluciones de bioingeniería para la salud en América Latina, 1173-1177, 2007. [ Links ]
[9]. Mehta S., Shete D., Lingayat N., Chouhan V. K-means algorithm for the detection and delineation of QRS-complexes in Electrocardiogram. Irbm, 31(1), 48-54, feb. 2010. Consultado el 5 de mayo de 2013 en: http://linkinghub.elsevier.com/retrieve/pii/S1959031809001079. [ Links ]
[10]. Kinoshita H., Yoshizawa M., Inagaki M., Uemura K., Sugimachi M., Sunagawa K. Development of an algorithm for detection of fatal cardiac arrhythmia for implantable cardioverter-defibrillator using a self-organizing map. Engineering in Medicine and Biology Society 5-7, 2006. EMBS '06. 28th Annual International Conference of the IEEE, Aug. 30 2006. Consultado el 5 de mayo de 2013 en: http://www.ieeeexplore.us/xpl/login.jsp?tp=$arnumber=4462770$url=http%3A%2F%2Fwww.ieeeexplore.us%2Fxpls%2Ficp.jsp%3Farnumber%3D4462770. [ Links ]
[11]. Scholarpedia. K-nearest neighbor (2009). Consultado el 5 de mayo de 2013 en: www.scholarpedia.org/article/K-nearest neighbor. [ Links ]
[12]. Wen C., Lin T.-C., Chang K.-C., Huang C.-H. Classification of ECG complexes using self-organizing CMAC. Measurement, 42 (3), 399-407, (abr. 2009). DOI: 10.1016. Consultado el 5 de mayo de 2013 en: http://linkinghub.elsevier.com/retrieve/pii/S0263224108001292. [ Links ]
[13]. Yeh M.F., Chang K.C. A self-organizing CMAC network with gray credit assignment. IEEE Transactions on Systems, Man, Cybernetics: Part B 36 (3) 623-635, 2006. [ Links ]
[14]. Goldberger AL, Amaral LAN, Glass L., Hausdorff JM, Ivanov PCh, Mark RG, Mietus JE, Moody GB, Peng C-K, Stanley HE. Physiobank, physiotoolkit, and physionet: Components of a new research resource for complex physiologic signals. (2013). Consultado el 20 de abril de 2013 en: http://circ.ahajournals.org/content/101/23/e215.full. [ Links ]
[15]. Powers D., Evaluation: from precision, recall and F-measure to roc, informedness, markedness and correlation. Journal of Machine Learning Technologies 2 (1), 37-63, 2011. Consultado el 13 de julio de 2013 en: http://www.bioinfo.in/upload_les/1303131155211JMLT.pdf. [ Links ]
[16]. Bustamante Arcila C., Duque Vallejo S., Orozco-Duque A., Bustamante Osorno J. Development of a segmentation algorithm for ecg signals, simultaneously applying continuous and discrete wavelet transform. 44-49, sep. 2012. Consultado el 20 de mayo de 2013 en: http://ieeexplore.ieee.org/xpl/login.jsp?tp=$arnumber=6340555$url=http%3A%2F%2Fieeexplore.ieee.org%2Fxpls%2Fabsall.jsp%3Farnumber%3D6340555. [ Links ]
[17]. Dib N., Benali R., H. S. Z. Delineation of the complex qrs and the t-end using wavelet transform and surface indicator. Biomedical Engineering, 83-86, 2011. [ Links ]
[18]. Smola A., Vishwanathan S. Introduction to machine learning, 2008. Consultado el 18 de mayo de 2013 en: http://alex.smola.org/drafts/thebook.pdf. [ Links ]
[19]. Marin J., Kohonen (SOM), 1-13. Consultado el 18 de mayo de 2013 en: http://halweb.uc3m.es/esp/Personal/personas/jmmarin/esp/DM/tema5dm.pdf. [ Links ]
[20]. CIS, Som toolbox 2.0 downloadables. Consultado el 20 de abril de 2013 en: http://www.cis.hut._/projects/somtoolbox/download/. [ Links ]