Introducción
Numerosas investigaciones en ciencias básicas han logrado una mayor comprensión del genoma humano. A principios de este siglo, se reportó su secuenciación completa, gracias al trabajo colaborativo de muchas instituciones en el conocido Human Genome Project (HGP) (1,2). El objetivo de este proyecto fue ampliar el conocimiento que se tenía sobre la evolución humana, la etiología de las enfermedades y la interacción entre el medio ambiente y la herencia (1). Tal vez el hallazgo más considerable ha sido el número de genes que conforman el genoma humano, aproximadamente una cuarta parte del que se esperaba. Así mismo, se evidenció que menos del 2 % de la secuencia del genoma humano codifica para exones de proteínas (1,2). El HGP, como primer proyecto biológico a gran escala, allanó el camino para numerosas empresas basadas en consorcios.
En el 2003, un consorcio de grupos de investigación inició el proyecto Encyclopedia of DNA Elements (ENCODE), cuyo propósito fue describir la funcionalidad del genoma anteriormente secuenciado, incluyendo los elementos que actúan en las proteínas y el ARN, al igual que los elementos regulatorios que controlan la actividad de cada gen (3). Teniendo en cuenta que únicamente un porcentaje del genoma muy limitado codifica proteínas, ¿qué codifica el resto? De acuerdo con lo reportado por el ENCODE, en el 2012 se evidenció que aproximadamente el 76 % del ADN del genoma se transcribe en algún tipo de ARN (4). Dentro de estas especies deARN, existen los ARN no codificantes o non-coding RNA (ncRNA), que tienen tan diversas funciones como regular la expresión génica de la transcripción, procesamiento de ARN y traducción. Debido a la notable variedad de funciones biológicas, se les pueden considerar líderes de una revolución, pues han forjado nuevos dogmas, anulando afirmaciones moleculares previamente establecidas (5). Estos ncRNA incluyen 8800 moléculas de ARN pequeño (small RNA molecules) y 9600 de ARN largo no codificante (long noncoding RNA o lncRNA) (4). Los lcnRNA regulan los estados de la cromatina (complejo de ADN y proteínas), procesos de transcripción, incluyendo actividad asociada con regiones enhancer (6). Otro de los reguladores de la expresión génica, dentro de la “revolución de los ARN”, comprende a los ARN micro (micro-ARN o miRNA), como reguladores citoplasmáticos de la expresión génica (7).
Dentro de los alcances de esta revolución, en agosto de 2018 la Administración de Medicamentos y Alimentos (FDA, por su sigla en inglés) de Estados Unidos anunció la aprobación de un medicamento basado en los procesos postranscripcionales de ARN interferente (RNA interference o RNAi) utilizando tecnología de silenciamiento de genes (8). Es un hito para el campo, que ha luchado durante más de década y media, desde que se descubrieron los ARNi, para demostrar su valor en la clínica.
A través del descubrimiento del ARNi, la bioquímica farmacéutica ha podido utilizar estos principios para bloquear la expresión de distintos genes y utilizarlos contra los tipos más severos de cáncer. En el Instituto de Tecnología de Massachusetts, investigadores están trabajando en el diseño de nanopartículas que puedan transportar ARN pequeño de interferencia o small interfering RNA (siRNA) mediante ingeniería de ácidos nucleicos en combinación con diseño de polímeros, lo cual está generando una plataforma de terapias basadas en ARNi (9).
Otra cooperación importante de este siglo ha sido el consenso al que se llegó en la reunión en Cold Spring Harbor, en el 2008, acerca de la definición de “rasgos epigenéticos”, la cual los define como un fenotipo heredable estable que resulta de cambios en un cromosoma sin alteraciones en la secuencia de ADN (10).
A pesar de que con estos descubrimientos se ha corrido la frontera del conocimiento desde las ciencias básicas, los profesionales de la salud no parecen tener suficiente entendimiento de estos conceptos (11,12). Más allá de ser un compendio de datos interesantes, la verdadera importancia de estos hallazgos radica en su posible aplicabilidad en la clínica, por lo cual es muy importante que el personal que podría estar implementando estos descubrimientos tenga un entendimiento básico de sus principios.
El campo de investigaciones básicas asociadas a la medicina avanza diariamente, y cada vez se encuentran más métodos terapéuticos para todo tipo de enfermedades (13). Por ejemplo, la epigenética ayuda a orientar diagnósticos e incluso está involucrada en el tratamiento de algunas enfermedades. Hoy, una nueva clase de medicamentos epigenéticos, conocidos como epi-drugs, actúan en la cromatina e intervienen en el control de la expresión génica (14). El interés en estos nuevos medicamentos ha crecido vertiginosamente (15).
Cada uno de los más de 200 tipos celulares en nuestro cuerpo interpreta la información del genoma de manera diferente para realizar las funciones vitales. Posiblemente, cualquier alteración conduce a su manifestación en una patología. Este artículo se escribió con el propósito de resumir algunos aspectos importantes de la regulación de la expresión génica; para que quien lo lea sea capaz de aprehender estos conceptos y, sobre todo en el caso del personal de la salud, aprecie su posible aplicabilidad en su vida laboral.
Expresión génica diferencial
Cada ser humano está constituido por alrededor de un billón de células (se estima que el cuerpo humano tiene entre 1012 a 1016 de células) (16). Cada una de ellas posee el mismo genoma, y este contiene toda la información necesaria para construir y mantener un organismo. El genoma está conformado por aproximadamente 3.000.000.000 pares de bases (1), que constituyen el ADN, y codifican alrededor de 19.000 genes (17).
Dentro de las secuencias de ADN se encuentran los genes, secuencias específicas de ADN responsables por las características físicas y heredables de un organismo. Un gen codifica para una proteína con una función específica, por lo cual se puede encontrar en estado activo o inactivo, y dependiendo de los genes que estén activos en una célula, se establece su estructura, función y respuesta al medio ambiente (18,19). La identidad celular, o más específicamente lo que distingue una célula de otra, es controlada en gran parte por la acción de factores de transcripción que reconocen y se unen a secuencias específicas del genoma, que consecuentemente regulan la expresión génica (20). En el genoma humano se han descrito más de 1600 factores de transcripción, donde aproximadamente la mitad de los factores de transcripción codificados en el genoma humano se expresan en cualquier tipo celular (21,22).
La transcripción de genes específicos define el tipo celular, por lo cual la expresión génica diferencial se refiere a los mecanismos que permiten que cada tipo celular sea distinto a otro, basado en una combinación única de genes que estén activos o sean expresados. La producción selectiva de proteínas permite generar diversidad celular. La dinámica de la expresión génica, además, permite a una célula que conforma un tejido responder a señales intrínsecas o extrínsecas provenientes del medio ambiente, con el fin de regular procesos celulares como el ciclo celular, responder en situaciones favorables o de estrés o establecer su destino celular. Estos mecanismos le permiten a la célula adaptarse para cumplir con su ciclo de vida. Consecuentemente, asegurando la expresión correcta de genes específicos, el sistema regulador transcripcional desempeña un papel central en el control de procesos biológicos (21).
Muchos mecanismos regulatorios que permiten el acceso al ADN, la síntesis de ARNm y su procesamiento, así como la producción de proteínas, conocido como el proceso de traducción y sus modificaciones, permiten la expresión génica diferencial (23). Los mecanismos de traducción, aunque relevantes, no forman parte del enfoque del presente artículo, por lo cual se sugiere leer a Alberts et al. (24), Clancy y Brown (25) y Schaefke et al. (26).
La expresión génica que le otorga la identidad a distintos tipos celulares en el proceso del desarrollo de un ser humano inicia en la primera semana posconcepción y se mantiene durante la vida de un individuo (27). Para lograr esto, primero se fusionan los gametos femenino (óvulo) y masculino (espermatozoide) durante la fertilización, proceso mediante el cual se unifica la información genética proveniente de la madre y del padre, formando un cigoto con un genoma nuevo, que tiene características genéticas de ambos padres. No obstante, al inicio, el genoma del nuevo individuo no está activado; se deben llevar dos a tres divisiones del cigoto para que esto se dé lugar. Hacia el tercer día posconcepción el proceso de transcripción es evidente (27). Entre cuatro y ocho células, las células comparten prácticamente la misma identidad, donde los genes activos son en mayor parte los mismos. Después del quinto día, se comienzan a activar e inactivar genes, de manera que se permite la formación de dos tipos celulares diferentes (28). El objetivo es sostener dos poblaciones para la formación de un individuo: las células del trofoblasto, que darán lugar a la placenta, y las células del embrioblasto, que formarán el embrión. Para alcanzar esta meta únicamente se deben seleccionar de todo el genoma los genes que cumplan esta función (29).
Así es como dependiendo de la expresión génica diferencial —la activación o inactivación de genes— cada célula adquirirá el fenotipo que la identifica y le proporciona su función. Una vez elegidos los genes que deben ser transcritos, se lleva a cabo la síntesis de ARNm. Después de un proceso de maduración (eventos que se describen a continuación), los transcritos deben salir del núcleo y mediante la traducción del ARNm finalmente se originan las proteínas necesarias para que cada célula tenga su identidad y función respectiva.
Los procesos de transcripción diferencial inician en el tercer día del desarrollo; pero persisten durante toda nuestra vida, otorgándoles a las células su identidad, función y respuesta al medio ambiente en situaciones fisiológicas; mas cuando hay desregulaciones en la expresión de genes se manifiestan las patologías (20,21). De hecho, algunos estudios se enfocan en establecer correlaciones entre niveles de expresiones génicas diferenciales y características clínicas (30). Para mencionar algunos ejemplos, se han realizado investigaciones que relacionan procesos de transcripción diferencial con condiciones como malformaciones vasculares en el cerebro, respuesta a tratamiento en pacientes hispánicos con leucemia y prognosis en cáncer pancreático, entre otros, con el propósito de encontrar biomarcadores que puedan ser utilizados en un futuro para realizar la búsqueda activa de diferentes patologías, estratificación de riesgo y determinar el pronóstico de las mismas (31,32,33).
Regulación epigenética de la expresión génica
El genoma puede tener una longitud aproximada de 2 m y se encuentra almacenado en el núcleo, un organelo con un diámetro promedio de 10 µm. Para permitir que la célula responda a su medio ambiente constantemente cambiante, dentro de este contexto, segmentos del genoma deben estar transcripcionalmente activos o reprimidos de manera coordinada (34). Las histonas son críticas, porque parecen ser en parte responsables de facilitar o inhibir la expresión de un gen. A continuación, se detallará cómo.
En el núcleo, el ADN se encuentra empaquetado en unidades funcionales denominadas nucleosomas, las unidades básicas de la estructura de la cromatina (ADN y proteínas) (34). El nucleosoma es un complejo entre el ADN y un octámero de proteínas, que contiene dos moléculas cada una de las histonas H2A, H2B, H3 y H4 (35). A su vez, un conjunto de nucleosomas, estabilizados por la histona H1, se compactan adicionalmente en solenoides (figura 1) para empaquetar el ADN dentro del núcleo (36).
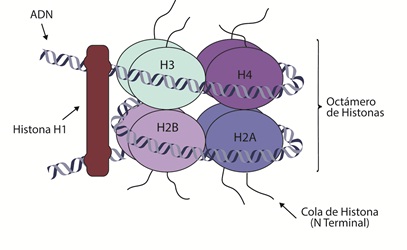
Figura 1 NucleosomaEs la unidad fundamental de la cromatina, la estructura mínima de condensación del ADN, que consiste en la secuencia de ADN que forma un complejo alrededor del núcleo de histonas. El nucleosoma contiene un núcleo compuesto por ocho histonas (H3, H4, H2B y H2A) sobre el cual se enrolla el ADN. La histona H1 es una histona de asociación que se encuentra por fuera del nucleosoma y es responsable de la condensación de la cromatina. Cada histona (menos la H1) tiene un N-terminal también conocido como la “cola” de la histona.
La conformación del nucleosoma dependiente de H1 inhibe la transcripción de genes en células somáticas, empaquetando nucleosomas adyacentes en arreglos que previenen el acceso por parte de factores de trascripción y ARN polimerasa (37,38). La regulación de la estructura de la cromatina es un proceso complejo que está conformado por una serie de modificaciones epigenéticas. Hoy, el modelo propuesto por Weintraub, en 1984, es más intrincado (38), a causa del hallazgo de otras proteínas que interactúan con H1. Se ha descrito que PTEN mantiene la condensación de la cromatina mediante interacciones con H1 (39). Por lo tanto, las regiones de cromatina altamente empaquetada se llaman heterocromatina, y en regiones donde la cromatina se encuentra relajada se denomina eucromatina, donde se puede dar el proceso de transcripción. Una manera de lograr la expresión génica diferencial es mediante la regulación de estados de eucromatina o heterocromatina.
La interacción entre ADN y proteínas desempeña un papel importante en prácticamente cada proceso celular, incluyendo la regulación de la expresión diferencial de genes (40). La estructura del ADN tiene una carga negativa, debido a los grupos fosfato en los esqueletos pentosa-fosfato de la estructura. Por otro lado, las histonas H2A, H2B, H3 y H4 están compuestas por un dominio globular en el extremo carboxi-, y por unas regiones conocidas como “colas”, que se encuentran en el extremo aminoterminal. Estas colas tienen un alto contenido de los aminoácidos lisina y arginina, que les brindan una carga positiva. La compactación de la cromatina ocurre debido a la interacción entre las cargas negativas del ADN y las positivas de los aminoácidos, principalmente ubicados en las colas de las histonas H3 y H4 (19,20) (figura 1).
Para iniciar el proceso de expresión diferencial, las proteínas que van a llevar a cabo la transcripción deben tener acceso a los genes. El primer paso es a través de procesos epigenéticos, mediante la remodelación de la cromatina. La epigenética son los cambios funcionales del genoma que no involucran modificaciones en la secuencia de los nucleótidos, pero tienen acceso al ADN para ser transcrito (41). De esta manera se integran señales intrínsecas del genoma con los del medio ambiente (42).
Ya en el siglo XIX, Lamarck describió cambios heredables y además influenciados por el medio ambiente (43). Evidencia emergente indica que tanto experiencias ancestrales, al igual que provenientes de los progenitores, como comportamiento de crianza, estrés social y hábitos nutricionales pueden afectar las funciones metabólicas, las fisiológicas y las celulares de un organismo. En algunos casos se pueden transmitir a través de varias generaciones mediante la epigenética. Es posible modular las modificaciones epigenéticas a través de metilaciones del ADN, modificaciones de las histonas y ARNs no codificantes pequeños (small non-coding RNAs) (43).
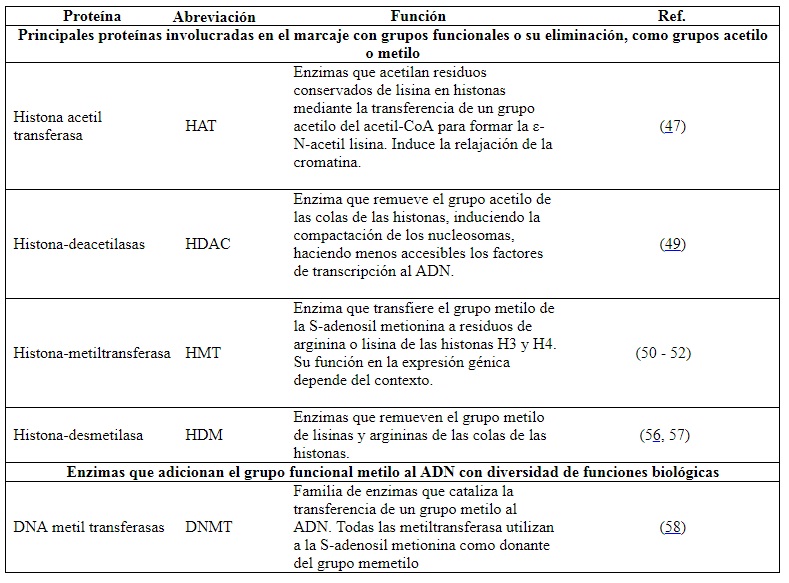
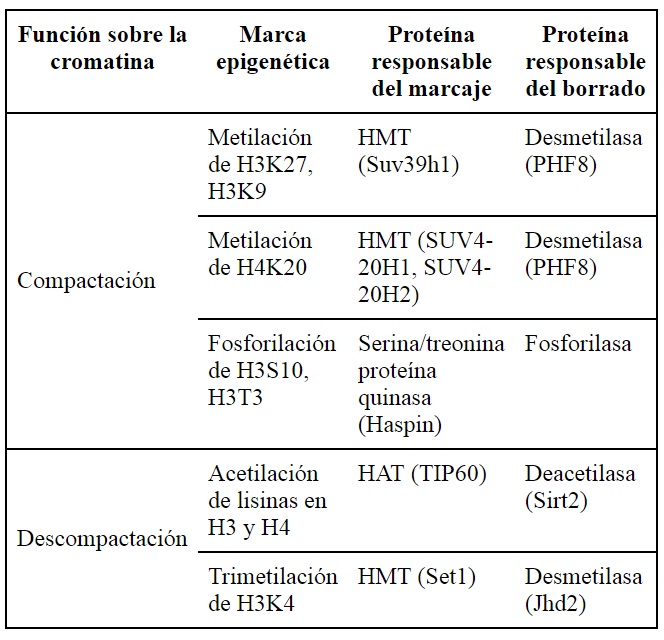
La expresión génica diferencial inicia con modificaciones sobre los aminoácidos de las colas de las histonas, lo que permite la remodelación de la cromatina (figura 2). El objetivo es tener acceso a las secuencias de ADN a ser transcritas, por las proteínas requeridas para llevar a cabo la transcripción (44). La represión o activación epigenéticas están principalmente controladas por la adición o remoción de grupos acetilo o metilo sobre estos aminoácidos (45); por esto, se requieren enzimas específicas que lleven a cabo estos procesos (46).
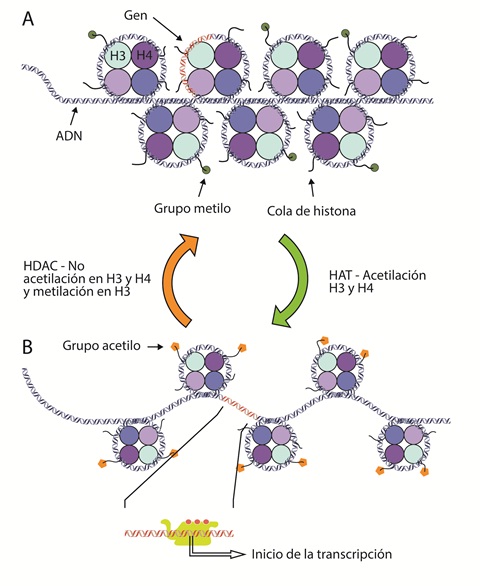
Figura 2 Esquema de heterocromatina y eucromatina.A) La heterocromatina está mayormente hipoacetilada, generando complejos de represión de la expresión génica. Adicionalmente, se ha descrito que la metilación en las lisinas (su símbolo de la IUPAC es K) de la lisina 9 (K9), la K20 y K27 de la histona H3 puede estar asociada a condensación de la cromatina. B) La remodelación de la cromatina está orquestada por la acción de factores de transcripción y enzimas que modifican las colas de las histonas como las histona acetiltransferasa (HAT). La lisina 4 de la histona H3 se puede encontrar metilada, permitiendo la expresión de un gen, mediante la maquinaria y procesos asociados.
La histona-acetiltransferasa (HAT, EC 2.3.1.48) adiciona grupos acetilo (de carga negativa) a las colas de las histonas, particularmente a H3 y H4. Esta nueva carga negativa neutraliza la carga positiva de la lisina y la arginina y relaja la cromatina (47) (figura 2B). La acetilación reduce la carga neta positiva de las histonas, debilitando la interacción entre histonas y el ADN. Otras proteínas de remodelación se unen a residuos de lisina acetilada, reconfigurando los nucleosomas para exponer otras secuencias de ADN a la unión de factores de transcripción y otras proteínas e iniciar la transcripción (48).
En contraposición, la histona-deacetilasa (HDAC, EC 3.5.1.98) remueve el grupo acetilo de las colas, haciendo que los nucleosomas se compacten y consecuentemente imposibiliten la transcripción (49). Por otro lado, la adición de un grupo metilo mediante la histona-metiltransferasa (HMT, EC 2.1.1.43) puede ocurrir en varios residuos básicos de las histonas. Dependiendo del grado de metilación y de la ubicación del residuo metilado, se da lugar a diferentes resultados funcionales (50) (figura 2A). El efecto dependerá del aminoácido metilado y de la presencia de grupos acetilo en la vecindad (51,52). Por ejemplo, la acetilación en las colas de las histonas H3 y H4, además de la trimetilación en la lisina 4 de la cola de la histona H3, permite la expresión de genes cercanos (53,54). Por el contrario, la ausencia de acetilación en las colas de las histonas H3, H4 y la metilación de H3 en la lisina (K) en la posición 9 o K27 y en H4K20, es indicativo de heterocromatina (55). En síntesis, el proceso de metilación de histonas es dinámico. Las marcas de metilo pueden ser adicionadas o removidas por enzimas específicas. Otras proteínas las reconocen y se unen a los residuos metilados para efectuar resultados en el fenotipo (50).
Los grupos metilo también pueden ser eliminados por la enzima histona-desmetilasa (HDM, EC 1.14.11.27), al igual que la HDAC elimina los grupos funcionales acetilo (56). La actividad de esta enzima está regulada por niveles de expresión, mediante su acción en sitios específicos en el genoma y modificaciones postraduccionales (57).
En síntesis, si la cromatina se encuentra condensada, es imposible que la maquinaria necesaria para la transcripción de un gen tenga acceso a este (44). Por esta razón, las modificaciones sobre las colas de las histonas son indispensables para que la expresión génica diferencial ocurra, pues ponen a disposición todo el material genético, permitiendo la transcripción (cuando se relaja) o inhibiéndola (cuando se condensa). Las tablas 1 y 2 resumen las enzimas y las funciones para llevar a cabo estos procesos (47,49,50,51,53,54,55,56,57,58).
Modificaciones epigenéticas sobre el ADN: metilación
Otra manera de regular la expresión génica diferencial es mediante la metilación de nucleótidos directamente sobre la secuencia del ADN. Así como las acetilaciones y metilaciones sobre las colas de las histonas pueden resultar en la remodelación o condensación de la cromatina (causando la inactivación o activación de un gen), las metilaciones de los nucleótidos, en especial sobre las citosinas en el ADN, inhiben la expresión génica (59). En humanos, algunas de estas metilaciones se encuentran sobre citosinas que se ubican en regiones promotoras, conocidas como islas CpGs (60).
Los genes tienen una estructura típica constituida por regiones de control y una unidad transcripcional (tabla 3). Dentro de las regiones de control existe la región promotora, en la cercanía del inicio de la transcripción. Allí se unen proteínas que posicionan la ARN polimerasa II para dar inicio a la transcripción. La región promotora del tipo isla CpG tiene un alto contenido de dinucleótidos citosina-guanina; de ahí su nombre. Generalmente, se encuentra secuencia arriba del gen en la hebra de ADN (figura 3) y promueve la unión de la maquinaria de transcripción.
Tabla 3 Componentes de un gen. Se describen los componentes del gen y los que influencian la expresión génica diferencial
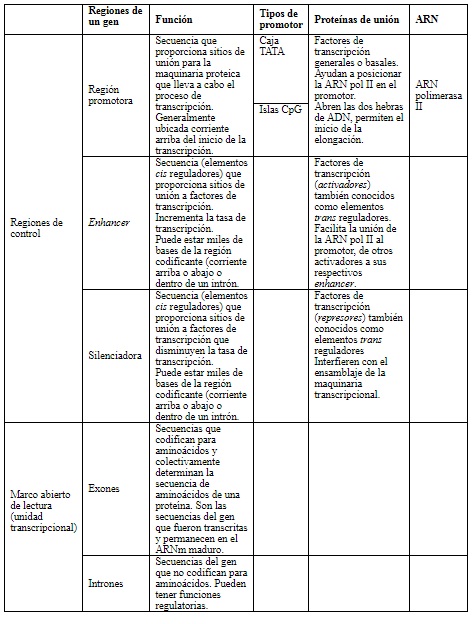
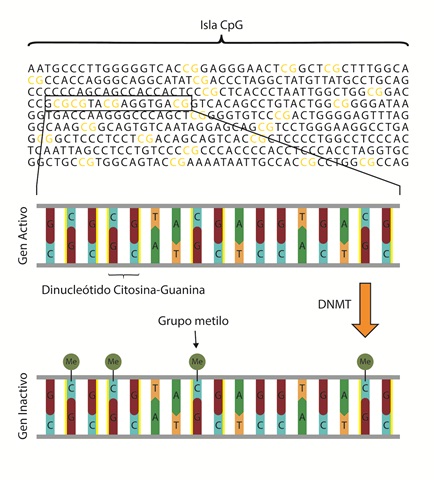
Figura 3 Metilación de citosinas en islas CpG. Las islas CpG corresponden a un tipo de promotor rico en dinucleótidos citosina-Guanina. Para la inactivación de un gen la DNMT metila la citosina del dinucleótido CpG.
Las metilaciones en las islas CpG regulan la expresión génica mediante el reclutamiento de proteínas asociadas a la represión de la expresión de un gen. Por otro lado, la metilación en citosinas en la región de control tipo promotora inhibe el acoplamiento de proteínas para dar inicio con la transcripción. Otro mecanismo evita la unión de los factores de transcripción a sus regiones de control tipo enhancer (61). La metilación sobre citosinas del ADN funciona como una señal que permite a proteínas reconocer regiones del ADN que deben ser silenciadas. De esta manera, se facilita el reclutamiento de otras proteínas que en conjunto pueden modificar la eucromatina en heterocromatina.
Aproximadamente, el 70 % de los promotores en humanos son del tipo islas CpG (62). Dentro de los genes regulados por este tipo de promotores están los genes constitutivos (también conocidos como housekeeping), genes tejido-específicos y genes del desarrollo (63,64). Las metilaciones sobre la citosina de una isla CpG se deben por la acción de la enzima ADN metiltransferasa (DNMT, EC 2.1.1.37) (65). Por esa razón la metilación en las citosinas de los promotores de tipo CpG es un mecanismo fundamental en la regulación de la expresión génica (tabla 1).
Por otro lado, una citosina metilada puede reclutar proteínas de unión que facilitan la metilación o desacetilación de las colas de las histonas, estabilizando los nucleosomas. Una citosina metilada en el ADN puede unir proteínas que forman complejos con capacidad de remover grupos acetilos en las histonas y adicionar grupos metilos (66). Por ejemplo, las citosinas metiladas del ADN pueden unir proteínas específicas como la metil-CpG proteína de unión 2 (MeCP2). Una vez unida la proteína a la citosina metilada, MeCP2 une HDACs y HMTs, las cuales remueven grupos acetilos y adicionan grupos metilos, respectivamente, en las colas de las histonas. Como resultado, los nucleosomas forman complejos altamente condensados, no permitiendo que se lleve a cabo la transcripción.
Lectura del gen: decodificando la información del genoma en transcrito
En la sección anterior se describió cómo las histonas sirven de guardianes, modulando el acceso de la maquinaria transcripcional a los genes mediante procesos epigenéticos. Para que finalmente se pueda expresar un gen, la secuencia específica de ADN debe ser transcrita a ARN mensajero (ARNm) en el núcleo (24). Posteriormente, este ARNm o transcrito debe ser traducido a proteína en el citoplasma mediante el proceso de traducción, ya que la proteína es la molécula funcional en el contexto celular (67). La expresión génica diferencial es, entonces, la selección de genes específicos en los cuales se llevan a cabo los procesos de transcripción y, tal vez, posteriormente de traducción (68). La regulación pos-transcripcional de la expresión génica es más intrínseca de lo que se había pensado. Un completo entendimiento de los mecanismos básicos del control postranscripcional será esencial para tener una visión holística de la expresión génica diferencial a diferentes niveles. Debido a su complejidad, no es tema de este trabajo. Para mayor entendimiento de estos procesos se sugiere leer Michlewski y Cáceres (69) y Corbett (70).
La transcripción es un proceso que se requiere para que la célula establezca su identidad dentro de un tejido y, por ende, su función. Por otro lado, le permite responder a eventos intrínsecos y/o extrínsecos, dependiendo de las señales percibidas. Por esto, dependiendo de la activación o inactivación de genes, puede haber grandes cambios en la regulación de la expresión génica.
La transcripción es llevada a cabo por la ARN polimerasa tipo II (EC 2.7.7.6) y un complejo de proteínas, los cuales son capaces de realizar una copia complementaria de una hebra del ADN del gen que se va a transcribir (71). La hebra del ARNm es de cadena sencilla, y tiene un inicio 5’ y un fin 3’. Como se había descrito, existen secuencias en el ADN que son requeridas para el proceso de la transcripción, pero que no forman parte del transcrito, como las regiones de control (tabla 3). Las regiones regulatorias (elementos reguladores en cis) pueden estar ubicadas en los extremos del gen, dentro de él o en secuencias a muchos pares de bases del inicio del gen, pero son necesarias para indicar cuándo y dónde se transcribe cada gen. En consecuencia, durante la transcripción se lee información proveniente de regiones no contiguas en el ADN (24,72).
El proceso de transcripción es un proceso específico que en un primer nivel se basa en interacciones específicas con alta afinidad de proteínas (elementos trans reguladores) con la secuencia blanco en el ADN (elementos cis reguladores) (73). Para que este proceso se inicie, se requieren señales codificadas dentro del ADN que indiquen a la ARN polimerasa II dónde iniciar. A esta región se le conoce como la región promotora (figura 4C). Las secuencias promotoras pueden ser de tipo isla CpG o caja TATA, llamada así por tener una secuencia TATAAA (secuencia consenso) a la cual se une la ARN polimerasa II. En humanos, este tipo de promotor está en aproximadamente el 24 % de los genes. Generalmente, las secuencias promotoras se ubican unos 30 nucleótidos corriente arriba de donde se une la ARN polimerasa II. Por otro lado, a las islas CpG se unen los factores de transcripción Sp1, que pueden activar o reprimir la transcripción en respuesta a estímulos fisiológicos o patológicos (74). Aproximadamente, el 76 % de los genes humanos carecen del promotor tipo TATA y son del tipo islas CpG (75).
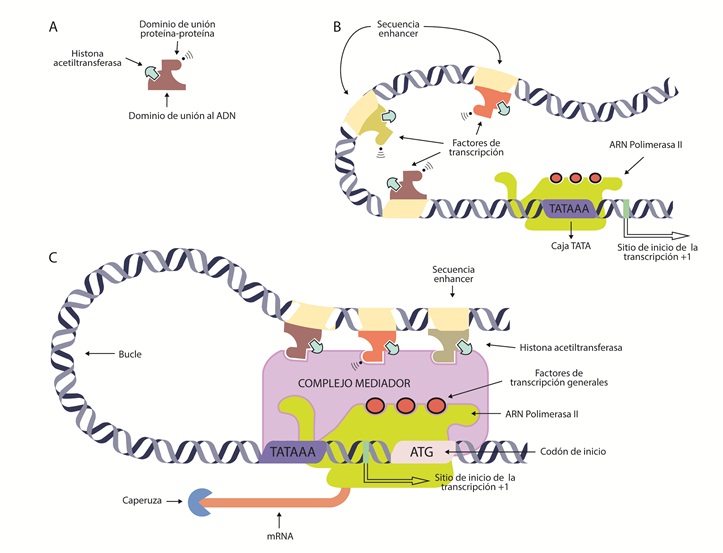
Figura 4 Expresión de un gen mediante la síntesis de mRNA.A) Factor de transcripción con sus dominios de unión para la regulación de la transcripción. B) Unión de un factor de transcripción a la región enhancer, la cual aumenta la transcripción del gen en cuestión. C) Unión de factores de transcripción al complejo mediador en asociación a ARN polimerasa II para iniciar la transcripción.
Existen dos tipos de factores de transcripción o elementos reguladores en trans-: los factores de transcripción generales, que se unen a secuencias de ADN en la región de control tipo promotora, permitiendo que la ARN pol II forme el complejo de iniciación. El otro tipo de factores de transcripción activadores o represores se unen a regiones de control tipo enhancer o silenciadora (que no permiten la expresión del gen mediante un mecanismo de represión), respectivamente; modulando la formación de un complejo de iniciación. Se explican más adelante en detalle (tabla 3).
Los factores de transcripción generales (TFIIA, TFIIB, TFIID, TFIIE, TFIIF, TFIIH) se unen a la secuencia promotora en un orden específico y permiten posicionar la ARN polimerasa II en la ubicación correcta. Además, estos factores separan las dos hebras de ADN para que inicie la transcripción y liberen la ARN polimerasa del promotor y se pueda llevar a cabo la elongación, una vez iniciada la transcripción (72). La eficiencia de este proceso puede ser baja, por lo cual la cantidad de ARNm sintetizado no es muy alta.
Los factores de transcripción (activadores) reconocen regiones específicas de ADN, para controlar la cromatina y la transcripción, formando un sistema complejo que guía la expresión en el genoma (22). Son elementos clave en el control de la expresión génica y sus actividades determinan la funcionalidad celular y su respuesta al medio ambiente (21). Operan de manera coordinada para dirigir procesos celulares durante toda la vida.
Para aumentar la eficiencia del proceso, adicional a la ARN polimerasa tipo II y los factores de transcripción generales, se requiere la unión de otras proteínas, los factores de transcripción (activadores) a regiones enhancer, a fin de modular la formación de un complejo de iniciación. Cada gen contiene una variedad de enhancers en su región de control (76). En ciertos momentos es preciso aumentar la expresión de un gen o genes, en respuesta a una señal en particular, como una hormona o un factor de crecimiento (77). La combinación exacta de un factor de transcripción en una célula en particular, dado en un momento específico define cuáles genes serán transcritos y en qué tasa. Por el contrario, dependiendo del estado de la célula, el gen puede ser inactivado mediante la unión del factor de transcripción a una región silenciadora.
La ubicación de los factores de transcripción depende del tipo celular, ya que es tejido específico y de esta manera se define el fenotipo y, por ende, la función celular (78). Los factores de transcripción son proteínas que deben tener, por lo menos, dos dominios reconocibles, pero se han descrito hasta tres regiones de interacción distintas en su estructura terciaria (figura 4A): un dominio de unión al ADN, que se une específicamente a una secuencia de ADN como un elemento cis regulador dentro de la región de control, sea promotora o enhancer (figura 4B). Estas secuencias no necesariamente están en la cercanía de la región promotora; pueden estar a miles de pares de base del gen que regulan, pueden estar corriente arriba, corriente abajo o dentro de un intrón del gen que regulan. La interacción del factor de transcripción con la secuencia de ADN genera un complejo que permite establecer conexiones con otras proteínas, de manera que queden formando bucles de ADN, estableciendo en últimas una asociación con la región promotora y todas sus proteínas mediante un complejo mediador (79). Adicionalmente, como se mencionó, estas secuencias tienen como función controlar la tasa de transcripción de un promotor en tiempo y espacio de manera específica (80). Varios tipos de dominios de ADN han sido caracterizados; de hecho, se utilizan para clasificar los factores de transcripción en familias: dentro de estas se encuentran: hélice-giro-hélice, hélice-bucle-hélice, dedos de zinc y cremalleras de leucina (81,82).
Por otro lado, el otro dominio del factor de transcripción le permite la unión con otros factores de transcripción, interacción con ARN polimerasa II y el reclutamiento de proteínas modificadoras de cromatina (68), mediante la interacción con enzimas que modifican las histonas, como HAT o HMT, lo cual remodela la cromatina. Con estas acciones se da la entrada de la ARN polimerasa II, su unión al ADN y la descondensación del ADN alrededor de las histonas en regiones vecinas para su transcripción. De esta manera, los factores de transcripción pueden estabilizar el complejo de iniciación. Por último, generalmente, el factor de transcripción debe tener un dominio de interacción proteína-proteína. Así, los factores de transcripción, a través de sus dominios, forman “asociaciones” por medio de distintas proteínas que permiten que la cromatina formen bucles y traigan las secuencias y las enzimas modificadoras de histonas a la cercanía de la región promotora (figura 4C). Consecuentemente, la unión del factor de transcripción a su enhancer regula el momento en el cual se debe dar lugar la expresión de un gen en una célula específica (83).
Una vez se haya asociado la ARN polimerasa II y todos los factores anteriormente descritos, se debe iniciar el proceso de transcripción. Por convención, al primer nucleótido del gen en ser transcrito se le asigna el número +1 y su síntesis ocurre en sentido 5’ a 3’ (figura. 4C). Adicionalmente, este extremo del ARNm debe estar protegido por una caperuza, que consiste en una modificación del extremo 5’ del transcrito de ARNm, donde se le adiciona un nucleótido de guanina (G) atípico con un grupo metilo en la posición 7 (24). La función de la caperuza es crítica para la maduración y estabilidad del transcripto (84). Seguido, se encuentra la secuencia líder, también conocida como región no traducida o untranslated region (5’UTR), que se transcribe en ARNm, pero no se traduce a proteína. Esta secuencia incluye el inicio de la transcripción, designado como +1, hasta el codón que dará inicio a la traducción. Es una secuencia con funciones regulatorias del ARNm, una de ellas es determinar la tasa de la traducción. A continuación, la ARN polimerasa II transcribirá el resto del primer exón.
Los exones son secuencias que codifican para la proteína definida por el gen. Dentro de este primer exón debe estar el codón ATG, que indicará posteriormente el inicio de la traducción, que en el transcripto es AUG. Entre los exones (secuencias que se expresan) se encuentran los intrones, que son secuencias transcritas, que no codifican los aminoácidos que conformarán la proteína (85). Estas secuencias tienen funciones reguladoras; no obstante, son eliminadas en el proceso de la maduración del ARNm (tabla 3).
En el último exón del gen se debe encontrar el codón de terminación de la traducción, ya que posteriormente en el citoplasma se traduce el ARNm a proteína, el ribosoma unido al ARNm encuentra el codón de parada, se libera del ARNm y se termina la traducción. Adicionalmente, en el extremo 3’ del último exón, se encuentra la región 3’ UTR, que al igual que la secuencia 5’ UTR se transcribe, pero no se traduce. La secuencia 3’UTR comprende desde el codón de terminación de la traducción (TAA) hasta la secuencia de terminación de la transcripción. Esta región contiene la secuencia AATAAA, que es requerida para la poliadenilación. La cola poli-A es crítica para conferir al ARNm estabilidad, la capacidad de salir del núcleo y ser traducido en proteína. Por último, corriente abajo de la secuencia de poliadenilación está la secuencia de terminación de la transcripción (79).
Los intrones son secuencias que se interponen entre los exones y deben ser eliminados durante la maduración del ARNm mediante splicing alternativo (figura 5). Evolutivamente, este proceso proporciona la aparición de proteínas nuevas de la misma familia, ya que permite la combinación de distintos exones de un mismo gen. Por lo tanto, contribuye a la diversidad proteómica y la especificidad tisular (86). En humanos se estima que el 95 % de los genes sufre splicing alternativo (87,88). Para este proceso se requiere una maquinaria compleja, que utiliza una serie de ARNs pequeños nucleares, los ARNsn (small nuclear RNAs o snRNAs), que en conjunto con un número importante de proteínas auxiliares pueden eliminar un intrón a la vez (89). Estos ARNs especializados reconocen secuencias de nucleótidos que especifican la ubicación del splicing. Para seleccionar qué exones se mantienen y cuáles secuencias se eliminan son necesarios elementos de secuencia de ARN y reguladores de proteínas. Esta elección puede ocurrir en los estadios tempranos del reconocimiento del sitio de splicing. Sin embargo, puede llevarse a cabo durante otros estados del ensamblaje del espliceosoma (complejo de corte de intrones y empalme de exones), inclusive durante los cambios conformacionales (90,91). Adicionalmente, se ha reportado que el proceso de transcripción también tiene funciones regulatorias sobre el splicing (92).
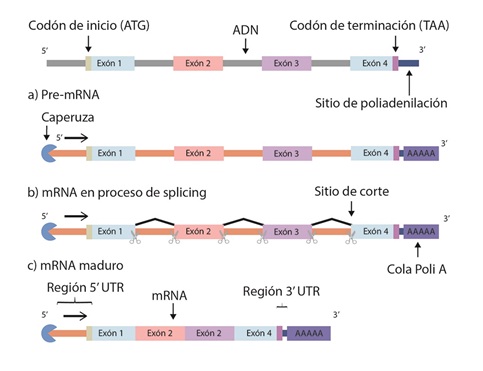
Figura 5 Maduración de ARNmEl splicing es el proceso que edita el ARNm sintetizado para que pueda salir del núcleo y ser traducido a proteína. La maduración del ARNm requiere la adición de la caperuza en el extremo 5’ y una cola de poliadenilación en el extremo 3’. Adicionalmente, los intrones deben ser removidos mediante una serie de reacciones, hasta obtener un ARNm maduro que contenga la caperuza en su extremo 5’, región 5’UTR los exones, región 3’ UTR y la cola poliadenilada, para que el transcrito pueda salir del núcleo y ser traducido e proteína en el citoplasma. Codón de inicio hace referencia a la secuencia que se leerá en el citoplasma cuando se lleve a cabo la traducción del ARNm por los ribosomas en proteína, en el transcrito es AUG. Codón de terminación se refiere a la secuencia que dará lugar a la terminación de la síntesis de la proteína en el citoplasma.
La transcripción de un gen requiere la coordinación de diversas proteínas en distintos momentos. Este transcrito puede salir del núcleo a ser traducido en proteína una vez se hayan llevado a cabo las modificaciones necesarias, como la adición de la caperuza, la eliminación de los intrones y la adición de la cola poli-A. La expresión génica diferencial para un gen ocurre mediante la selección de exones específicos para generar proteínas con funciones diferentes. Por ejemplo, el gen del colágeno 2 puede producir dos ARNm maduros. El transcrito que retiene el exón 2, conocido como COL2A, codifica para una proteína que es sintetizada por células que se van a diferenciar en condrocitos (93); mientras que el ARNm que no lo retiene se conoce como COL2B y es sintetizado por condrocitos maduros (94). Estos principios descritos han servido de base para adelantar estudios que permitan aplicaciones en la clínica de muchos de los paradigmas biológicos con fines diagnósticos o terapéuticos.
Aplicaciones clínicas de los factores de transcripción
Debido a que los factores de transcripción controlan cada proceso fisiológico dentro de la célula, las posibilidades diagnósticas y terapéuticas de los factores de transcripción en la clínica son de gran alcance (95). Por ejemplo, en cáncer se han identificado una serie de factores de transcripción linaje específicos que forman parte de cambios patofisiológicos y que favorecen la proliferación tumoral e invasión (96,97). Por lo anterior, los factores de transcripción tienen una gran utilidad diagnóstica en análisis histológicos de biopsias de tejidos con sospechas de cáncer como marcadores de diferentes neoplasias.
El cáncer surge de alteraciones genéticas que conducen a programas transcripcionales desregulados (20). Los reguladores moleculares de estos programas son proteínas involucradas en el control transcripcional, de las cuales cada vez se avanza más en su identificación. Con fines diagnósticos, algunos factores de transcripción son utilizados en inmunohistoquímica para determinar neoplasias. Para mencionar algunos, se encuentra el factor de transcripción tiroideo (TTF-1) y el Pax8 (98), que sirven para identificar tumores de linaje pulmonar y de tiroides. El factor de transcripción de microftalmia es un biomarcador para metástasis de melanoma (99), y los factores de transcripción E3 y EB, en carcinomas de células renales (100). El TTF-1 en cáncer de pulmón es utilizado para distinguir entre tumores primarios y metástasis, y para diferenciar entre los diferentes tipos histológicos de tumores (adenocarcinoma vs. escamocelular, carcinoma de células pequeñas vs. carcinoma de células de Merkel) (101) (figura 6).
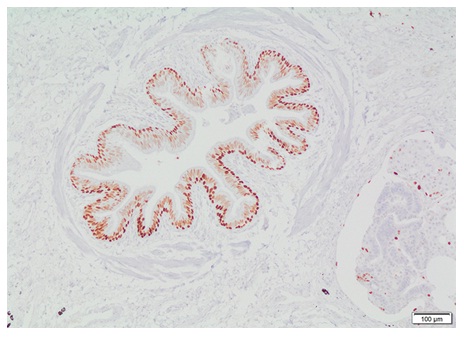
Figura 6 Factor de transcripción TTF-1 como marcador celular en patología.Células epiteliales cilíndricas positivas para TTF-1 en bronquiolo rodeado de músculo liso sin cartílago. Fotografía a 10X tomada de biopsia de pulmón con firma de consentimiento informado. El factor 1 de transcripción tiroideo (TTF-1) es un polipéptido que hace parte de la familia del factor de transcripción homeodominio (homebox). Es uno de los actores de la regulación de genes expresados en pulmón, tiroides y cerebro. Es utilizado como ayuda diagnóstica al momento de realizar pruebas inmunohistoquímicas. Debido al papel que cumple con relación a la expresión génica de los órganos mencionados, los anticuerpos TTF-1 son útiles en la diferenciación de algunos carcinomas pulmonares, como son el caso del adenocarcinoma primario de pulmón versus carcinoma metastásico de mama y mesotelioma maligno, también es útil para diferenciar carcinoma de células pequeñas del pulmón de infiltrados linfoides. La tinción inmunohistoquímica permite observar una reacción visible, y al contrateñir la muestra y aplicarla sobre un cubre portaobjetos, la interpretación del resultado se hace fácilmente con un microscopio óptico.
Por otro lado, en los laboratorios de investigación básica, desde inicios del siglo XXI se ha descrito el uso de los factores de transcripción para cambiar el fenotipo celular. Estos experimentos se han trabajado para lograr células con características diferentes a las originales y ser utilizados como modelos de enfermedad (102). En 2006, Takahashi y Yamanaka reportaron (103) un método por medio del cual es posible inducir células con propiedades de células madre a partir de células maduras. Posteriormente, utilizaron fibroblastos humanos —que son células completamente diferenciadas— las cuales fueron reprogramados a células pluripotenciales. Esta metodología involucra la expresión de genes que codifican factores de transcripción característicos de células madre pluripotenciales en los núcleos de las células maduras —por ejemplo, los fibroblastos— y como resultado se obtiene que dichas células se regresen hacia un estado pluripotente, como las células en estados preembrionarios. Estas células resultantes recibieron el nombre de células inducidas a pluripotencia. Por este trabajo, Shinya Yamanaka y John B. Gurdon recibieron el Premio Nobel de Medicina, en el 2012. Otro ejemplo de estos avances es la diferenciación in vitro de fibroblastos a células con características de neuronas mediante la aplicación de modificaciones sobre la metodología descrita, salvo que se requiere la utilización de factores de transcripción específicos para linaje neural (104,105). Se espera utilizar estas células inducidas a neuronas (iN) como modelos de enfermedades neurológicas. Lo anterior podría llegar a tener implicaciones importantes en el tratamiento de enfermedades y lesiones neurológicas y su rehabilitación.
Por último, estudios más ambiciosos han desarrollado la entrega in vivo de factores de transcripción en un modelo de ratón con daño hepático, como blanco terapéutico (95). Tratamientos médicos basados en la administración de factores de transcripción serían una verdadera revolución; no obstante, tendrían que superar todas las fases de estudios clínicos antes de poder llegar a ser considerados seguros y eficaces.
Aplicaciones clínicas de la epigenética
Así como los factores de transcripción se están utilizando en investigaciones básicas para entender la etiología de enfermedades, establecer herramientas diagnósticas y proponer opciones terapéuticas, la epigenética también tiene las mismas proyecciones. En humanos, se están descubriendo las causas genéticas de patologías de una manera sin precedentes (106). Ha venido en aumento el número de enfermedades que podrían ser agrupadas en una subclase de mutaciones que involucran cambios en el epigenoma o un incremento en las proteínas que regulan la estructura de la cromatina (106).
Se está dilucidando el paisaje epigenético del cáncer, enfermedades neurodegenerativas, dolor, enfermedades infecciosas, enfermedades metabólicas o mecanismos de envejecimiento (107,108,109,110,111,112). Las investigaciones que median los fenómenos epigenéticos se han enfocado en dos mecanismos moleculares: las modificaciones sobre las histonas y la metilación sobre el ADN (42). Las enfermedades pueden ser causadas por mutaciones de orden genético en los modificadores epigenéticos, que afecten la cromatina, a través de las proteínas que están implicadas en el proceso. Por otro lado, pueden tener un efecto en la secuencia del ADN que altera la configuración de la cromatina. Igualmente, las enfermedades también pueden ser causadas por cambios directos en las marcas epigenéticas, como la metilación sobre el ADN (106).
Para la generación del cáncer se han realizado múltiples esfuerzos con el fin de reconocer cuáles son los genes asociados a su etiología, lo que ha permitido identificar un número significativo de ellos que están directa o indirectamente asociados con la aparición de los cánceres más prevalentes en el ámbito clínico (113,114,115). Adicionalmente, dentro de los mecanismos involucrados en la aparición del cáncer se encuentran las modificaciones epigenéticas. Se han descrito procesos biológicos fundamentales, como la metilación del ADN: alteraciones en las enzimas implicadas en la metilación del ADN y patrones de metilación aberrantes sobre el ADN (116), modificaciones de las histonas, remodelación de la cromatina o regulación dirigida por especies de ARN (117).
Se ha descrito un aumento en el número de metilaciones en el ADN de las células cancerígenas responsables de la aparición de las neoplasias (118), específicamente dentro de las islas CpG, que en circunstancias normales se encuentran no metiladas. Estas metilaciones derivan su potencial carcinogénico del silenciamiento de genes de supresión tumoral (119). En la clínica, la hipermetilación en las islas CpG normalmente no metiladas, podría ser usado para el pronóstico de pacientes con esta patología (120). De igual manera, defectos en la DNMT se han visto asociados con la aparición y progresión de tumores —hepáticos, pancreáticos, pulmonares, uroteliales, gástricos, uterino-cervicales, de seno (121) y esofágicos (122)—. La sobreexpresión de las DNMT puede ser uno de los primeros eventos de la carcinogénesis. Igualmente, se ha propuesto que esta sobreexpresión puede causar mutaciones de novo en las islas CpG y contribuir al desarrollo tumoral (123). La reversibilidad de la sobreexpresión de las DNMT es de gran valor en este campo; de hecho, la FDA ha autorizado el uso de inhibidores de la expresión de las DNMT, como Vidaza® (azacitidina) (124), para el tratamiento de síndromes mielodisplásicos.
Tecnologías de última generación en la secuenciación del ADN, como la next-generation sequencing (NGS), han ayudado al entendimiento de enfermedades complejas como el cáncer (113). Esta nueva técnica ha revelado cambios epigenéticos que se pueden usar como marcadores tumorales, y que anteriormente eran inaccesibles debido a limitaciones metodológicas. Debido a que los fluidos de los pacientes con cáncer tienen por lo general un aumento en los niveles de ADN tumoral libre, estos marcadores epigenéticos pueden ser identificados en muestras de sangre periférica, saliva, orina, líquido peritoneal y líquido broncoalveolar, lo que podría hacer más oportuno el diagnóstico y el tratamiento de estos pacientes, mejora su pronóstico (114).
La NGS también se usa para detectar mutaciones heterocigotas en cáncer de seno y ovario. A diferencia del proyecto genoma humano, el cual inició en la década de los noventa y requirió 13 años de trabajo y billones de euros para su culminación, actualmente la NGS secuencia la totalidad del genoma en unos pocos días y por menos de mil dólares (114), lo que supone que puede ser utilizada como una herramienta diagnóstica.
Con respecto a epigenética de 31 genes y alteraciones de histonas en relación con enfermedades neurodegenerativas, una revisión epidemiológica sistemática del papel de modificaciones de las histonas y metilaciones del ADN en enfermedades neurodegenerativas publicada en el 2016 concluyó que el número de estudios asociados a cambios epigenéticos del genoma, como modificaciones de las histonas y metilaciones del ADN en Alzheimer y Parkinson, es importante. Se analizaron cerca de siete mil reportes, donde únicamente el 1 % incluyó estudios de casos-control, lo cual satisfizo su criterio de inclusión en el estudio. El trabajo recomendó una cohorte más grande para identificar cambios epigenéticos clínicamente significativos (125). Aunque existan muchos estudios, el rigor científico es importante para establecer su significancia en un ámbito clínico.
De igual manera, existen estudios que relacionan las modificaciones epigenéticas con dolor (112,126). La regulación de la expresión génica a través de la epigenética cumple un papel importante en la plasticidad neuronal y en la respuesta celular a los cambios ambientales (127). Existen reportes que asocian la epigenética con la transición del dolor agudo a crónico después de una lesión (112), y dolor y analgesia (126). Geranton (128) refiere la existencia de vías de señalización presentes en la percepción de dolor, que al mismo tiempo inducen cambios epigenéticos. También, como en el cáncer, existen terapias dirigidas a modificar estos cambios epigenéticos. En dolor, se abre la posibilidad de que diferentes medicamentos con base en el mismo principio tengan algún efecto como moduladores del dolor crónico (128). Todos estos pueden resultar en la aparición de esta enfermedad u otras entidades, como trastornos mentales o dolor neuropático (121).
Para enfermedades infecciosas virales, una revisión del 2018, Tarakhovsky y Prinjha (109), sugieren que la estrategia viral utiliza mimetismo de las histonas para utilizar un trastorno intrínseco en las proteínas y maximizar su impacto sobre las redes proteicas del hospedero, con el fin de minimizar el impacto de un genoma viral pequeño. Por otro lado, eventos epigenéticos han sido asociados a enfermedades autoinmunes como la enfermedad intestinal inflamatoria, donde la hipermetilación se asocia a la inflamación a largo plazo, específicamente en pacientes con colitis ulcerativa. De este modo, las modificaciones epigenéticas, como la metilación, se han relacionado con diferencias en el pronóstico clínico de la enfermedad, y estas podrían usarse como un marcador en el tamizaje para riesgo de cáncer colorrectal (118). También se han descrito modificadores epigenéticos como terapias inmunomoduladoras para tumores sólidos (129).
Actualmente se han implementado estas terapias o medicamentos dentro de ensayos clínicos (130). Más aún, hoy en día existen en el mercado medicamentos epigenéticos. Algunos usan mecanismos de inhibición selectiva de la metiltransferasa en dosis bajas (EC 2.1.1.43 Dacogen® (decitabina) y Vidaza® (azacitidina) para tratar el síndrome mielodisplásico; otros, inhibidores de la histona deacetilasa, Zolinza® (vorinostat) e Istodax® (romidepsina), para el tratamiento de linfoma de células T cutáneas. Adicionalmente, se han comenzado a integrar a ensayos clínicos controlados una segunda generación de estos medicamentos, en los que por medio de moléculas de menor tamaño interfieren con la actividad de enzimas epigenéticas y proteínas adaptativas, como la acetiltransferasa (EC 2.3.1.48) y la metiltransferasa de las histonas (130).
Con referencia a enfermedades metabólicas como la diabetes, Zhang y Pollin (131), en el 2018, informaron acerca de la variación epigenética en la patogénesis de la diabetes. De acuerdo con esta revisión, los estudios recientes ilustran el rol de cambios en los patrones de metilación, modificaciones de las histonas, impronta génica y ARNsmi a través de varios tipos de diabetes y sus complicaciones. En particular, se encontró que cambios en la metilación del antígeno leucocitario humano (HLA) preceden al desarrollo de diabetes tipo 1 (131).
Como se ha descrito, desregulaciones en procesos epigenéticos pueden estar asociados a cáncer. Así mismo, fallas en mecanismos epigenéticos de impronta genómica e inactivación del cromosoma X se han asociado a un subconjunto de síndromes congénitos (132). El clásico ejemplo de las enfermedades congénitas de impronta Angelman y Prader-Willi, las enfermedades congénitas asociadas a la inactivación del cromosoma X, enfermedades congénitas asociadas a defectos en el mecanismo epigenético o trastornos generalizados del desarrollo, potencialmente asociados con defectos en el mecanismo epigenético. Estas enfermedades y sus mecanismos patológicos asociados a procesos epigenéticos son descritos en la revisión de Kubota del 2008 (132).
El estudio de las alteraciones en los mecanismos de modificación epigenética en cáncer y otras enfermedades es un tema de interés general, debiéndose al papel que desempeñan en la regulación de la expresión génica y el mantenimiento de la integridad cromosomal. Hasta el momento se han descrito principalmente dos procesos moleculares: modificación de las histonas y metilación del ADN. Algunos estudios comienzan a dimensionar el papel que pueden ejercer los ARNsmi. Se espera que un mejor entendimiento de estos procesos permita el acceso a nuevas estrategias diagnósticas y terapéuticas.
Conclusión
Este milenio ha evidenciado un éxito incomparable en la identificación de las bases genéticas para cientos de enfermedades en humanos. Gracias al establecimiento de consorcios, se ha logrado correr la frontera del conocimiento de manera acelerada con respecto al entendimiento del genoma humano. Se han identificado regiones codificantes (2 %), así como regiones transcritas no codificantes y no transcritas. El porcentaje codificante no puede dar cuenta de toda la complejidad que permite a cada uno de los más de 200 tipos celulares conformar los tejidos en nuestro cuerpo e interpretar la información del genoma de manera diferente para realizar sus funciones vitales.
El genoma humano, constituido por aproximadamente 3000 millones de pares de bases, que codifican cerca de 19.000 genes, debe dar cuenta de la expresión de un conjunto definido de genes, que le otorgan a cada tipo celular su identidad y función. Los mecanismos que permiten dar respuesta a los retos a los cuales se enfrenta una célula, sean extrínsecos o intrínsecos, son dinámicos. La expresión génica diferencial es el proceso mediante el cual cada célula desarrolla su propio fenotipo y función por medio de la activación o inactivación de genes en diferentes momentos. Ello equipa al cuerpo humano con tejidos capaces del mantenimiento de la homeostasis.
A pesar de que cada célula cumple una función diferente, todas cuentan con el mismo genoma; lo que marca esta diferencia son los genes que serán expresados en cada célula. De manera que en células diferenciadas, los genes no utilizados no mutan ni se destruyen, pero mantienen su potencial de expresión génica (133). La identificación y entendimiento de las bases moleculares en procesos fisiológicos permiten dilucidar eventos patológicos en las células, que conforman los tejidos de un organismo. Por consiguiente, constituyen nuevas oportunidades de acercamiento de la investigación básica a la práctica clínica. Como se ha descrito, basados en principios de la regulación de la expresión génica, es posible establecer herramientas diagnósticas. De igual modo, el entendimiento de estos procesos se espera permita la identificación de nuevas dianas terapéuticas para una gran variedad de enfermedades.
Anteriormente se creía que la secuencia codificante del ADN nos definía; hoy se han descrito mecanismos que tienen funciones regulatorias sobre la expresión de un gen, comenzando por el estado de condensación y remodelación de la cromatina. La estructura de la cromatina define el estado en el cual la información del genoma está organizada en una célula. Esta organización influencia en gran parte la capacidad de activar o inactivar la expresión de genes.
Seguido de estos eventos epigenéticos, también se sabe que metilaciones directas sobre la secuencia del ADN, específicamente la citosina, pueden inactivar la expresión de un gen. Alteraciones de estas funciones se han descrito en diversas patologías. Avances en tecnologías de edición del genoma prometen dilucidar la contribución de la genética a las enfermedades. Algunos investigadores esperan, mediante técnicas moleculares, poder llegar a editar el genoma (134). Otros buscan establecer herramientas diagnósticas mediante la secuenciación del ARN (135). Por otro lado, el descubrimiento y desarrollo de medicamentos puede ser una aplicación estratégica de la expresión diferencial (136). Si el personal clínico entiende los principios que rigen la vida desde el punto de vista molecular, pueden valorar la importancia de las investigaciones básicas y sus hallazgos.
Un mejor entendimiento de los mecanismos de expresión diferencial establece los modelos que definen la enfermedad y pueden orientar estrategias terapéuticas. El horizonte de la medicina se encuentra aquí, pues con todos los avances que se han logrado se presentan nuevas alternativas diagnósticas y terapéuticas, y a futuro es posible dimensionar los beneficios que la investigación podría traer.