INTRODUCCIÓN
El lulo o naranjilla (Solanum quitoense Lam.), es un frutal de la familia Solanaceae de origen andino, cultivado principalmente en Ecuador y Colombia en sistemas de producción campesinos (<1,2 ha). Sus frutos se caracterizan por sus excelentes características organolépticas, un alto contenido nutricional como fuente importante de vitamina C, hierro, calcio y fósforo y gran potencial para su procesamiento industrial en la fabricación de jugos, helados, néctares y pulpas congeladas (Franco et al., 2002; Cardona et al., 2016). En Ecuador se cultivan cerca de 10.000 ha, principalmente con materiales híbridos derivados de cruces entre S. sessiliflorun X S. quitoense, mientras que en Colombia para el año 2015, se registraron unas 8.600 ha, principalmente con las variedades Lulo de Castilla var. septentrionale (con espinas) y var. quitoense (sin espinas), además del híbrido La Selva (S. hirtum X S. quitoense) (Cámara de Comercio de Bogotá, 2015; Agronet, 2018). Los departamentos de Huila (1.853 ha), Valle del Cauca (1.055 ha), Tolima (988 ha) y Antioquia (620 ha) son los principales productores de este frutal en Colombia, y aunque el rendimiento promedio nacional se estima en 9,63 t ha-1, existen grandes diferencias entre dichas regiones, pues en Antioquia se alcanzan 12,37 t ha-1, mientras que en Huila este valor es tan sólo de 6,69 t ha-1; lo que dista mucho de los rendimientos óptimos estimados para este frutal (17 t ha-1) (Franco et al, 2002).
Diferentes factores relacionados con la selección adecuada de las zonas de siembra, calidad de las semillas, prácticas agrícolas (ej. podas, fertilización) y condiciones de la cosecha inciden en los bajos rendimientos alcanzados en este cultivo en Colombia (Ardila et al., 2015; Cámara de Comercio de Bogotá, 2015; Cardona et al., 2016). Adicionalmente, el lulo se ve afectado por diferentes plagas y enfermedades, siendo el gusano perforador del fruto (Neoleucinodes elegantalis) y el barrenador del tallo (Faustinus sp.) dos de los insectos más limitantes; mientras que la gota (Phytophthora infestans), la antracnosis (Colletotrichum gloeosporioides) y la fusariosis (Fusarium oxysporum) son las enfermedades micóticas más importantes; así como lo son la marchitez bacterial (Ralstonia so-lanacearum), los nemátodos agalladores (Meloidogyne spp.) y el virus de la hoja pequeña, que inicialmente se registró en Antioquia en 1986, y que en la actualidad se considera endémico para todas las zonas productoras de Colombia (Tamayo et al., 2003; Cámara de Comercio de Bogotá, 2015). Desafortunadamente, la etiología de esta enfermedad viral no se ha confirmado aún, aunque estudios preliminares sugieren su afinidad serológica con el Potato leafroll virus (PLRV), un polerovirus que afecta drásticamente cultivos de papa en todo el mundo (Tamayo et al., 2003; Marín y Gutiérrez, 2016). Además de esta enfermedad, en cultivos de lulo de Colombia se ha registrado otra de posible origen viral denominada como "Amarillamiento de la hoja u hoja en forma de abanico" (Zuluaga, 1996); aunque nuevamente, su etiología específica se desconoce, siendo inicialmente descartado por Vaca et al. (1999) que fuera causada por un potyvirus.
En el mundo sólo se ha confirmado plenamente la infección de una especie viral en plantas de lulo bajo condiciones naturales, registro que se realizó recientemente en la provincia de Pastaza en Ecuador, sobre plantas con síntomas de mosaicos y manchas cloróticas del híbrido "Puyo", y que corresponde a un nuevo tymovirus cuyo nombre tentativo es Naranjilla chlorotic mosaic virus (NarCMV) (Green et al., 2018); mientras que utilizando inoculaciones bajo condiciones de invernadero, se identificaron reacciones de hipersensibilidad de S. quitoense ante el Tobacco necrosis virus (TNV) y de lesiones locales cloróticas/ necróticas y mosaicos sistémicos al Alfalfa mosaic virus (AMV), Potato aucuba mosaic virus (PAMV), Potato virus X (PVX), Potato virus Y (PVY) y Tobacco rattle virus (TRV) (Horvath, 1987); estos virus también se reportan afectando diferentes plantas solanáceas (King et al., 2012; Marín y Gutiérrez, 2016).
Recientemente, en un estudio de reconocimiento de enfermedades virales en cultivos de plantas solanáceas del Oriente de Antioquia (Colombia), se encontraron plantas con una sintomatología mixta asociada a virosis, que incluía mosaicos, moteados, amarillamientos intervenales y deformación de brotes. Ante las evidentes deficiencias de información que actualmente se tiene sobre los agentes causales de virosis en este cultivo, se decidió realizar el presente estudio, tendiente a identificar las especies de virus presentes en dichos tejidos sintomáticos, utilizando la metodología molecular de secuenciación de nueva generación (NGS) y la posterior confirmación de los virus detectados mediante pruebas de RT-PCR con cebadores específicos.
MATERIALES Y MÉTODOS
Material vegetal
Durante los meses de Abril a Junio de 2017, se realizaron recorridos de reconocimiento de enfermedades virales en cultivos de lulo de los municipios de Marinilla (2 lotes), Sonsón (2 lotes) y San Vicente Ferrer (2 lotes), ubicados en el Oriente de Antioquia. En los dos lotes de Marinilla y San Vicente Ferrer y en un lote de Sonsón, se evidenciaron plantas con síntomas posiblemente asociados a infecciones virales (ej. mosaicos, moteados, amarillamientos intervenales y deformación de brotes) (Fig. 1), seleccionándose dos plantas de cada lote para la extracción de ácidos nucleicos en el laboratorio.
Secuenciación de nueva generación (NGS)
A cada una de las 10 muestras sintomáticas obtenidas, se les realizó la extracción del RNA total utilizando el método del Trizol (Ambion, Thermo-Fisher Scientific, Waltham, MA, USA), previa maceración con nitrógeno líquido, y se evaluó la concentración de RNA y su pureza con lecturas a 260 y 280 nm en Nanodrop 2000C (Thermo-Fisher Scientific, Waltham, MA, USA). Posteriormente, se realizó una mezcla (bulk) de 10 μL de RNA de cada muestra, para ser utilizada como base del NGS, utilizando el sistema llumina HiSeq-2500 (Macrogen, Seúl, Corea del Sur), previa degradación del RNA ribosomal con el tratamiento Ribo-Zero Plant (Illumina, San Diego, CA, USA) y la construcción de la librería de cDNA con el kit TruSeq RNA Sample Preparation Kit (Illumina, San Diego, CA, USA). Una vez obtenidas las secuencias, se procedió a la remoción de los adaptadores y de las bases de baja calidad (Phred <30) utilizando el software Trimommatic (Bolger et al., 2014) y al ensamblaje de contigs mediante Trinity (Grabherr et al., 2011). Los contigs asociados a genomas virales fueron identificados mediante BLASTN y BLASTX (Gish y States, 1993). Las secuencias de los genomas virales fueron confirmadas por mapeo con respecto a genomas de referencia utilizando el programa BWA (Li y Durbin, 2009) y los marcos abiertos de lectura (ORF) fueron identificados por comparación con GenBank usando BLASTX (Gish y States, 1993). Los análisis filogenéticos con las secuencias obtenidas de los ge-nomas virales, se realizaron con el programa MEGA7 (Kumar et al., 2016), utilizando el método de máxima verosimilitud con el modelo general de tiempo reversible y 1000 réplicas de bootstrap (Nei y Kumar, 2000).
Confirmación viral por RT-PCR
Una vez identificados mediante NGS, los virus presentes en las muestras sintomáticas de lulo, se procedió a su confirmación en las 10 plantas individuales mediante pruebas de RT-PCR con cebadores específicos diseñados en este trabajo a partir de las secuencias de los genomas completos de CMV (CMV1RP_F: CGTCCACTGACATGATTGTTACC y CMV1RP_R: CTCCACGACTGACCATTTTAGC) y ANSV (Tospo_S_F: CAGAATCAGGCTGCATTTAATTTCC y Tospo_S_R: CAACGCTTCCTTTAGCATTAGG) con ayuda del software Primer-3 (Untergasser et al., 2012); así como con los cebadores reportados por Álvarez (2016) para PYVV (PYVV_F_CP: TCAGG-TTAGAGCAGACAGAGG y PYVV_R_CP: AGTT-GCTGCATTCTTGAACAGG). Para esto, se realizó la síntesis del DNA complementario (cDNA) de primera cadena utilizando 100 pmol de cada cebador reverso, 200 U de la enzima RevertAid Reverse Transcriptase (Thermo-Fisher Scientific, Waltham, MA, USA), 1X de buffer RT, 0,5 mM de los cuatro dNTPs, 20 U de inhibidor de RNAsas (RiboLock, Thermo-Fisher Scientific, Waltham, MA, USA) y de 100 a 500 ng de RNA. Las reacciones se incubaron a 42°C por 60 min y finalmente la enzima se desactivó a 70°C por 10 min. De esta forma, 50-100 ng del cDNA fueron utilizados para las reacciones de PCR en un volumen total de 25 μL, que contenían 1 U de la enzima Taq DNA polymerase (Thermo-Fisher Scientific, Waltham, MA, USA), 1X de buffer, 1,8 mM de MgCl2, 0,2 mM de los cuatro dNTPs, 0,2 /μM de los cebadores específicos para cada virus, y 1 μL de albumina de suero bovino (10 mg mL-1). La amplificación se realizó en un termociclador T3 (Biometra, Goettingen, Alemania) y consistió de una desnaturalización inicial a 95°C por 3 min, seguido por 40 ciclos de: 94°C (30 s), 52°C (1 min) y 72°C (1 min); y una extensión final por 5 min a 72°C. La presencia de amplicones se determinó por electroforesis en gel de agarosa al 1,8% y tinción con GelRed 1X (Biotium, Fremont, CA, USA), utilizando un visualizador de luz ultravioleta Bio Doc Analyze transilluminator (Biometra, Goettingen, Alemania). Finalmente, al menos tres de los amplicones del tamaño esperado fueron purificados del gel utilizando el kit GeneJET Gel Extraction (Thermo-Fisher Scientific, Waltham, MA, USA) y su naturaleza viral fue confirmada por secuenciación Sanger en ambas direcciones en un equipo ABI Prism 3730xl sequencer (PE Applied Biosystems, Foster City, CA, USA) de la compañía Macrogen (Seúl, Corea del Sur).
RESULTADOS Y DISCUSIÓN
Secuenciación de nueva generación
En el NGS se obtuvieron 10.777.822 reads de 101 nt, siendo posible identificar mediante análisis bioinfor-máticos un 58,54% de los reads (6.309.549) como correspondientes a diferentes tipos de RNA vegetal (ej. mensajero, ribosomal, no codificante, etc.) de origen nuclear (32,24%) y citoplasmático (3,14% mitocondrial y 23,16% cloroplástico), especialmente derivados de secuencias de plantas de la familia Solanaceae.
Adicionalmente, se encontró una baja cantidad de transcritos asociados a insectos (ej. áfidos, moscas blancas, trips), bacterias (ej. Niastella spp., Methylo-bacterium spp.) y hongos (ej. Colletotrichum spp., Bipo-laris spp.). De estos reads, 4.356.916, equivalentes al 40,4% del total obtenido, correspondieron a secuencias de origen viral, lo que confirma que la sintomatología observada en los cultivos de lulo efectivamente estaba asociada a muy altos niveles de infección viral. Al realizar los ensamblajes en contigs, se lograron obtener los genomas completos de los virus de RNA: Cucumber mosaic virus (CMV), Potato yellow vein virus (PYVV) y Alstroemeria necrotic streak virus (ANSV).
Cucumber mosaic virus (Bromoviridae)
Los reads derivados del genoma del CMV fueron mayoritarios en el transcriptoma bajo análisis, al representar el 99,6% (4.340.953) de los reads virales, siendo posible ensamblar su genoma completo de 8.523 nt, distribuidos en tres segmentos de RNA de cadena sencilla positiva y con una profundidad promedio de 73.169x, 93.172x y 155.105x, para el RNA 1, 2 y 3, respectivamente (Fig. 2A). El RNA 1 presentó un tamaño de 3.342 nt y un ORF que codifica para la proteína 1a de 995 aminoácidos (aa) implicada en la replicación del virus, pues presenta los dominios funcionales metil-transferasa en el extremo N-terminal y helicasa en el C-terminal. Por su parte, el RNA 2 de 3.024 nt, contiene dos ORF (2a y 2b), que codifican para la replicasa viral (2a), una RNA polimerasa dependiente de RNA (RDRP) y para una proteína supresora de silenciamiento de genes (2b) que se expresa a partir de la estrategia de RNA subgenómico y es referido en este virus como RNA 4A (Zitter y Murphy, 2009). El RNA 3 presentó una extensión de 2.157 nt y tiene dos ORF: el 3a que codifica para una proteína de movimiento (MP) de 279 aa, esencial para el desplazamiento del virus de célula a célula y el 3b codifica para la proteína de la cápside (CP) de 218 aa, que se expresa a partir de un segundo RNA subgenómico, denominado como RNA 4. Esta proteína CP, además de su función protectora del genoma viral, presenta los determinantes de transmisión no persistente del CMV por parte de sus insectos vectores, que corresponden a cerca de 80 especies de áfidos, incluyendo Myzus per-sicae y Aphis gossypii (Zitter y Murphy, 2009; Nouri et al., 2014). Las secuencias completas de este genoma fueron depositadas en GenBank con los códigos de accesión: MG696854, MG696855 y MG696856.
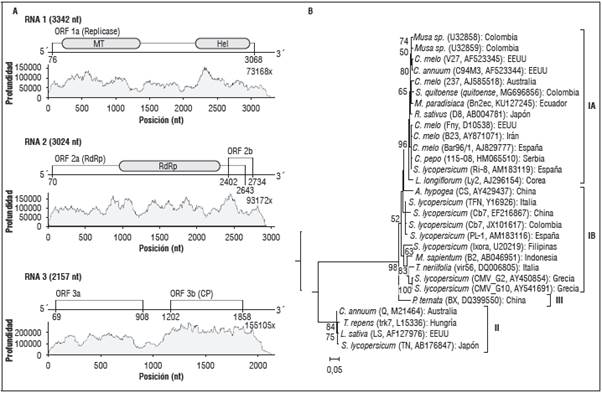
Figura 2 A. Diagrama del genoma del Cucumber mosaic virus (CMV) indicando las posiciones de los ORF identificados y los niveles de profundidad para cada segmento de RNA; B. Árbol filogenético basado en secuencias de la cápside para el aislamiento de CMV de lulo y de otros hospedantes. Los números sobre las ramas indican los valores de bootstrap y las denominaciones en la derecha corresponden a los subgrupos hasta ahora identificados en este virus.
El CMV, especie tipo del género Cucumovirus (Bromoviridae) (King et al., 2012), es posiblemente uno de los virus de plantas con mayor distribución geográfica y amplio rango de hospedantes, pues se ha reportado infectando cerca de 1.200 especies de plantas de 100 familias botánicas diferentes, incluyendo un alto número de solanáceas (Nouri et al., 2014; Thompson et al., 2015), lo que es compatible con el presente registro sobre S. quitoense, que por lo demás se constituye en el primer reporte de este virus sobre este hospedante. El análisis filogenético realizado con base en la secuencia de CP, ubicó al aislamiento obtenido en lulo, en el clado que representa el subgrupo I de dicha especie, y específicamente en el linaje IA (Fig. 2B). En este clado, también se ubicaron aislamientos obtenidos en diferentes países a partir de plantas cucurbitáceas, musáceas y solanáceas, entre otros hospedantes, siendo el aislamiento Bn2ec (KU127245) procedente de banano de Ecuador, el que compartió mayores niveles de identidad (98%) con el CMV de lulo; mientras que dichos niveles fueron del 90-94%, 81% y del 90% con respecto a los aislamientos de los subgrupos IB, II y III, respectivamente. La especie CMV inicialmente se dividió en los subgrupos I y II, con base en criterios biológicos, serológicos, fisicoquímicos y de porcentajes de hibridización entre genomas (Jacquemond, 2012). Posteriormente, el subgrupo I se separó en dos linajes definidos con base en secuencias del gen CP y de los extremos 5' del RNA 3 (Roossinck et al., 1999) y más recientemente, se encontró un tercer subgrupo (III) a partir de análisis de genomas completos de este virus (Liu et al., 2009; Jacquemond, 2012). Los subgrupos IA y II tienen una distribución global y amplio rango de hospedantes, mientras que el IIB se ha detectado principalmente en Asia; además se han encontrado diferentes cepas de CMV que no se agrupan en ninguno de los tres subgrupos y que representan variantes recombinantes de este virus (Nouri et al., 2014; Thompson et al., 2015). En este sentido, mediante pruebas de RT-PCR convencional y secuenciación Sanger, en este trabajo se confirmó de manera independiente la identidad del linaje IA de otros dos aislamientos de CMV procedentes de plantas individuales de lulo con síntomas de mosaicos y deformación de brotes de un lote de San Vicente Ferrer y otro de Marinilla, obteniéndose un amplicon de 920 pb del RNA 1 con los cebadores diseñados a partir del genoma obtenido mediante NGS. En este caso, ambos amplicones compartieron niveles de identidad superiores al 98% con respecto a secuencias depositadas en GenBank para el linaje IA de este virus (ej. HE793683, AM183117) (Tepfer et al, 2016). Sin embargo, dados los muy altos niveles de diversidad que presenta CMV como resultado no sólo de las altas tasas de mutación propias de los virus de RNA, sino también de la frecuencia de eventos de recombinación homóloga y heteróloga reportados para esta especie viral, y de los re-arreglos genómicos no sólo entre segmentos de cepas de diferentes subgrupos de CMV, sino también entre especies de cucumovirus (ej. CMV y Peanut stunt virus -PSV) (Roossinck et al., 1999; Jacquemond, 2012; Thompson et al., 2015), es necesario emprender estudios de variabilidad genética de aislamientos de CMV obtenidos en cultivos de lulo de diferentes regiones del país. Dicha información, será fundamental para diseñar las estrategias de manejo más apropiadas para evitar la dispersión del virus entre regiones del país, identificar reservorios naturales (ej. arvenses), vectores específicos y para el diseño de herramientas de diagnóstico que apoyen futuros programas de certificación de semilla y de mejoramiento genético de este frutal andino.
Alstroemeria necrotic streak virus (Bunyaviridae)
Este virus fue detectado en el transcriptoma de lulo a partir de 6.582 reads, que una vez ensamblados permitieron obtener la secuencia completa (16.708 nt) de los tres segmentos genómicos (RNA: S, M y L) con profundidades de ensamblaje de 55x, 67x y 159x, respectivamente (Fig. 3A). El RNA pequeño (S) tiene una extensión de 3.113 nt y dos ORF, el que codifica para la proteína no estructural (NSs) de 477 aa en el sentido del virión (vs) y que actúa como supresor de RNA de interferencia (RNAi); y el que codifica para la proteína de la nucleocápside (N) de 259 aa en el sentido complementario al virión (vc) (Domínguez y Crespo, 2008; King et al., 2012). El RNA mediano (M) tiene un tamaño de 4.839 nt e igualmente dos ORF ambisentido: el que codifica para la proteína de movimiento (NSm) de 304 aa, en el sentido del virión, y el GnGC (vc) que codifica para el precursor de las glicoproteínas G1 y G2 asociadas a la envoltura externa del virión (Domínguez y Crespo, 2008; King et al., 2012). Finalmente, el RNA grande (L) de polaridad negativa tiene 8.756 nt y un sólo ORF en el sentido vc que codifica para la RNA polimerasa viral de 2.875 a.a. La secuencia del ANSV se depositó en GenBank con los números de accesión MG696853, MG696852 y MG696851, y corresponde al primer registro en el mundo del genoma completo de este virus.
El ANSV (Tospovirus, Bunyaviridae) fue reportado inicialmente en Colombia infectando plantas de Alstroemeria sp. (Hassani-Mehraban et al., 2010), obteniéndose la secuencia de un segmento de 1.240 nt del RNA S, que incluía el ORF N completo y cerca de 152 nt de la región UTR 5' y 311 nt de la región intergénica; los niveles de identidad encontrados en ese trabajo para la proteína N, correspondieron a niveles del 82, 81 y 80% con respecto a las especies Tomato chlorotic spot virus (TCSV), Groundnut ringspot virus (GRSV) y Tomato spotted wilt virus (TSWV), respectivamente, valores inferiores al 90% indicado como base para diferenciar especies dentro de este género (King et al., 2012). Análisis filogenéticos recientes basados en secuencias del ORF N y de la región 5'-UTR de 28 especies de tospovirus, ubicaron al ANSV en el clado Americano 1 (AM-C1) en conjunto con otras ocho especies que incluyen el TSWV, TCSV y Pepper necro-tic spot virus (PNSV) (Hassani-Mehraban et al., 2016), previamente reportadas afectando plantas solanáceas en diferentes países suramericanos (Williams et al., 2001; Torres et al., 2012; Ebratt et al., 2013). Este mismo resultado fue obtenido cuando se realizó el análisis filogenético con la secuencia del ORF N derivada del análisis NGS, pues el aislamiento de ANSV de lulo se ubicó en el clado AM-C1 y compartió un 99% de identidad con la secuencia ACZ18222, inicialmente reportada por Hassani-Mehraban et al. (2010) en la descripción de esta especie (Fig. 3B). Adicionalmente, Olaya et al. (2017) reportaron la infección natural del ANSV en plantas de pimentón y tomate con síntomas de lesiones y anillos necróticos en hojas y tallos, obtenidas en los departamentos de Risaralda y Antioquia, y específicamente para este último, en el municipio de Marinilla, lo que coincide con el presente reporte de ANSV en lulo, pues algunas de las muestras fueron también obtenidas en dicho municipio. De gran interés resultará en el futuro evaluar la patogenicidad cruzada entre aislamientos de ANSV procedentes de estas solanáceas, así como identificar sus vectores específicos, que para los tospovirus, generalmente corresponden a trips (Hassani-Mehraban et al., 2010). La confirmación independiente de la infección de ANSV en plantas de lulo del Oriente Antioqueño, se realizó mediante RT-PCR con cebadores específicos diseñados con base en el genoma del ANSV obtenido en este estudio, y fue positiva (613 pb) para cuatro de las muestras (Tab. 1). Las secuencias obtenidas para dichas muestras presentaron un 99% de identidad con respecto a dos secuencias del ORF N de aislamientos de ANSV de Colombia y depositadas en GenBank con los números de accesión KX833218 y GQ478668. La detección de ANSV en este trabajo, reafirma el llamado de atención planteado por Olaya et al. (2017) sobre los riesgos fitosanitarios que puede representar el ANSV en Colombia y otros países andinos sobre cultivos de solanáceas de importancia económica y por esto, de la necesidad de evaluar su distribución geográfica y sus posibles mecanismos de transmisión en campo, evaluaciones en las que se podrían utilizar los cebadores específicos diseñados en este trabajo.
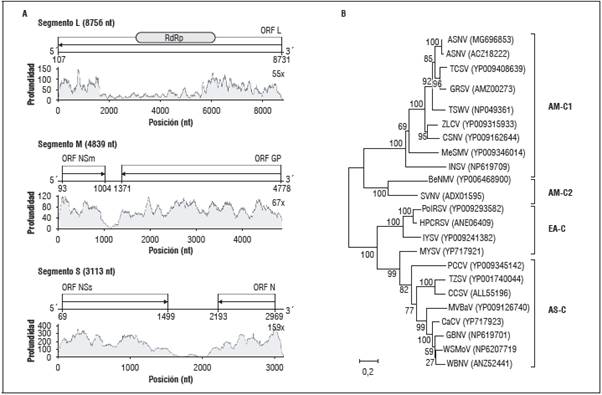
Figura 3 A. Diagrama del genoma del Alstroemeria necrotic streak virus (ANSV) indicando las posiciones de los ORF identificados y los niveles de profundidad para cada segmento de RNA; B. Árbol filogenético basado en secuencias de la cápside para el aislamiento de ANSV de lulo y de otros hospedantes. Los números sobre las ramas indican los valores de bootstrap y las denominaciones en la derecha corresponden a los subgrupos hasta ahora identificados en este virus.
Tabla 1 Detección en plantas de lulo de tres virus de RNA mediante RT-PCR con cebadores específicos.

+/-: Resultado de la amplificación por RT-PCR de un fragmento de 495, 920 y 613 pb para PYVV, CMV y ANSV, respectivamente. Los símbolos +/- representan los resultados de las evaluaciones para dos plantas por cada lote.
PYVV (Closteroviridae)
De los reads virales identificados en el transcriptoma de lulo, 10.480 estuvieron asociados a los tres segmentos genómicos del PYVV (Crinivirus, Closteroviridae), siendo posible ensamblar sus secuencias completas con tamaños de 7.829 nt (RNA 1), 5.296 nt (RNA 2) y 3.875 nt (RNA 3) y niveles de profundidad de 42x, 174x y 214x, respectivamente (Fig. 4A). En el RNA 1 se identificaron dos ORF, el ORF1 (a y b) que codifica para las proteínas de replicación del virus que incluyen los dominios de L-proteasa, metiltransferasa, helicasa (1a) y la RDRP (1b); mientras que el ORF2 lo hace para una pequeña proteína hidrofóbica (p7) de 61 aa (Livieratos et al., 2004; Álvarez et al., 2017). En el RNA 2 se identificaron los cinco ORF reportados para este segmento en los closterovirus y que incluyen la proteína de choque térmico 70 (HSP70h), la proteína pequeña p7 (de función desconocida hasta ahora), la proteína estructural p60, la proteína p10 (de función desconocida) y la cápside viral (CP); el número estimado de aa para dichas proteínas en el aislamiento de PYVV de lulo, correspondió a 554, 55, 521, 84 y 255 aa, respectivamente (Livieratos et al., 2004; Álvarez et al., 2017). Por su parte, el RNA 3 presentó los ORF que codifican para la proteína pequeña p4 (35 aa), la cápside menor (CPm, 674 aa) y la proteína P26 (230 aa) de función desconocida (Livieratos et al., 2004; Álvarez et al., 2017). Los números de accesión del genoma de este aislamiento de PYVV corresponden a MG696857, MG696858 y MG696859. Al comparar las secuencias completas de los tres segmentos de este virus con las depositadas en GenBank, se encontró que el RNA 1 compartía 96% de identidad con respecto a cepas de PYVV obtenidas en papa criolla (KR998193) y tomate (KX573901) en el Oriente de Antioquia y reportadas por Álvarez et al. (2017) y Muñoz-Baena et al. (2017), respectivamente; mientras que los RNA 2 y 3 presentaron 99% de identidad con la cepa LU1 de papa criolla de La Unión (Antioquia) (KR998194, KR998195) y con la cepa Chota de Cajamarca (Perú), originalmente utilizada por Livieratos et al. (2004) en la descripción del genoma de esta especie viral (AJ557129, AJ508757). El análisis filogenético realizado a partir de la secuencia de CP, generó un dendrograma con dos clados, uno mayoritario en donde se ubicó el aislamiento de PYVV de lulo en conjunto con otros 22 aislamientos de Colombia y Perú obtenidos principalmente en papa (S. tuberosum y S. phureja); mientras que el otro clado sólo incluyó aislamientos de PYVV en tomate, los que recientemente fueron identificados por Muñoz-Baena et al. (2017) como una nueva variante de PYVV en Colombia (Fig. 4B). La confirmación independiente por RT-PCR con los cebadores diseñados por Álvarez et al. (2017), resultó positiva para nueve de las diez muestras de plantas individuales (Tab. 1). Al secuenciar tres de los amplicones de 495 pb, se encontraron niveles de identidad del 99% con respecto a diferentes aislamientos de PYVV obtenidos en papa en el Oriente Antioqueño (KR998194, KR998190) y en Cundinamarca (AJ586117, KC257443). En estas zonas productoras de papa de Colombia, el PYVV es uno de los virus que presenta mayores niveles de incidencia en cultivos de papa y papa criolla (Guzmán et al., 2012), e incluso se ha estimado que en dichos hospedantes puede ocasionar pérdidas en rendimiento del 33-48% y 25-50%, respectivamente (Salazar et al., 2000; Guzmán et al., 2012). Este virus induce amarillamientos de venas secundarias en la lámina foliar y es reconocido como restringido al floema y de transmisión natural por moscas blancas de la especie Trialeurodes vaporariorum (Guzmán et al., 2012; Álvarez et al., 2017). Además de la papa, su rango de hospedantes incluye el tomate y diversas arvenses como Catharanthus roseus, Malva sp., Polygonum sp., Tagetes sp. y Rumex sp. (Salazar et al., 2000; Guzmán y Rodríguez, 2010). En el presente estudio, se realiza este nuevo registro en lulo, pues, aunque Salazar et al. (2000) habían evaluado su infección en este hospedante en Rionegro (Antioquia), su detección resultó negativa empleando la metodología NASH (Nucleic Acid Spot Hybridization). La secuencia completa del genoma de PYVV en lulo, se constituye en el cuarto reporte de este tipo para este virus en el mundo y amplía la base de información para evaluar niveles de variación genética intraespecífica, búsqueda de determinantes de transmisión y la función de algunas de sus proteínas (ej. p4, p26, p7, p10).
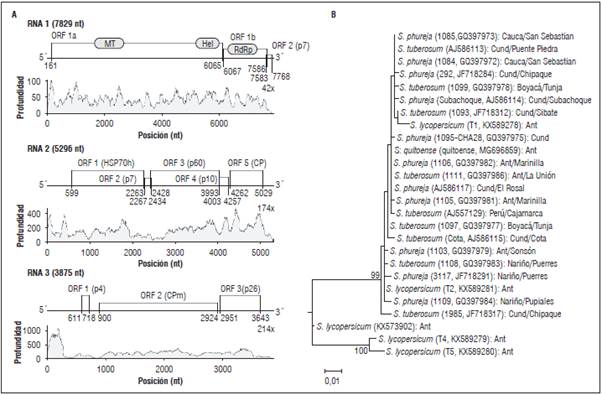
Figura 4 A. Diagrama del genoma del Potato yellow vein virus (PYVV) indicando las posiciones de los ORF identificados y los niveles de profundidad para cada segmento de RNA; B. Árbol filogenético basado en secuencias de la cápside para el aislamiento de PYVV de lulo y de otros hospedantes. Los números sobre las ramas indican los valores de bootstrap.
En este estudio, utilizando la metodología molecular NGS y confirmación independiente por RT-PCR, se identificaron tres virus multipartitas de RNA (CMV, ANSV, PYVV) infectando plantas sintomáticas de lulo obtenidas en lotes de cultivo del Oriente Antioqueño. Estos hallazgos se unen al reciente reporte de Green et al. (2018) en Ecuador, en donde se detectó una nueva especie de tymovirus nombrada tentativamente como Naranjilla chlorotic mosaic virus (NarCMV). En Colombia, aunque se han descrito al menos dos enfermedades de posible origen viral en cultivos de lulo de la región andina del país (departamentos Cundinamarca, Boyacá y Antioquia), denominadas como "virus de la hoja pequeña" y "amarillamiento de la hoja" (Zuluaga, 1996; Vaca et al., 1999); no se ha confirmado plenamente la identidad de sus agentes causales, aunque se ha sugerido que en el primer caso podría ser el PLRV (Tamayo et al., 2003) y en el segundo, aunque se detectaron secuencias de potyvirus, no se pudo asociar su infección con la sintomatología propia de dicha enfermedad (Vaca et al., 1999). La ocurrencia de infecciones virales como las detectadas en este trabajo asociadas a plantas con diversos de síntomas que incluyen algunos de los descritos para las dos enfermedades indicadas anteriormente (ej. amarillamientos intervenales, deformación de hojas y mosaicos), ya se ha reportado en Colombia para otros frutales solanáceos como tomate de árbol (Jaramillo et al., 2011) y uchuva (Gutiérrez et al., 2015; Álvarez, 2016) y son muestra de que el conocimiento que tenemos del viroma de estos hospedantes es aún incipiente. El hallazgo de estas tres especies de virus afectando cultivos de lulo, conduce al planteamiento de muchos interrogantes sobre la biología, patogenicidad, mecanismos de transmisión, métodos de detección específica y estrategias de manejo de estos problemas fitopatológicos; aunque inicialmente resultará fundamental evaluar la sintomatología específica que cada virus induce en plantas de lulo de las diferentes variedades cultivadas en Colombia, así como su posible patogenicidad cruzada entre solanáceas y otros cultivos frecuentemente intercalados o escalonados con lulo en los andes Colombianos (ej. frijol, arveja). Por último, será fundamental evaluar su posible transmisión por semilla sexual en lulo de Castilla, y por estacas, chupones o injertos para los materiales híbridos que se cultivan en el país.
CONCLUSIONES
Utilizando la metodología molecular de secuenciación de nueva generación del transcriptoma de plantas de lulo con síntomas asociados a enfermedades virales, se identificó la infección de tres especies de virus de RNA: CMV ANSV y PYVV con genomas multipartitas; siendo el CMV, el que presentó mayor porcentaje de reads (99,6% de los reads virales) en el set de datos obtenido.
En los tres virus detectados fue posible el ensamblaje de sus genomas completos, los que presentaron extensiones de 8.523 nt, 16.708 nt y 17.000 nt para CMV, ANSV y PYVV, respectivamente. El reporte de la secuencia del genoma completo de ANSV es el primero para este tospovirus, mientras que para CMV y PYVV corresponde a los primeros registros de este tipo para aislamientos que infectan lulo.
La infección de los tres virus en muestras sintomáticas foliares de plantas de lulo individuales fue confirmada mediante pruebas de RT-PCR, para lo que fue necesario diseñar cebadores específicos con base en los genomas de CMV y ANSV; mientras que el PYVV se detectó con cebadores previamente diseñados para aislamientos de papa. Estos cebadores podrán ser empleados en evaluaciones futuras de niveles de incidencia viral en lotes de cultivo, para apoyar programas de certificación de semilla de lulo o para evaluar los insectos vectores específicos de dichos virus en las diferentes regiones de este cultivo en Colombia y otros países.