INTRODUCCIÓN
La alpaca (Vicugna pacos) es un camélido que habita las zonas altoandinas de Sudamérica, principalmente los andes del Perú, y es por ello una importante fuente de ingreso económico para los habitantes de esta región. Motivo por el cual es sumamente importante establecer programas de mejoramiento genético para esta especie.
Uno de los principales pilares del mejoramiento genético es la selección de reproductores, la cual se debería realizar tomando en cuenta parámetros productivos y reproductivos; lamentablemente en alpacas, no se evalúan las muestras seminales de forma rutinaria 1. Esto se podría deber a la dificultad de obtención y manejo del semen de esta especie 2, y al hecho de que la evaluación mediante espermiograma básico no permite predecir la fertilidad del reproductor 3. Con lo cual observamos que en campo se realiza una deficiente selección de reproductores, y por ende un inadecuado mejoramiento genético. Una solución viable a esta problemática es el empleo de marcadores moleculares 4 como parte del plan de selección y evaluación reproductiva en alpacas, tal y como se viene empleando en otras especies productivas.
Existen diversos marcadores moleculares de fertilidad estudiados en especies de producción, siendo uno de ellos la familia de las protaminas (PRM's), la cual está formada por 3 proteínas. Estas proteínas son las responsables del hiper empaquetamiento del ADN espermático (PRM 1 y 2) 5,6 y participan en la motilidad espermática (PRM 3). Por ello, han sido catalogadas como marcadores moleculares de gran importancia en la fertilidad de bovinos 7.
De las 3 proteínas integrantes de la familia, la menos estudiada es la PRM3, cuya expresión ha sido cuantificada en citoplasma espermático y se ha relacionado como marcador de motilidad 7,8; sin embargo, estudios recientes en bovinos han determinado que es un marcador de fertilidad post-criopreservación 9,10,11, lo cual la hace una opción interesante de estudio, sobre todo en alpaca, que es una especie en la cual aún no se ha terminado de secuenciar el genoma, ni la expresión del mismo.
MATERIALES Y MÉTODOS
Identificación de la muestra. Primero se identificó el gen PRM3 en el genoma de la alpaca (Código de ID: 102543875) y la secuencia del ARNm (Código de acceso: XM_006204352.3) dentro de la base de datos del National Center for Biotechnology Information (NCBI) (https://www.ncbi.nlm.nih.gov/). Luego, se realizó el alineamiento de las secuencias empleando la herramienta bioinformática blast nucleotides (https://blast.ncbi.nlm.nih.gov/Blast.cgi7PAGE TYPE=BlastSearch), seleccionando el programa "megablast" (secuencias altamente similares).
Para desarrollar los primers, se empleó como base las secuencias conservadas del alineamiento previo y el programa Primer-BLAST (https://www.ncbi.nlm.nih.gov/tools/primer-blast/).
Evaluación in vitro. Para la evaluación in vitro de los primers, se trabajó un total de 51 muestras de tejido testicular obtenidas del camal municipal de Huancavelica. Los criterios de inclusión para las muestras fueron machos en un rango de edad de 1 a 4 años según dientes, con descenso de ambos testículos, sin patologías visibles y con tamaño testicular mayor a 3 cm 12. Las extracciones de ADN se realizaron en el Laboratorio de Biotecnología Reproductiva y Celular de la Universidad Científica del Sur, empleando el kit InnuPREP DNA Mini Kit (cod. 845-KS-1040250). Para la PCR, se usó un termociclador Thermal Cycler 0.2ml x 64 PCR-300 (mcr), aplicando el siguiente protocolo: 1 ciclo de desnaturalización inicial de 952C por dos minutos, 40 ciclos de 952C de 40 segundos (desnaturalización), 582C de 40 segundos (anillamiento), 722C de 40 segundos (elongación) y 1 ciclo de elongación final 72°C de 10 minutos. El producto obtenido mediante la PCR se corrió en gel de agarosa al 2% y se observaron los fragmentos amplificados en un fotodocumentador (Enduro™ GDS).
RESULTADOS
Identificación. Se identificó la secuencia del gen PRM 3, dentro del cromosoma 18, perteneciente del fichero VicPac3.1, localizado en una zona aproximada (NW_021964194.1). Según la información del NCBI, cuenta con un solo exón localizado entre el nucleótido 9.457.123 y 9.457.520 pb.
Desarrollo y evaluación de primers. Respecto al desarrollo de los primers, aplicando los criterios de diseño mencionados y las secuencias conservadas en el software Primer-Blast, se obtuvieron un total de 10 pares como se muestran en la tabla 1. Luego, empleando un muestreo no probabilístico aleatorio, se seleccionaron 2 pares: el número 1 y 6 de la tabla 1, los que fueron renombrados como par PRM3-P31 y PRM3-P32 respectivamente.
Tabla 1 Secuencias y características fisicoquímicas de los primer diseñados mediante el programa Primer-BLAST para el gen PRM 3, en base al alineamiento de secuencias de alpaca presentes en el NCBI.
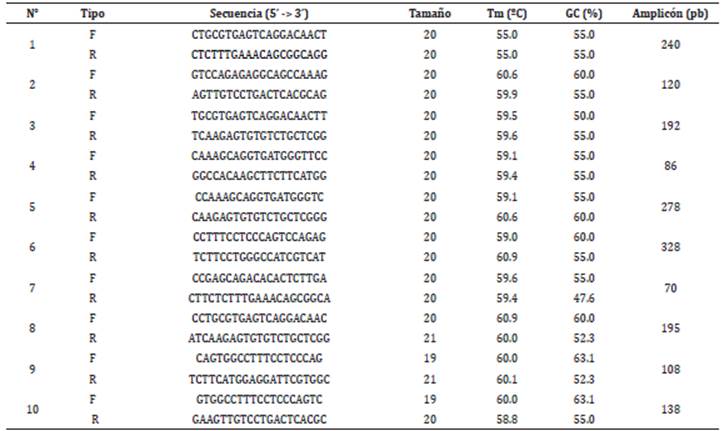
Posteriormente, se evaluaron posibles subproductos (horquillas, homodímeros y heterodímeros) con la herramienta oligoanalyzer (https://www.idtdna.com/pages/tools/oligoanalyzer). (Tabla 2). Los valores que se observar se dan por la valoración de la estabilidad termodinámica (AG°) de las interacciones entre los primers. En ambas parejas de primers se pueden producir siendo más o menos estables, ya que los valores de energía libre de Gibbs son cercanos a 0.
Tabla 2 Evaluación de posibles subproductos de PCR in silico de los primers PRM3-P31 y PRM3-P32 mediante la herramienta OligoAnalyzer.
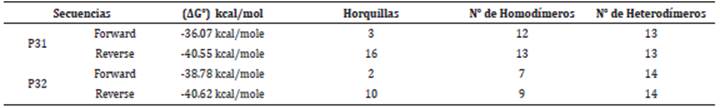
La evaluación in silico del primer PRM3-P31 in silico, determinó una amplificación de 240 pb, que el forward está ubicado entre los nucleótidos 9457395 - 9457396 pb y el reverse entre 9457156 y 9457175 pb (Figura 1) dentro del cromosoma 18. Sin embargo, se observó que tiene localización con otras porciones en diferentes cromosomas, pero con productos mayores a 600 pb (Tabla 3).
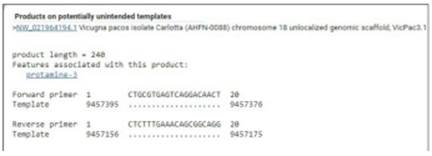
Figura 1 Alineamiento de la secuencia nucleotídica de ADN reverse) obtenida con el programa Primer-BLAST
Tabla 3 Determinación de otras posibles zonas de unión del par de primer PRM3-P31 con el genoma VicPac3.1 de alpaca.

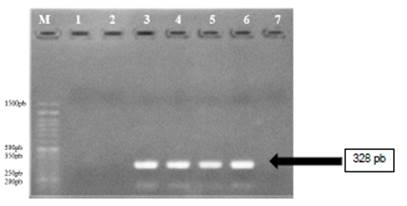
La evaluación del primer PRM3-P32 in silico, determinó una amplificación de 328 pb, el forward está entre los 9457395 y 9457376 pb y el reverse está entre los 9457180 y 9457199 pb (Figura 2) dentro del cromosoma 18. Sin embargo, también se observó que tiene localización con otras porciones en diferentes cromosomas; con diversos tamaños de amplicones (Tabla 4).

Figura 2 Alineamiento de la secuencia nucleotídica de ADN del genoma de la alpaca y el segundo par de primers PRM3-P32 (forward y reverse) obtenida con el programa Primer-BLAST
Tabla 4 Determinación de otras posibles zonas de unión del par de primer PRM3-P32 con el genoma VicPac3.1 de alpaca.

Respecto a la evaluación en vitro, se observó la amplificación esperada para el par de primers PRM3-P31 es de 240 pb (Figura 3), por lo que coindice el tamaño de producto obtenido in silico y observado en la tabla 1, valor que no corresponde a ningún otro punto de unión observado en la tabla 3.
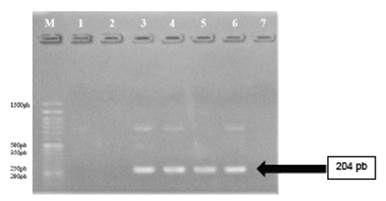
Figura 3 Primers PRM3-P31. Carril M: Marcador de peso 3 - 6: Repeticiones de amplicones de ADN de alpaca.
En el caso del par de primers PRM3-P32, la amplificación observada (Figura 4) coincide con el tamaño de producto esperado en la evaluación in silico; sin embargo, este valor es muy cercano a los puntos de localización 12 y 28 observados en la tabla 4.
DISCUSIÓN
Este es el primer reporte que desarrolla y valida primers para el gen protamina 3 (PRM3) en alpacas.
Identificación. Se realizó la búsqueda del gen dentro del genoma de la alpaca (GCF_000164845.3), ubicándolo en el cromosoma 18, lo que coincide con la ubicación del mismo gen en dromedarios, especie filogenéticamente relacionada. Así mismo, cumple las características indicadas en otros artículos 13.
Desarrollo de primers. Respecto al desarrollo de los primers, el no tener datos corroborados en el NCBI, el emplear como base la zona conservada obtenida del alineamiento del gen con el ARNm, nos brindó mayor confiabilidad de los resultados.
La evaluación de la energía libre de Gibbs (AG°), nos permite evaluar que tanta posibilidad existe de que se formen horquillas, homodímeros y heterodímeros 14 time consuming, and less sensitive. En ambos primers se observaron valores bastante cercanos, siendo la diferencia más resaltante la presencia de más horquillas y homodímeros en el par PRM3-P31; no obstante, esto no se vio reflejado en la evaluación in vitro con el gel de agarosa.
Validación de primers. Respecto a la validación in silico, esta se logró enfrentando los primers con el genoma completo presente en la base de datos del NCBI, determinando que los primers no son específicos. Esto podría deberse a que la secuencia del gen está presente en el cromosoma 18 pero no ordenado ni orientado en este, lo que también podríamos verificar por la secuencia del ARNm. Para el par PRM3-P31, este obtuvo 5 distintos puntos de unión en diversos cromosomas, mientras que el par PRM3-P32, tiene en total 44 distintos puntos; esto hace que el primer par sea mucho más específico que el segundo par.
Otro punto importante es el tamaño de los amplicones. Para el par PRM3-P31, el amplicón más pequeño fue el obtenido con la localización del gen PRM3 (204 pb) y al observar el gel de este par, se observó que efectivamente cumplió con la localización, verificándose así la especificidad para encontrar al gen PRM3. En el caso del par PRM3-P32, de los 44 puntos de localización, determinamos la cercanía de 3 puntos en distintos cromosomas: 1 en el cromosoma 18 con un amplicón de 328 pb, 2 en el cromosoma 4 con un amplicón de 307 pb y 3 sin localización específica con un amplicón de 331 pb. Al observar el gel, se observó un tamaño de 328 pb correspondiente al gen PRM3; sin embargo, la cercanía de las otras zonas hace dudar que realmente corresponda al gen. Por otro lado, se observa en el gel otra banda mucho más débil y de mucho menor tamaño. Estas bandas podrían ser amplificados de los mismos primers o por la unión a otras zonas del genoma; lo que descarta la fiabilidad del empleo de este, dejando al par PRM3-P31 como mejor elección.
En conclusión, este estudio ha permitido desarrollar y evaluar primers para detectar el gen Protamina 3 en alpacas. Este estudio da pie a iniciar la investigación de este gen en alpacas.