1. Introducción
La familia Cactaceae incluye entre 1500 a 2000 especies (Porras-Flores et al., 2017), las cuales son de gran interés por las adaptaciones físicas y fisiológicas; además por la coevolución con microrganismos endófitos, como las bacterias, Rhizobium, Burkholderia y hongos Aspergillus, Fusarium, ayudando a la fijación biológica del nitrógeno atmosférico, a la solubilización de los fosfatos, y a la protección contra estrés salino y térmico, permitiendo colonizar ambientes rocosos, con suelos superficiales y déficit hídrico (Rodríguez-Mendoza et al., 2021).
Los cladodios, las flores y los frutos de las especies del género Opuntia son claves como vectores de polinización, pero son utilizados en la industria agrícola, alimentaria y farmacéutica, razón por la cual algunos taxones han llegado a ser catalogados como especies en vía de extinción (Cardeño-Londoño & Rodríguez-Herrera, 2020; Dennis et al., 2007). Para mantener las poblaciones naturales sanas y hacer uso sostenible de las mismas es importante conocer su variabilidad genética, para proponer planes de conservación y manejo. A nivel molecular, se utilizan diferentes marcadores moleculares para evaluar la variabilidad genética de estas especies, caracterizar el germoplasma, conservarlas, mejorarlas genéticamente y/o realizar análisis de genética de poblaciones (Dennis et al., 2007; Hoisington et al., 1999).
Sin embargo, para realizar ese tipo de análisis es indispensable obtener ácido desoxirribonucleico (ADN) en altas cantidades y de buena calidad, con procesos de extracción que sean económicos, eficientes y rápidos y, que permitan obtener resultados confiables y reproducibles (Mondragon-Jacobo & Bordelon, 1996; Wang et al., 1998; Mondragón-Jacobo et al., 2000). Generalmente se utilizan kits comerciales porque reúnen todas esas características, aunque se ha evidenciado que los tejidos contienen diferente cantidad de polifenoles, polisacáridos y componentes ácidos que afectan el proceso de extracción y los futuros análisis (Shedbalkar et al., 2010; Sánchez-Hernández & Gaytán-Oyarzún, 2006). Para contrarrestar estas dificultades se han descrito protocolos de extracción de ADN en plantas utilizando el método bromuro de cetiltrimetilamonio (CTAB) y modificaciones de este, que se basan en ajustar las concentraciones según las especies estudiadas (Doyle & Doyle, 1990; Sue-Porebski et al., 1997).
En las especies de Cactaceae, los procesos de extracción de ácidos nucleicos se ven afectados por la cantidad y presencia en la zona cortical (en las células del tejido de clorénquima y parénquima) de mucílago, sustancia viscosa, compuesta por polisacáridos (pectina) y metabolitos secundarios en altas cantidades (Lorenzo et al., 2017). También, el mucílago se presenta en otras familias suculentas como Asphodelaceae, Portulacaceae, Malvaceae (Sánchez-Olaya et al., 2019) entre otras, su contenido es variable entre los taxones, puede unirse con el agua y se libera al causar lesiones en la planta (Nobel & Cavelier, 1992).
En las especies cultivadas y silvestres de Opuntia se ha encontrado mucílago en los cladodios e hipanto (De la Cruz et al., 1997). También, se ha reportado una tendencia a la formación de complejos insolubles entre los polisacáridos y los ácidos nucleicos, lo cual dificulta su aislamiento a partir de material fresco (Shedbalkar et al., 2010; De la Cruz et al., 1997; Guillemaut & Maréchal-Drouard, 1992), he inhiben la actividad de las taq polimerasas y las enzimas de restricción (Pandey et al., 1996). Durante la extracción, cuando el mucílago se libera en el macerado del tejido, se forma una textura viscosa parecida al pegante (Rowland & Nguyen, 1993), la cual se adhiere a los componentes celulares y moléculas orgánicas como proteínas y fenoles, dificultando su separación del ADN en los siguientes pasos de extracción.
Utilizando plantas ricas en polisacáridos, se han reportado métodos de extracción de ADN partiendo de material fresco de las hojas (Doyle & Doyle, 1990; Rowland & Nguyen, 1993; Haymes, 1996; Dellaporta et al., 1983). En cactus, incluyendo Opuntia se ha aislado ADN de pequeños segmentos de tejido (Wang et al., 1998; De la Cruz et al., 1997; Keb-Llanes et al., 2002), utilizando CTAB o dodecil sulfato de sodio (SDS) para la lisis celular. Sin embargo, no se modificaron para realizar extracciones de especies de Opuntia y ecotipos (Keb-Llanes et al., 2002; Cota-Sánchez et al., 2006), ni para las especies del presente estudio (Austrocylindropuntia subulata, Opuntia dillenii y Opuntia ficus-indica).
Para evitar el problema del mucílago, también se ha realizado la extracción a partir de las espinas, con un protocolo extenso que incluye el secado de las espinas y el rompimiento del tejido, teniendo bajos rendimientos de ADN (Fehlberg et al., 2013). En contraste y con el fin de aumentar los rendimientos y disminuir el tiempo de extracción, se ensayaron diferentes métodos de extracción de ADN para cuatro especies del género Opuntia (O. dillenii, O. elata, O. ficus- indica y O. robusta), encontrándose un método combinado que permitió obtener ADN genómico con el mejor rendimiento y calidad, que puede ser utilizado para analizar la diversidad genética de las poblaciones a través de microsatélites (Raimundo et al., 2018).
Algunos estudios sobre análisis de la composición química de las cactáceas, específicamente de O. ficus-indica, mostraron que el contenido de calcio, cenizas de base húmeda, cobalto, cobre, fósforo, hierro, magnesio, proteínas y sílice varían entre las distintas especies y dentro de una misma especie (Guzmán & Chávez, 2007; Abraján, 2008). Para O. ficus-indica se demostró la variación del contenido de calcio, carbohidratos, cenizas, fibra, hierro, minerales, potasio, proteínas, sodio y vitamina C de acuerdo con la edad de los cladodios (Guzmán & Chávez, 2007). En el mucílago extraído de los cladodios de esta especie se encontraron azúcares (% en moles) como arabinosa (44.54%), galactosa (18.16%), (ramnosa 6.58%), xilosa (23.98%), y ácido galacturónico (6.80%) (Abraján, 2008). En O. dillenii se presentaron azúcares como arabinosa (38.8%), galactosa (33%), glucosa (5.1%), ramnosa (15.70%), y xilosa (5.1%) (Kalegowda et al., 2017; Qiu et al., 2007). Para las tres especies de nuestro interés (A. subulata, O. dillenii y O. ficus-indica) el contenido de cenizas, fibra, proteína, Ca, P y compuestos fenólicos presentes en el mucílago es variable (Pérez et al., 2015; Abraján 2007).
Teniendo en cuenta las diferencias en la composición química de los cladodios y el mucílago de las especies estudiadas, y su efecto en la obtención de ADN de buena calidad, en este trabajo se propusieron los siguientes objetivos: i) evaluar diferentes protocolos de extracción de ADN, ii) encontrar el método que brinde el mayor rendimiento y calidad de ADN, y iii) probar el protocolo mediante la amplificación de dos regiones del cloroplasto rpl16 y trnL-trnF.
2. Metodología
2.1 Selección y colecta del material vegetal
Muestras de tallo de A. subulata, O. dillenii y O. ficus-indica fueron recolectadas en las regiones semiáridas de los municipios de Cucaita, Motavita, Oicatá, Ráquira, Sáchica, Samacá, Soatá, Soracá, Sutamarchán, Tipacoque y Tunja, en el departamento de Boyacá, y fueron mantenidas en macetas en el laboratorio del grupo de estudios en Genética y Biología Molecular (GEBIMOL), de la Escuela de Ciencias Biológicas de la Universidad Pedagógica y Tecnológica de Colombia, sede Tunja (UPTC), hasta la extracción de ADN.
El muestreo fue completamente al azar, dejando mínimo 1 km de distancia entre los individuos muestreados y se seleccionaron individuos maduros y saludables. El número de individuos colectados en cada municipio dependió de la abundancia de los taxones en cada lugar. En total se recolectaron 63 segmentos.
2.2 Extracción y amplificación del intrón rpl16 y el espaciador intergénico trnL-trnF
Los procedimientos de extracción de ADN y amplificación por reacción en cadena de la polimerasa (PCR) fueron realizados en el Laboratorio GEBIMOL.
Para superar los problemas de contaminación del ADN debido a la presencia de componentes ácidos, polifenoles, polisacáridos y proteínas, se modificaron y adaptaron los métodos de extracción de ADN de los protocolos de Albesiano y Terrazas (2012), y Martínez-González et al., (2017); además de utilizar columnas de purificación del kit de purificación de ADN genómico de plantas PureLink®. Se utilizó el kit de purificación de ADN genómico de Wizard (Promega Corp., Madison, WI, USA), siguiendo las instrucciones del fabricante e incluyendo un paso adicional. Finalmente, se utilizó el método propuesto por Phillips et al., (2003), combinado con el protocolo de limpieza propuesto por Falcón y Valera (2007), utilizado para extraer ácidos nucleicos de hojas de algodón (Gossypium hirsutum) (Rache-Cardenal et al., 2013). El método fue ajustado para obtener ADN de buena calidad y las modificaciones para cada protocolo se resumen en la tabla 1.
Tabla 1 Modificaciones a los protocolos de extracción de ADN utilizados.
| Protocolos de extracción ensayados | Modificaciones |
|---|---|
| Albesiano y Terrazas (2012) modificado | Buffer de lisis CTAB 2X. Eliminación de lípidos y proteínas: dos lavados adicionales con fenol y cloroformo. Precipitación y eliminación de sales: adición de 300 µl de etanol 76% y 10 mM de acetato de amonio durante 15 minutos a -20°C. |
| Martínez-González et al., (2017) modificado | Precipitación con: - 10 µl de NaCl (3M) con 260 µl de etanol (100%) durante 1 hora a -80°C. Centrifugar a máxima velocidad durante 20 minutos, lavar con etanol (75%). |
| Kit Wizard modificado | Eliminación de lípidos, proteínas y grasas: Adición de 400µl de PCIA (fenol: cloroformo: alcohol isoamílico), centrifugación a 14000 rpm por 5 minutos. |
| Rache-Cardenal et al., (2013) modificado | Buffer de lisis CTAB 2X. Precipitación con: - 10 µl de NaCl (2M) y 250 µl de isopropanol durante 30 minutos a -20°C. |
Cada uno de los protocolos se evaluó utilizando tejido subepidérmico fresco y pulverizado con nitrógeno líquido almacenado a -80°C, tomando una muestra de cada especie. El ADN de cada una de las muestras se cuantificó en un varioscanTM Thermo ScientificTM multimode microplate reader. El protocolo completo y modificado utilizado para extraer ADN de estas especies se describe a continuación:
Uno a dos fragmentos de cladodio de 1.5 cm2 se colocaron en un tubo eppendorf de 1,5 ml, se adicionaron 200 µl de 2x CTAB, se maceraron los fragmentos de tallo, se adicionaron 300 µl más de tampón 2x CTAB, se continuó macerando, luego se adicionaron 300 µl de 2x CTAB y se mezcló utilizando un vórtex. Las muestras se centrifugaron a 12 rpm durante 10 minutos, se eliminó el sobrenadante, se agregaron 500 µl de 2x CTAB y 7 µl de β- mercaptoetanol, se mezcló en vórtex y se colocaron los tubos en baño María 50°C por 50 minutos dando vórtex cada 10 minutos a las muestras. Pasado el tiempo, a cada muestra se le agregaron 50 µl de fenol y 50 µl de cloroformo, se agitó en vórtex y se centrifugó a 10000 rpm durante 5 minutos, se rescató la fase superior y se colocó en un tubo limpio. Posteriormente, se agregaron 50 µl de fenol y 100 µl de cloroformo, se agitó utilizando un vórtex y se centrifugó a 10000 rpm durante 5 minutos, se rescató la fase superior con cuidado y se colocó en un tubo limpio. Se repitió el paso anterior. Luego se agregaron 100 µl de cloroformo, se mezcló en vórtex, se centrifugó a 10000 rpm durante 5 minutos y se rescató la fase superior en un tubo limpio. Para precipitar el ADN se adicionaron 10 µl de NaCl (2M) y 250 µl de isopropanol y se dejaron las muestras a -20°C durante 30 minutos. Pasado este tiempo se centrifugaron a 12000 rpm, 4°C durante 20 minutos, se eliminó el sobrenadante, se lavó el precipitado con 1 ml de etanol al 90% frío, se dejó secar y se resuspendió el ADN en 50 µl de tampón TE, adicionando 2 µl de RNasa.
La identificación de los taxones recolectados se realizó utilizando como marcadores moleculares de cloroplasto la secuencia del espaciador intergénico trnL-trnF y el intrón rpl16. La amplificación de estos dos marcadores se realizó en tubos de 200 µl con 10X taq buffer, 0.2 mM de cada dNTP, 0.1 µM de cada primer, 25 mM de MgCl2, 1.25u de Taq DNA polimerasa (Thermo Scientific Inc., Waltham, M.A, USA.) y ADN a 30 ng/µl en un volumen total de 50 µl de reacción. Las condiciones de la PCR fueron las siguientes: denaturación inicial a 95°C por 3 minutos, 35 ciclos comprendiendo denaturación a 95°C por 30 segundos, 61°C para amplificar el gen rpl16, 62°C para el gen trnL-F y extensión a 72°C por 1.40 minutos, una extensión final a 72°C por 10 minutos. Se utilizaron los cebadores reportados por Albesiano y Terrazas (2012), los cuales fueron sintetizados por Macrogen.
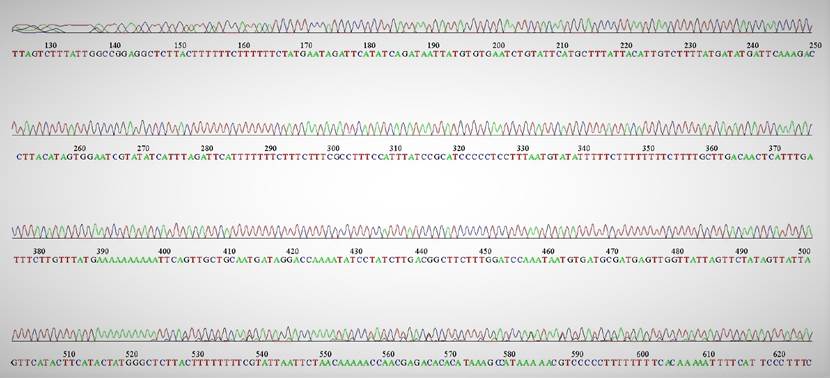
Los productos de PCR fueron visualizados por electroforesis en gel de agarosa al 1%. Los amplicones obtenidos se secuenciaron por Sanger dos sentidos en Macrogen con 30 µl del producto de PCR (no purificado) a una concentración de 100 ng/µl y 2 µl de cada uno de los cebadores a una concentración de 5 pmoles/µl (5pM). Los cromatogramas de las secuencias fueron analizados, editados y ensamblados utilizando el programa Sequencher 5.4.6.
2.3 Edición y ensamblaje de las secuencias
Se realizó en el programa CLC Genomics Workbench, versión 3.6.5, usando como estándar una secuencia obtenida del GenBank (Opuntia monacantha: https://www.ncbi.nlm.nih.gov/nuccore/MT359369.1).
Las reacciones de secuenciación fueron realizadas en la Unidad Genómica de Macrogen (Korea), utilizando como método de rutina la técnica de Sanger, y la electroforesis en secuenciador automático de capilares (Applied Biosystems 3500xL Genetic Analyzer).
3. Resultados y discusión
3.1 Selección y colecta material vegetal
Se sembraron 63 segmentos de los cuales 18 correspondieron a A. subulata, 10 a O. dillenii y 35 a O. ficus-indica. Los resultados de cuantificación y calidad del ADN extraído utilizando tejido pulverizado y fresco fueron mejores al trabajar con tejido fresco (resultados no mostrados), por lo cual los ensayos subsecuentes se realizaron utilizando tejido fresco.
3.2 Amplificación del gen rpl16 y el espaciador intergénico trnL-trnF
La calidad del ADN se consideró buena cuando se obtuvo una proporción de absorbancia A260/230 entre 1.8 a 2.2 y A260/280 entre 1.8 a 2.0. Las relaciones de absorbancia 260/230 menores a 1.8 indican presencia de EDTA, carbohidratos y fenol, mientras que relaciones 260/280 menores a 1.8 indican contaminación por proteínas, fenoles u otros contaminantes que se absorben a 280 nm (Sambrook et al., 1982). De esta forma, ensayando el protocolo de Albesiano y Terrazas (2012), se obtuvieron relaciones 260/280 muy bajas (0.1) y 260/280 mayores a 2,2; aunque las modificaciones que se realizaron al protocolo como aumento de lavados con fenol y cloroformo incrementaron los valores 260/280, éstos no superaron 1.0.
Con el protocolo propuesto por Martínez-González et al. (2017), se observaron valores mayores a 2.2 para la relación 260/280, los cuales disminuyeron con las modificaciones realizadas al protocolo, que incluyeron un paso de precipitación con NaCl 3 M. Las relaciones 260/230 mejoraron con la modificación; sin embargo, solo en una de las especies se llegó a un valor de 1,8 para la relación 260/280. Resultados similares fueron obtenidos utilizando columnas de purificación y el kit de extracción Wizard con modificaciones, obteniéndose un aumento en la relación 260/280, pero disminución en la relación 260/230 (valores menores a 0,7, Figura 1A). A pesar de la mejoría en la calidad del ADN, los contaminantes inhibieron la amplificación de los genes de interés.
Respecto a la cantidad de ADN obtenida, es de resaltar la alta concentración alcanzada aplicando el protocolo de Martínez-González et al. (2017). Este resultado puede explicarse teniendo en cuenta que se estaría sobreestimando la cantidad por contaminación del ácido nucleico con fenol (Figura 1B).
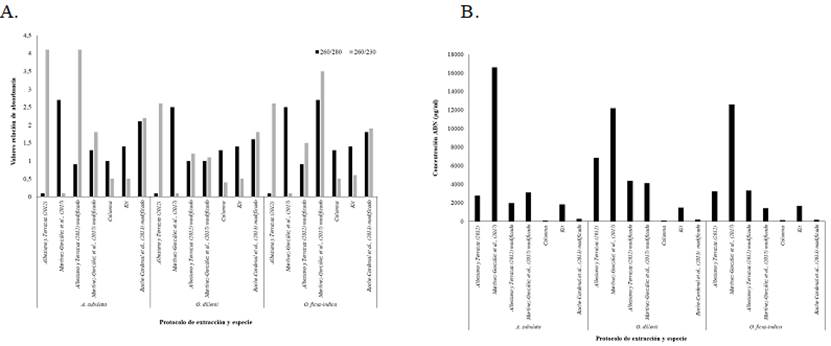
Figura 1 Cuantificación ADN obtenido utilizando diferentes protocolos y tejido fresco de una muestra de cada taxón. A) Calidad del ADN según las relaciones de absorbancia 260/280 y 260/230. B) Concentración del ADN.
Finalmente, después de realizar diferentes modificaciones al protocolo utilizado para extraer ADN de hojas de algodón (Rache-Cardenal et al., 2013), se obtuvo ADN de las tres especies evaluadas, de buena calidad (relaciones 260/280 y 260/230 entre 1.6-2.1 y 1.8-2.2, respectivamente, Figura 1A) lográndose amplicones del intrón rpl16 y el espaciador intergénico trnL-trnF en cada una de las muestras analizadas (Figura 2). Los productos de PCR se enviaron a secuenciar, obteniéndose buenos electroferogramas con picos muy bien definidos de buena resolución (Figura 3).
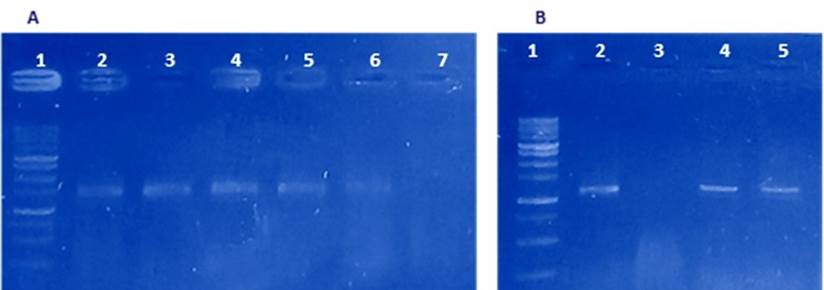
Figura 2 Amplificación del gen rpl 16 y del espaciador intergénico trnL-trnF. A) rpl 16: (1) Marcador de peso molecular 1Kb, (7) Control negativo, (2) A. subulata, (3 y 4) O. dillenii, (5 y 6) O. ficus-indica. B) Espaciador intergénico trnL-trnF: (1) Marcador de peso molecular 1Kb. (3) Control negativo, (2) A. subulata, (4) O. dillenii, (5) O. ficus-indica.
3.3 Edición y ensamblaje de las secuencias
Se editaron y ensamblaron 56 secuencias correspondientes a 224 reacciones; sin embargo, solo se cargaron en GenBank las secuencias de una muestra de cada taxón. Opuntia ficus-indica (OM685013) que tiene un alto grado de similaridad, 98%, con O. monacantha MT359369.1, lo cual puede explicarse por una mala identificación taxonómica de esta última especie. Austrocylindropuntia subulata (ON241265) y Opuntia dilleni (ON256561).
3.4 Discusión
En especies de cactáceas, distintos protocolos de extracción de ADN se utilizan con éxito para análisis de diversidad genética con diferentes marcadores moleculares (Mondragón-Jacobo et al., 2000; Mihalte et al., 2008; Barbosa et al., 2020; Díaz et al., 2006; Rosas-Reinhold et al., 2022; Realini et al., 2015; Yáñez et al., 2016; Mondragón-Jacobo et al., 2000; Paz-Guerrero et al., 2019). En particular, para dos de las especies de interés en este estudio (O. ficus-indica y O. dillenii) se encontraron varios reportes con procedimientos de extracción de ADN; en la mayoría de los estudios se menciona la dificultad para extraer su ADN debido a las diferencias en contenido de mucílago que presentan (Raimundo et al., 2018). En esos reportes se identificó la necesidad de realizar diferentes modificaciones a los protocolos para obtener ADN de buena calidad, concordando con los ensayos que se realizaron en este estudio. Sin embargo, cabe destacar que para la especie A. subulata no se encontraron protocolos de extracción de ADN y que fue difícil obtenerlo, debido a que presenta diferencias morfológicas y de contenido de mucílago que dificultan su manejo y la extracción de ADN de buena calidad, con protocolos rápidos y económicos.
Parece ser que A. subulata presenta mayor cantidad de mucílago, el cual se adhiere al ADN y actúa como inhibidor de PCR. A pesar de las modificaciones realizadas a las metodologías ensayadas en este estudio, principalmente respecto a componentes del buffer lisis y utilización de diferentes procesos de precipitación, se observó presencia de sales catiónicas en el ADN las cuales disminuyeron la calidad del mismo e inhibieron la amplificación de los genes de interés.
En este estudio, aunque se combinaron protocolos de extracción usando kit con pasos de purificación adicionales, aumentaron los valores de las relaciones 260/280 y 260/230, pero no se alcanzó a tener relaciones entre los rangos sugeridos por Sambrook et al. (1982). Mientras que Raimundo et al. (2018), usaron un protocolo en el cual combinaron lisis con CTAB y SDS y DNeasy® plant mini kit, obteniendo relaciones de absorbancia 260/280 de 1.91 y 2.01 para O. ficus indica y O. dillenii, respectivamente, plantas con contenido de mucílago variable.
La adaptación del protocolo modificando el proceso de precipitación y el buffer de lisis, permitió superar los problemas relacionados con la contaminación del ADN, tanto la formación de complejos insolubles del ADN con polisacáridos (De la Cruz et al., 1997; Guillemaut & Maréchal-Drouard, 1992), como con sales catiónicas de las tres especies en estudio, particularmente con A. subulata.
4. Conclusiones
Los valores por debajo de 1.8 en la relación de absorbancia A260/230 inhibieron la amplificación por reacción en cadena de la polimerasa, indicando presencia de impurezas como fenoles, hidratos de carbono y sales.
En la extracción del ADN de los taxones de cactáceas generalmente se usa el protocolo de CTAB en Cactoideae, Echinopsis, Lobiva y Trichocereus, pero este presenta problemas al utilizarse con los géneros de la subfamilia Opuntioideae, los cuales presentan especies poliploides y variaciones en el contenido de ceras, fibras y mucílago.
Un protocolo de extracción modificado permitió obtener ADN de buena calidad, a partir del cual se amplificaron las regiones rpl16 y trnL-trnF de cloroplasto de A. subulata, O. dillenii y O. ficus-indica, el cual se puede aplicar en estudios con recursos limitados, al ser de bajo costo. Este es el primer protocolo de extracción efectivo para obtener ADN de la especie A. subulata.