Introducción
La autoinmunidad se refiere a las anormalidades en la actividad y la función del sistema inmune que llevan a efectos deletéreos de una pérdida de tolerancia a autoantígenos. La respuesta inmune puede manifestarse a través de disfunción multiorgánica y desencadenar en enfermedades sistémicas como el lupus eritematoso sistémico (LES), que se caracteriza por la presencia de autoanticuerpos dirigidos contra antígenos nucleares que forman inmunocomplejos (IC), principalmente depositados en los órganos afectados1.
Entre las manifestaciones orgánicas del LES se encuentra el compromiso renal definido con nefritis lúpica (NL); este es el más temido, ya que incrementa de manera significativa la morbimortalidad. La NL es una glomerulonefritis mediada por IC que afecta a más del 60% de los pacientes con LES y se considera una causa importante de enfermedad renal crónica (ERC)2-4.
La incidencia y la severidad del compromiso renal varían entre individuos con LES, pero parecen estar asociadas a la etnicidad, ya que los afroamericanos, asiáticos e hispanos son más propensos a desarrollar enfermedad renal en comparación con pacientes euroamericanos5. A pesar de la disponibilidad de inmunoterapias agresivas como la ciclofosfamida y el micofenolato de mofetilo6, la ERC en pacientes con LES finaliza en un pobre desenlace clínico, pues estos medicamentos producen una vasta gama de efectos colaterales7. Por lo tanto, hay una necesidad constante y crítica de elucidar los mecanismos inmunes detrás de la NL para poder identificar nuevos biomarcadores que sean fácilmente evaluables y faciliten el diagnóstico y monitoreo de los pacientes, lo cual llevará al desarrollo de terapias mucho más efectivas con mejores resultados y menos efectos adversos.
En la presente revisión se exploró el estado del arte relacionado con estudios cuyo foco fuera el descubrimiento de nuevos marcadores de NL en individuos con LES usando abordajes transcriptómicos. Se entendió como transcriptoma a todos los transcritos que se producen en una célula bajo cierta condición fisiológica y que son clave para la compresión del efecto de la actividad biológica de un grupo de genes en el contexto de salud/enfermedad8. Por su parte, la transcripción se entendió como un proceso nuclear cuya activación depende de estímulos intra o extracelulares que activan cascadas de señalización para determinar cuáles genes deben expresarse o reprimirse según el tipo de estímulo inicial8.
Expresión diferencial de factores de transcripción en el LES y la NL
Diversos estudios han sugerido que existe una correlación marcada entre el LES y la expresión diferencial de diversos factores de transcripción (FT)9-13que juegan un papel central en la regulación de la expresión génica14, por lo que alteraciones en su regulación, estructura y función podrían tener un impacto importante en el desarrollo y función del sistema inmune.
Sui et al.15 realizaron un estudio con pacientes con LES y controles sanos en células mononucleares de sangre periférica (CMSP) cuyo objetivo principal era identificar FT diferencialmente expresos durante el LES. En dicho estudio se detectaron 92 de estos factores expresados diferencialmente: 78 regulados de forma aumentada y 14 regulados de forma negativa en individuos con LES en comparación con los controles sanos. Entre esos FT expresos diferencialmente, se encontró que la proteína activadora 1 (AP-1), el Pbx1 y el factor potenciador de miocitos 2 (MEF-2) estaban asociados a la patogénesis y eran potenciales biomarcadores de diagnóstico15.
Los FT FOXO forhhead juegan un papel importante en el control de la activación y proliferación de los linfocitos. Al respecto, Kuo & Lin16 realizaron un estudio en CMSP evaluando la desregulación de la expresión transcripcional de FOXO en pacientes con LES y artritis reumatoide (AR) en comparación con controles sanos, y encontraron que los niveles de transcripción de FOXO1 en pacientes con LES y AR activa fueron significativamente más bajos que los de controles sanos, relacionado posiblemente con la patogénesis16.
Las células T activadas, las células B y la secreción potenciada de citocinas proinflamatorias pueden dañar los tejidos y órganos. Varias citocinas proinflamatorias, tales como IL-4, IFN-g, IL-10, IL-17 y TGF-b, juegan funciones importantes durante el LES. T-bet y GATA-3 son los principales FT que conducen la diferenciación de los linfocitos Th1 y Th2, respectivamente.
Sui et al.14 encontraron niveles aumentados de ARNm de T-bet e IFN-γ y niveles disminuidos de ARNm de GATA-3 e IL-4 en pacientes con LES, lo que se ha correlacionado con enfermedad activa; estos autores también relacionaron el halotipo de riesgo de STAT-4 que tiene su función en la señalización del interferón tipo I y que se sobreexpresa por acción de algunos FT, incluidos AP-1 y NF-kB, y encontraron que el TF-IR5 es un mediador importante de la expresión activada por el receptor Toll-like de citocinas proinflamatorias tales como IFN tipo I y TNF-α, que además está relacionado con la expresión del gen STAT-4. Asimismo, Frangou et al.17 establecieron que la expresión de genes dirigidos por IRF-5 y STAT-4 podría distinguir a los pacientes con LES de individuos sanos.
MicroARN (miARN) y su papel en el LES y la NL.
La epigenética es el estudio de los cambios reversibles y potencialmente heredables en la expresión génica sin alteraciones del código genético. Entre las principales modificaciones epigenéticas se encuentra la metilación del ADN, la modificación de histonas y los miARN18, que son secuencias cortas de ARN no codificantes que regulan la expresión génica al bloquear la traducción de proteínas o inducir la degradación de ARNm19. Diferentes estudios han abordado el papel de los miARN como biomarcadores diagnósticos del LES y complicaciones frecuentes como la NL.
En relación con la epigenética asociada al LES, para desentrañar un papel potencial de los microARN en la hipometilación aberrante del ADN en el LES se realizó un perfil de microARN en las células TCD4+ de pacientes con la enfermedad y ratones con tendencia al lupus. Asimismo, se descubrió que tres microARN sobreregulados (miR‐21, miR‐148a y miR‐126) promovían la hipometilación de las células TCD4+. Además, las modificaciones de histonas y la metilación del ADN en regiones reguladoras de la secuencia miR-142 disminuyen su expresión, lo que contribuye a la activación de células TCD4+ y a la hiperestimulación de células B en LES17. Este mismo estudio reportó que un grupo de miARN (miR‐25, miR‐106b y miR‐21) se encontraba con expresión aumentada tanto en células B como en células T de individuos con LES, y que estos tuvieron la correlación más fuerte con enfermedad activa17. En otro estudio, Stagakis et al.20 encontraron que el miR-21 regulado negativamente afecta la expresión de PDCD4, un inhibidor selectivo de la traducción de proteínas de genes involucrados en las respuestas inmunes que regula las respuestas aberrantes de células T en el LES humano, lo que podría representar un biomarcador de enfermedad y un objetivo terapéutico en esta enfermedad.
El grupo de Nefrología de la Universidad Simón Bolívar viene trabajando activamente en la búsqueda de miARN como biomarcadores de daño renal en pacientes con LES: en un estudio observacional en el que se caracterizaron los perfiles de expresión diferencial de miARN en pacientes con LES con diferentes grados de NL y se compararon con pacientes con LES sin afectación renal e individuos de control sanos, Navarro-Quiroz et al.21 encontraron cinco microARN (miR-221-5p, miR-380-3p, miR-556-5p, miR-758-3p y miR-3074-3p) que en su conjunto permiten identificar, con altos valores de sensibilidad y especificidad y con valores predictivos positivos y negativos, individuos con afectación renal y LES. Estos resultados sugieren que dichas moléculas circulantes en plasma pueden ser potenciales biomarcadores diagnósticos de la NL en pacientes con LES. Es importante mencionar que el patrón diferencial observado en la abundancia de miARN puede tener implicaciones funcionales en la fisiopatología del daño renal por LES.
En otro trabajo, cuyo objetivo era identificar in silico las relaciones entre los microARN y los genes que codifican FT, ubiquitinación, metilación del ADN y modificaciones de histonas en el LES, Navarro et al.22 identificaron 226 miARNs con expresión diferencial. De manera interesante, Navarro et al.23 también observaron que alteraciones de miARN como hsa-miR-30a-5p, hsa-miR-16-5p, hsa-miR-142-5p y hsa-miR-324-3p están más asociadas a modificaciones postraduccionales, mientras que hsa-miR-16-5p, hsa-miR-374a-5p, hsa-miR-34a-5p, hsa-miR-31-5p y hsa -miR-1-3p se asocian con mayor frecuencia a genes relacionados con la susceptibilidad del LES; en este mismo trabajo se reportaron 24 miARN circulantes en plasma con abundancia diferencial, 14 descritos por primera vez en NL (hsa-miR-589-3p, hsa-miR-1260b, hsa-miR-4511, hsa-miR-485-5p, hsa-miR-584-5p, hsa-miR-543, hsa-miR-153-3p, hsa-miR-6087, hsa-miR-3942-5p, hsa-miR-7977, hsa-miR-323b-3p, hsa-miR-4732-3p y hsa-miR-6741- 3p).
Estos cambios en la abundancia de miARN podrían interpretarse como alteraciones en la red reguladora de miARN-mARN en la patogénesis de la NL antes del inicio clínico de la enfermedad, por lo tanto contribuyen a comprender el proceso de la enfermedad y es probable que allanen el camino hacia la identificación de biomarcadores para el diagnóstico temprano de LES.
Martínez-Ramos et al.24, usando TaqMan Human MicroRNA Array, Identificaron 377 miARN en células B CD19+ y células T CD4+, de los cuales cuatro (hsa-miR-143, hsa-miR-224, hsa-miR-10a y hsa-miR-345) tuvieron expresión aumentada en estas poblaciones celulares en individuos con LES respecto a individuos clínicamente sanos. En otro estudio, Kusaoi et al. 25 reportaron que los pacientes con LES presentan patrones de expresión distintos de miARN según la progresión de la enfermedad, además encontraron ocho miARN (hsa miR 494, hsa miR 188, hsa miR 501, mmu miR 298, HMP PREDICTED MIR61, HMP PREDICTED MIR78, hsa miR 296 y hsa miR 299 3p) que mostraron un patrón de expresión diferencial aumentada en pacientes con LES con enfermedad activa al compararlos con individuos con LES inactivo, lo que permite hipotetizar que estos miARN pueden estar involucrados en la progresión de la enfermedad y el daño de órganos.
Por otra parte, Jafari-Ghods et al.26 aislaron ARNm y miARN totales de muestras de sangre para ser usados en ensayos de microarreglos de ADN. Ellos describieron dos miARN (hsa-miR-766-3p y has-mir-5571-5p) con expresión significativamente reducida en sujetos con LES y afectación renal en relación a los individuos con LES y sin afectación renal. De manera interesante, los investigadores encontraron que el hsa-miR-766-3p puede jugar un rol trascendental en la vía PI3K-AKT-mTOR26.
Te et al.27 identificaron cinco miARN (hsa-miR-371-5P, hsa-miR-423-5P, hsa-miR-638, hsa-miR-1224-3P y hsa-miR-663) expresados diferencialmente en distintos grupos raciales (afroamericanos, europeos y americanos) asociados a NL, aunque solo el hsa-miR-342-3P se expresó diferencialmente en muestras de sujetos afroamericanos. Carlsen et al.28, en un estudio con 409 muestras de plasma de 364 participantes (pacientes con LES, sujetos de control sanos y sujetos de control con otras enfermedades autoinmunes) reportaron que cuatro miARN (miR‐142‐3p, miR‐106a, miR‐17 y miR‐20a) pueden ser biomarcadores confiables de LES y que un subconjunto específico de perfiles de miARN se asoció con NL. Todos los miARN característicos se dirigen a genes en las vías de señalización del factor de crecimiento transformante β. Otras dianas moleculares incluyen la regulación de la apoptosis, los receptores de citocinas, el desarrollo de células T y la organización del citoesqueleto. Estos hallazgos resaltan posibles vías desreguladas en el LES y sugieren que los patrones de miARN circulantes distinguen al LES de otros fenotipos inmunoinflamatorios.
Se ha reportado que la expresión intrarrenal de ciertos miARN tiene relación con la actividad de la enfermedad29-31. Por ejemplo, Krasoudaki et al.32 concluyeron que, en comparación con el tejido normal renal, un grupo de 24 miARN define la NL humana: nueve mostrando expresión aumentada y 15 mostrando expresión disminuida. Entre estos, el miR-422a exhibió la más alta regulación, cuya diana pareció ser la calicreína 4 (KLK4), una serina esterasa secretada con propiedades de remodelación de la matriz extracelular y angiogénica en la patogénesis de la NL. La familia de genes de calicreína tiene un papel importante en la regulación de la inflamación, apoptosis, coagulación y fibrosis en los riñones.
Sui et al.15 compararon la expresión de miARN en biopsias renales de pacientes con NL y controles normales, e identificaron 30 miARN regulados negativamente y 36 con expresión aumentada. En otro estudio realizado por Carlsen et al.28, siete miARN se expresaron estadísticamente de manera diferencial en plasma de pacientes con LES y la expresión de miR-142-3p y miR-181ª aumentó, mientras que la de miR-106a, miR-17, miR-20a, miR-203 y miR-92ª disminuyó. Además, la expresión de miR-342-3p, miR-223 y miR-20a disminuyó significativamente en pacientes con LES y nefritis activa.
MiARN, autoanticuerpos y sus implicaciones biológicas
Trabajos anteriores han mostrado que es posible agrupar pacientes con LES según la especificidad de autoanticuerpos33-36. Rai et al.33 identificaron patrones diferenciales de expresión de miARN en sujetos con LES con diferentes estados de autoanticuerpos y establecieron hipótesis acerca de sus implicaciones biológicas. En individuos con autoanticuerpos positivos para el antígeno nuclear extraíble (ENA, por su sigla en inglés para extractable nuclear antigen) (anti-ENA+), estos autores hallaron 28 miARN regulados negativamente. También encontraron que las vías que controlan el ciclo celular (regulación mediada por proteínas de la familia BTG, CHK y señalización de CDK5) y la remodelación del citoesqueleto (señalización de actina, de RhoGDI, de unión estrecha y la regulación de la motilidad de actina basada en Rho) estaban afectas en este subconjunto de pacientes.
En los pacientes con autoanticuerpos contra dsDNA positivos (antidsDNA+) se hallaron 33 miARN regulados positivamente que, a su vez, regulan el control de varias vías de señalización de citocinas tales como IL-6, IL-17, IL-4, IL-2, CXCR4, CNTF y IL-10. Se encontraron también tres miARN regulados negativamente y sin información biológica relevante31. En cuanto a los sujetos con Anti-dsDNA + y anti-ENA +, se evidenció que los miARN asociados con las vías de respuesta viral y del huésped están desregulados en los subconjuntos.
Los pacientes con LES muestran firmas transcripcionales sanguíneas únicas relacionadas con el IFN tipo 1 y granulocitos, y se sugiere que estas firmas se pueden utilizar para evaluar la enfermedad activa. La mayoría de los estudios se han centrado en las transcripciones o proteínas inducidas por IFN como biomarcadores y sufren limitaciones de tamaño de la muestra y heterogeneidad clínica y terapéutica inherente a la enfermedad, lo que dificulta la interpretación de los datos32.
¿Podrían los miARN y ARN mensajeros circulantes en orina correlacionarse con enfermedad renal activa?
Diferentes tipos de abordajes “ómicos” han explorado el potencial de la orina como biofluido ideal para el descubrimiento de nuevos biomarcadores del LES y la NL en particular, lo cual se debe básicamente a que la orina ofrece muchas ventajas sobre otros biofluidos, entre las que resaltan cuatro aspectos principales:
1) La concentración de proteínas, de pequeños metabolitos y de ciertos transcritos es más abundante en la orina que en plasma o suero.
2) La orina contiene muchos metabolitos que permiten predecir el desbalance de casi todas las rutas bioquímicas del cuerpo.
3) Comparado con el plasma, la orina es colectada de forma no invasiva.
4) La orina se considera como una biopsia no invasiva del riñón37,38.
En ese sentido, la búsqueda del biomarcador ideal de daño renal en pacientes con LES podría tener la respuesta en la orina39. Cárdenas-González et al.40 realizaron un perfil global de miARN urinarios e identificaron dos biomarcadores (miR-3201 y miR-1273e) que se correlacionaron con la NL; en los sujetos con NL ambos estaban regulados negativamente y se relacionaron con la presencia de inflamación glomerular endocapilar. Estos miARN permitieron discriminar con una fuerte asociación estadística entre pacientes con enfermedad renal versus sujetos sanos o con LES40. Asimismo, en el estudio de Abulaban et al.41 se reportó que los niveles de miR-125a, miR-150 y miR-155 en orina están asociados con la expresión de biomarcadores de NL-Panel en niños, lo que sugiere un papel de estos en el diagnóstico de la NL en edad infantil.
Se ha propuesto que los miARN exosomales urinarios pueden reflejar de manera precisa la disfunción renal y el daño estructural en el riñón, haciendo de estas pequeñas moléculas excelentes dianas para la exploración de biomarcadores de enfermedades del tracto urinario42-45. Solé et al.46 reportaron que miR-21, miR-150, y miR-29c se correlacionaron con NL activa y podrían predecir un riesgo aumentado de progresión a ERC. Además, se ha reportado un aumento en los niveles de miR-146a y miR-29c exosomal urinario en pacientes con NL activa y se sugiere que estos pueden servir como predictores de fibrosis renal temprana18. Otro estudio reciente encontró que la expresión de miR-3135b, miR-654-5p y miR-146a-5p en exosomas urinarios se correlaciona con NL clase IV con buena eficacia diagnóstica47.
Guan et al.48realizaron un estudio en el que se midieron los niveles urinarios de miR-221, miR-222, miR-339-3P y miR-339-5P involucrados en la regulación de la molécula 1 de adhesión intercelular urinaria (ICAM-1), un potencial biomarcador de la NL y encontraron que los niveles de sedimento urinario de miR-339-3P están significativamente asociados con proteinuria, y que miR-221 y miR-222 están inversamente relacionados con el anti-dsDNA en suero. Además, miR-221 se correlacionó específicamente con los niveles séricos de C3.
En un análisis de transcriptoma de sedimento urinario de pacientes con LES, Jakiela et al.49 reportaron que en pacientes con NL activa hubo una marcada regulación positiva de transcritos de respuesta inmune tipo I relacionados con células T (tales como CD3G) y, a la inversa, genes de respuesta inmune tipo II (tales como GATA3 y CCL17) significativamente reducidos. Estos resultados demuestran que una desregulación a nivel del control de la expresión génica tiene fuertes repercusiones en la evolución de la enfermedad renal.
Conclusiones
La búsqueda de un biomarcador que permita diagnosticar el compromiso renal en pacientes con LES en etapas tempranas es un campo activo de investigación, pues en la actualidad solo la biopsia renal puede mostrar con relativa sensibilidad el daño histológico. Sin embargo, por ser esta una técnica invasiva y por la imposibilidad de aplicarla de forma serial para seguimiento de pacientes con NL, se hace necesario seguir explorando la utilidad diagnóstica de nuevos marcadores en fluidos biológicos cuya recolección sea menos invasiva. En ese contexto, diversas moléculas producto de la regulación de la expresión génica a nivel del riñón surgen como potenciales dianas para diagnóstico. La Tabla 1 recoge información de diversos miARN que han sido encontrados en fluidos biológicos y que han mostrado una fuerte asociación con daño renal en pacientes con LES. Se prevé que la combinación de varios biomarcadores de LES y NL podrá direccionar de forma eficiente el tratamiento de estos pacientes.
Tabla 1 MicroARN identificados en diversos fluidos biológicos con potencial diagnóstico del lupus eritematoso sistémico.
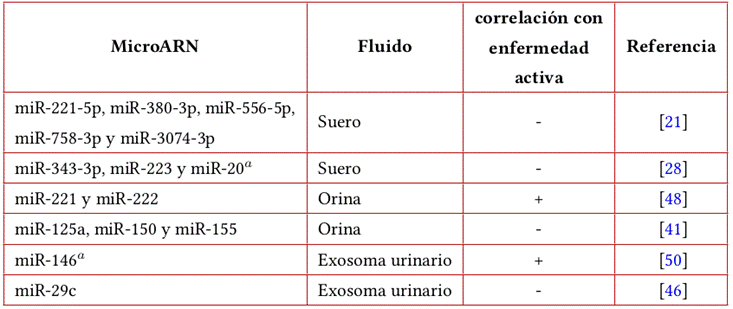
Fuente: elaboración con base en Wu et al.18.
Consideraciones éticas
Los autores declaran que los procedimientos seguidos se realizaron conforme a las normas éticas del comité de experimentación humana responsable y de acuerdo con lo establecido por la Asociación Médica Mundial en la Declaración de Helsinki; que han seguido los protocolos de su centro de trabajo sobre la publicación de datos de pacientes, y que han obtenido el consentimiento informado de los pacientes y/o sujetos referidos en el artículo.















