Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Revista Colombiana de Ciencias Pecuarias
Print version ISSN 0120-0690On-line version ISSN 2256-2958
Rev Colom Cienc Pecua vol.20 no.1 Medellín Mar./Jan. 2007
COMUNICACIONES BREVES
Polimorfismos en la longitud de fragmentos amplificados (AFLP´s) a partir de muestras de sangre almacenadas en tarjetas FTA® para la especie Cavia porcellus Lin. (Rodentia: Caviidae).
William Burgos-Paz1, Zoot; Carol Rosero-Galindo2, Biol, MSc; Heiber Cárdenas-Henao2, Biol, MSc; Carlos Solarte-Portilla 1, Zoot, PhD.
1Grupo Producción y Sanidad Animal, Facultad de Ciencias Pecuarias, Programa de Zootecnia, Universidad de Nariño, Ciudad Universitaria Torobajo, A.A.1175, Pasto, Colombia; 2Laboratorio de Biología Molecular, Departamento de Biología, Facultad de Ciencias, Universidad del Valle, A. A. 25360, Cali, Colombia.
Williamobur@yahoo.com
(Recibido: 28 febrero, 2006; aceptado: 8 febrero, 2007)
Resumen
Una manera eficaz de establecer el grado de variabilidad entre y dentro de poblaciones, es a través del análisis de polimorfismos de ADN con marcadores moleculares como los AFLP`s. En este artículo se presenta una metodología que combina la utilización de tarjetas de FTA® (Whatman Bioscience, Cambridge) para colección y conservación de muestras de sangre, con los procedimientos de extracción de ADN y obtención de marcadores AFLP´s, aspectos sobre los cuales no existen antecedentes para la especie Cavia porcellus. Se utilizaron muestras de ADN procedentes de tres poblaciones, dos criollas y una mejorada genéticamente obtenida a partir de un pie de cría procedente del Perú y sometida a selección en Colombia durante varias generaciones. Todos los animales procedieron de la Granja “Botana”, propiedad de la Universidad de Nariño, Pasto-Colombia. Para la detección de polimorfismos en la longitud de los fragmentos (AFLP`s) se utilizaron uno, tres y cinco discos FTA® de 1.2 mm, cada disco con aproximadamente 25 ng de ADN. Los ensayos indicaron que los mejores productos de amplificación, para la visualización de AFLP´s, se obtuvieron de muestras con tres discos de FTA® por individuo, lo que sugiere que con esta metodología, 75 ng de ADN por animal son suficientes para detectar polimorfismos de alta calidad en el genoma de Cavia porcellus. Se recomienda el uso de las tarjetas de FTA® para el estudio genético de poblaciones de Cavia porcellus, con las modificaciones metodológicas descritas en este artículo para marcadores AFLP´s.
Palabras Clave: AFLP´s, Cavia porcellus, FTA.
Introducción
Para analizar genéticamente a un individuo se requiere inicialmente el aislamiento de su ADN. Las exigencias en la calidad y cantidad de ADN aislado varían de acuerdo con el marcador genético que seleccione el investigador. Sin embargo, pese al gran número de protocolos existentes para la extracción de ADN, no todos tienen el mismo grado de reproducibilidad en los laboratorios por cuanto los métodos de conservación de la muestra tales como sangre u otros tejidos (11) difieren en el control de la acción de las DNAasas después de la toma de la muestra.
En la mayoría de los vertebrados, el tejido comúnmente escogido para la obtención de ADN de alta calidad es la sangre (11). Sin embargo, en los cuyes dadas las características de su hemograma (5, 8), la coagulación de la sangre ocurre de manera casi instantánea después de la toma de la muestra, lo que dificulta obtener una muestra óptima para llevar a cabo los análisis del ADN.
Las tarjetas FTA® (Whatman Bioscience, Cambridge, United Kingdom) están diseñadas para la colección, almacenaje y posterior purificación del ADN. Dichas tarjetas están constituidas de un papel filtro impregnado con una fórmula química patentada por la casa fabricante. La fórmula está diseñada para producir lisis de las membranas celulares y desnaturalización de las proteínas y de las enzimas con capacidad para desnaturalizar los ácidos nucleicos, los cuales quedan atrapados en una matriz tan pronto entran en contacto con el papel, con lo cual se reduce el riesgo de contaminación (15). Los componentes restantes de la muestra son eliminados mediante el lavado con un reactivo, producido por la misma casa fabricante.
Las principales ventajas del uso de estas tarjetas, frente a métodos convencionales de aislamiento de ADN son las siguientes:
1. Facilidad para la colección y transporte de las muestras, ya que en el caso de sangre se requiere una o dos gotas (13).
2. Almacenamiento de gran cantidad de muestras en un menor espacio (9).
3. Conservación de la muestra a temperatura ambiente por largos períodos (2, 10).
4. Alta sensibilidad en la detección de polimorfismos de ADN vía PCR, dado que el ADN que contiene la tarjeta está libre de compuestos como proteínas, DNAsas y sustancias tampón utilizadas en los procesos convencionales de extracción y que pueden afectar la calidad del ADN (13).
5. Rápida purificación de la muestra (16).
Aunque no es posible cuantificar el ADN aislado en la tarjeta de celulosa, se estima que un disco de 1.2 mm puede contener 25 ng de ADN (15).
La técnica AFLP (del inglés Amplified Fragment Length Polymorphisms) combina la aplicación de enzimas de restricción con el poder de la PCR a nivel de todo el genoma del individuo (4). Con esta técnica se utilizan entre 100 y 250 ng de ADN de alta pureza para generar un gran número de bandas sin previo conocimiento del genoma (14).
En el presente estudio, se evaluó el uso de las tarjetas FTA® para la colección, almacenamiento y transporte de las muestras de sangre de Cavia porcellus y además se estandarizó el protocolo para detectar polimorfismos mediante el uso de los marcadores moleculares AFLP´s.
Materiales y métodos
Muestras
Los ejemplares utilizados para este trabajo procedían de la Granja “Botana”, propiedad de la Universidad de Nariño, situada a nueve kilómetros al sur de la ciudad de Pasto, en la República de Colombia a una altura de 2820 m.s.n.m, una temperatura promedio anual de 12.7οC, 967 mm de precipitación media anual y una humedad relativa del 73% (6).
En los análisis se incluyeron ejemplares de tres poblaciones, dos “criollas” y una mejorada genéticamente, conocida como “peruana”, para lo cual se tomaron muestras de sangre de 12 individuos por población.
Colección y almacenamiento de las muestras de sangre en tarjetas FTA®
Las muestras de sangre se obtuvieron mediante punción cardíaca (véase Figura 1). La cantidad extraída varió entre 0.5 y 2.5 ml por animal. Dos gotas de sangre fueron aplicadas directamente a las tarjetas FTA® debidamente marcadas, las cuáles, según lo indicado por la casa fabricante (15), se dejaron secar a temperatura ambiente durante una hora y posteriormente se marcaron, envolvieron en papel aluminio y se enviaron al Laboratorio de Biología Molecular de la Universidad del Valle en la Ciudad de Cali para realizar los procedimientos de extracción de ADN y obtención de los marcadores AFLP´s (véase Figura 2).
Purificación de las muestras para la obtención de ADN
De cada muestra de sangre almacenada en la tarjeta se extrajeron uno, tres y cinco discos FTA® de 1.2 mm, usando un sacabocados, y se colocaron en un tubo Eppendorf.
Para la purificación de los discos de ADN se adicionaron 200 µl de “FTA Purification Reagent” por disco a cada tubo y se incubó por cinco minutos agitando por inversión. Posteriormente, sin remover los discos del tubo, se extrajo y descartó el Reagente; este mismo procedimiento se repitió tres veces (15).
Posteriormente se adicionó en cada tubo, 200 µl de tampón TE (10 mM Tris-HCl, 0.1 mM EDTA, pH 8.0) por disco y nuevamente se incubó por cinco minutos, agitando por inversión. Se descartó el tampón TE sin remover los discos del tubo y se repitió el proceso dos veces. Una vez descartado el tampón TE se dejaron secar los discos a temperatura ambiente durante una hora (15).
Amplificación de fragmentos polimorficos a partir de las muestras de ADN almacenadas en los discos FTA®
La amplificación del ADN se realizó con el kit, AFLP® Analysis System I de Invitrogen, siguiendo el protocolo de Vos et al (14), las recomendaciones de la casa fabricante (7) y los ajustes efectuados en el laboratorio de Biología Molecular de la Universidad del valle, los cuales se describen a continuación:
Digestión. En una solución que contiene 5μl de tampón 5X, 2 μl de EcoRI/MseI y 15.5 μl de agua destilada, se hizo la digestión del ADN almacenado en los discos purificados durante 2 horas a 37 ºC seguido de 15 min a 70 ºC, utilizando un termociclador Perkin Elmer 9700.
Ligación de adaptadores. Terminado el tiempo de digestión se adicionó a cada tubo de PCR 25 µl de la solución de ligación de los adaptadores la cual contiene 24 μl de adaptadores y 1 μl de T4 DNA ligasa, se mezcló suavemente y se incubó a 20ºC por dos horas.
Preamplificación (AFLP+1). Para la preamplificación de las muestras se extrajeron los componentes utilizados en la digestión y ligación de adaptadores, dejando los discos en el tubo. A cada uno de los tubos se les adicionó 40 µl de pre-amp premix, 5 μl tampón 10X plus Mg y 1 μl de Taq polimerasa como lo indica la casa fabricante. El programa de amplificación utilizado para el AFLP+1 se realizó bajo las siguientes condiciones: 20 ciclos, cada uno de 94ºC por 30 seg; 56ºC por 60 seg y 72ºC por 60 seg.
Finalizados los 20 ciclos, las muestras se diluyeron 1:50 en tampón TE, utilizando 1µl de muestra en 49 µl de tampón TE, cantidad suficiente para 10 análisis AFLP+3, y la amplificación de los fragmentos se visualizó en un gel de agarosa al 1.5%.
Para la amplificación selectiva se utilizaron cinco combinaciones de cebadores, tres reportadas por Dragoo et al (3) para la familia Muridae (Rodentia) así: combinación 1, EcoRI-ACG/ MseI-CAT; combinación 2, EcoRI-ACG/ MseI-CAG; combinación 3, EcoRI-ACG/ MseI-CTA y dos reportadas por la casa fabricante (7); combinación 4, EcoRI-AAG/ MseI-CAC; combinación 5, EcoRI-ACA/ MseI-CTG, como combinaciones que son altamente reproducibles en el laboratorio para la estimación de la variabilidad genética en el genoma de roedores y plantas.
Amplificación selectiva (AFLP+3). Dos soluciones premix fueron preparadas para la amplificación selectiva, la primera muestra contenía 0.5 μl de cebador EcoRI y 4.5 μl de cebador MseI y se aplicó a cada par de cebadores. El premix 2 contenía 7.9 μl de agua destilada, 2 μl de tampón 10X plus Mg y 0.1 μl de Taq polimerasa por muestra.
A cada tubo se le adicionaron 5 μl de muestra diliuda, 5 μl del premix 1 y 10 μl del premix 2. Para la amplificación selectiva AFLP+3 se inició con un ciclo a 94 ºC por 30 seg; luego a 65 ºC por 30 seg y 72ºC por 60 seg, seguido de 12 ciclos donde la temperatura de apareamiento de los cebadores descendió 0.7ºC por cada ciclo y 23 ciclos finales a 94ºC por 30 seg, 56 ºC por 30 seg y 72ºC por 60 seg.
La detección de los fragmentos amplificados de longitud variable de Cavia porcellus se realizó en gel denaturante de poliacrilamida al 6% y 3 M de úrea, utilizando una cámara vertical de electroforesis (Sequi-Gen GT Sequencing Cell) de BIO-RAD y tinción con nitrato de plata.
Resultados
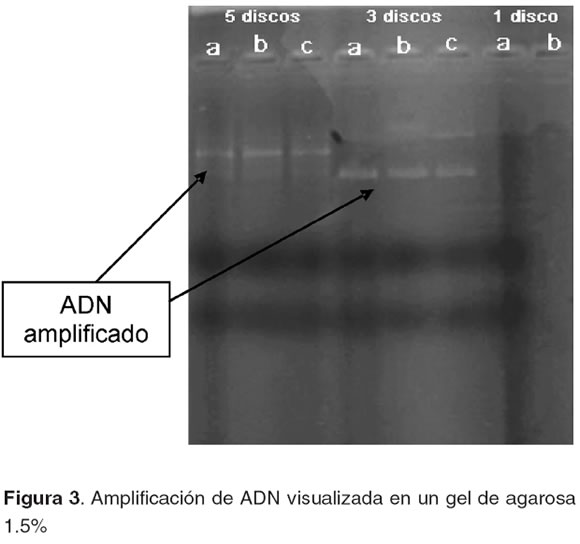
Los mejores productos de amplificación, para visualizar y llevar a cabo análisis de polimorfismos con marcadores AFLP, se obtuvieron a partir de muestras con tres discos de FTA® por individuo, (véase Figura 3) lo que sugiere que aproximadamente 75 ng de ADN por individuo, extraído con está metodología, son suficientes para obtener bandas polimórficas de longitud variable en el genoma del C. porcellus.
Discusión
En Cavia porcellus, como ya se mencionó, no existen referencias sobre análisis moleculares realizados en Colombia, por lo tanto para profundizar en esta área se requiere llevar a cabo un trabajo sistemático de investigación, que parta desde la estandarización de los procesos de colección de muestras de sangre, amplificación del ADN y visualización de fragmentos de dicha molécula, que conduzcan a realizar los análisis correspondientes. Este estudio permite concluir que las tarjetas FTA ofrecen varias ventajas para los análisis de muestras de ADN extraído de la sangre del cuy, principalmente en cuanto a facilidad de transporte y almacenamiento de las muestras.
Al utilizar tarjetas FTA, es necesario purificar los discos con el reagente y no se requiere realizar la cuantificación del ADN. Este hecho obligó a determinar el número de discos necesarios para obtener el producto amplificado de la mejor calidad, aspecto que cobra mayor importancia cuando se estudian especies, sin técnicas previamente estandarizadas y que presenten dificultades en cuanto a la toma y conservación de las muestras de sangre.
Los resultados obtenidos permiten recomendar la utilización de tres discos de FTA por individuo, para analizar el ADN del cuy, ya que de este modo se obtuvo un producto amplificado con la calidad necesaria para visualizar fragmentos de ADN. Con un disco sólo fue posible comprobar la amplificación, pero no se obtuvieron resultados de buena calidad, luego de efectuar la amplificación selectiva y los productos finales, obtenidos con cinco y tres discos, no presentaron diferencias en cuanto a la calidad de las bandas, por lo tanto, al utilizar cinco discos se incrementan los costos por la utilización de mayores cantidades de reactivos, especialmente en los procesos de purificación.
Por otra parte, la calidad de los productos amplificados podría atribuirse, en gran medida, a la seguridad que ofrecen las tarjetas FTA, puesto que evitan la contaminación del ADN de las muestras con ADN de otra procedencia o el contacto con componentes que los desnaturalizan.
Otra de las ventajas de las tarjetas FTA, observada con la realización de este estudio, es que se pueden almacenar discos previamente purificados a -20 ºC hasta por un periodo de 30 días, sin que se vea afectada su calidad para realizar los procedimientos de amplificación.
Todo lo anterior permite recomendar el uso de las tarjetas de FTA® para el estudio genético de poblaciones de Cavia porcellus, con el marcador AFLP´s y los procedimientos descritos en el presente artículo. De igual manera, se abre la posibilidad de hacer uso de las tarjetas de FTA® para el estudio de otros marcadores moleculares en la misma especie y en otras en las que resulte la colección de muestras de sangre con el volumen requerido en los análisis tradicionales.
Agradecimientos
A la vicerrectoría de Investigaciones, Posgrados y Relaciones Internacionales de la Universidad de Nariño. Al Departamento de Biología, Sección Genética de la Universidad del Valle.
Summary
Amplified Fragments Length Polymorphisms (AFLP´s) from blood samples stored in FTA® cards for Cavia porcellus.
A methodology that includes the use of FTA® (Whatman Bioscience, Cambridge) to collect and store animals` blood samples and the procedures to extract and to get AFLP markers is presented in this paper. A review of the literature indicates that there are no reports concerning both aspects for the Cavia porcellus case. To reach our goal blood samples of three populations - Two native ones and other genetically improved- were obtained through heart puncture. This blood was stored in the FTA cards in order to extract, purify, amplify and analyze their DNA forms. All of the animals came from “Botana” farm of the Universidad de Nariño, located in Pasto, Colombia. For amplifying the AFLP one, three and five 1.2 mm FTA disks of approximately 75 ng of DNA per disk where used. The tests indicated that the best products to amplify and to visualize the AFLP where those ones obtained from samples of three FTA disks per animal. This suggests that 75 ng of DNA per animal is enough to generate AFLP of high quality in the Cavia porcellus` genome. We recommend the use of FTA cards to carry out genetic analyses in the Cavia porcellus, including the methodology modifications presented in this paper.
Key words: AFLP´s, Cavia porcellus, FTA.
Referencias
1. Chauca L. Producción de cuyes Cavia porcellus. FAO INIA 1997; [20 Septiembre 2005] URL: http://www.fao.org. [ Links ]
2. Dobbs L, Madigan M, Carter A, Earls L. Use of FTA gene guard filter paper for the storage and transportation of tumor cells for molecular testing. Arch Pathol Lab Med 2002; 126: 56-63[ [ Links ]STANDARDIZEDENDPARAG]
3. Dragoo JW, Salazar-Bravo J, Layne LJ, Yates TL. Relationships within the Calomys callosus species group based on amplified fragment length polymorphisms. Bioch Sys and Ecol 2003; 31: 703-713. [ Links ]
4. Ferreira ME, Grattapaglia D. Introducción al uso de marcadores moleculares en el análisis genético. 1 ed. brasilia: embrapa-cenargen; 1998. [ Links ]
5. Florian AA. Mermas de producción por infestaciones de Dermanysus gallinae. informe final proyecto sistemas de producción de cuyes en el Perú, fase I y II. Lima: Inia; 1995. [ Links ]
6. Instituto Nacional de Estudios Ambientales (IDEAM). Reporte de climatología de la estación Botana. Pasto-Colombia: El instituto; 1999. [ Links ]
7. Invitrogen. Instruction manual AFLP Analysis System I, Starter Primer Kit version B 2003; URL http://www.invitrogen.com/content/sfs/manuals/aflpI.pdf [ Links ]
8. Leguia, PG. Mermas de producción debido a enfermedades parasitarias. informe final proyecto sistemas de producción de cuyes en el perú, fase I y II. Lima: inia; 1995. [ Links ]
9. Schifferli C, Villaroel M, Arruga M. Obtención de DNA para el estudio de BLAD en toros de Argentina y España. Arch Zoot 2000; 49:188; 505-508. [ Links ]
10. Sitaraman K, Darfler M, Westfall B. amplification of large DNA from blood stored at room temperature. Focus 1999; 21:1-10. [ Links ]
11. Smith L, Burgoyne L. Collecting, archiving and processing DNA from wildlife samples using FTA® databasing paper. BMC Ecol 2004; 4:4-11. [ Links ]
12. Solarte C, Soto F, Pérez T. Modelo animal multicarácter para la estimación de parámetros genéticos del Cavia porcellus en Colombia. Rev Cuba Cien Agrí. 2002; 36:19-24. [ Links ]
13. Subrungruang I, Mungthin M, Chavalitshewinkoon-petmitr P, Rangsin R, Naaglor T, et al. Evaluation of DNA extraction and PCR methods for detection of Enterocytozoon bienuesi in stool specimens. J Clin Microbiol 2004; 42:3490-3494. [ Links ]
14. Vos P, Hogers R, Bleeker M, Reijans M, Van de Lee T, et al. AFLP: a new technique for DNA fingerprinting. Nucl Acid Res 1995;23:4407-4414. [ Links ]
15. Whatman. FTA® protocols: collect transport, archive and access nucleic acids all at room temperature 2002; http:// www.cosmobio.com.ar/docs/fta%20protocols.pdf [ Links ]
16. Whatman. Room temperature sample collection, storage and purification of nucleic acids 2003; URL http:// www.touchbriefings.com/pdf/16/fdd031_t_whatman.pdf [ Links ]
Polimorfismos en la longitud de fragmentos amplificados (AFLP´s) a partir de muestras de sangre almacenadas en tarjetas FTA® para la especie Cavia porcellus Lin. (Rodentia: Caviidae).
William Burgos-Paz1, Zoot; Carol Rosero-Galindo2, Biol, MSc; Heiber Cárdenas-Henao2, Biol, MSc; Carlos Solarte-Portilla 1, Zoot, PhD.
1Grupo Producción y Sanidad Animal, Facultad de Ciencias Pecuarias, Programa de Zootecnia, Universidad de Nariño, Ciudad Universitaria Torobajo, A.A.1175, Pasto, Colombia; 2Laboratorio de Biología Molecular, Departamento de Biología, Facultad de Ciencias, Universidad del Valle, A. A. 25360, Cali, Colombia.
Williamobur@yahoo.com
(Recibido: 28 febrero, 2006; aceptado: 8 febrero, 2007)
Resumen
Una manera eficaz de establecer el grado de variabilidad entre y dentro de poblaciones, es a través del análisis de polimorfismos de ADN con marcadores moleculares como los AFLP`s. En este artículo se presenta una metodología que combina la utilización de tarjetas de FTA® (Whatman Bioscience, Cambridge) para colección y conservación de muestras de sangre, con los procedimientos de extracción de ADN y obtención de marcadores AFLP´s, aspectos sobre los cuales no existen antecedentes para la especie Cavia porcellus. Se utilizaron muestras de ADN procedentes de tres poblaciones, dos criollas y una mejorada genéticamente obtenida a partir de un pie de cría procedente del Perú y sometida a selección en Colombia durante varias generaciones. Todos los animales procedieron de la Granja “Botana”, propiedad de la Universidad de Nariño, Pasto-Colombia. Para la detección de polimorfismos en la longitud de los fragmentos (AFLP`s) se utilizaron uno, tres y cinco discos FTA® de 1.2 mm, cada disco con aproximadamente 25 ng de ADN. Los ensayos indicaron que los mejores productos de amplificación, para la visualización de AFLP´s, se obtuvieron de muestras con tres discos de FTA® por individuo, lo que sugiere que con esta metodología, 75 ng de ADN por animal son suficientes para detectar polimorfismos de alta calidad en el genoma de Cavia porcellus. Se recomienda el uso de las tarjetas de FTA® para el estudio genético de poblaciones de Cavia porcellus, con las modificaciones metodológicas descritas en este artículo para marcadores AFLP´s.
Palabras Clave: AFLP´s, Cavia porcellus, FTA.
Introducción
Para analizar genéticamente a un individuo se requiere inicialmente el aislamiento de su ADN. Las exigencias en la calidad y cantidad de ADN aislado varían de acuerdo con el marcador genético que seleccione el investigador. Sin embargo, pese al gran número de protocolos existentes para la extracción de ADN, no todos tienen el mismo grado de reproducibilidad en los laboratorios por cuanto los métodos de conservación de la muestra tales como sangre u otros tejidos (11) difieren en el control de la acción de las DNAasas después de la toma de la muestra.
En la mayoría de los vertebrados, el tejido comúnmente escogido para la obtención de ADN de alta calidad es la sangre (11). Sin embargo, en los cuyes dadas las características de su hemograma (5, 8), la coagulación de la sangre ocurre de manera casi instantánea después de la toma de la muestra, lo que dificulta obtener una muestra óptima para llevar a cabo los análisis del ADN.
Las tarjetas FTA® (Whatman Bioscience, Cambridge, United Kingdom) están diseñadas para la colección, almacenaje y posterior purificación del ADN. Dichas tarjetas están constituidas de un papel filtro impregnado con una fórmula química patentada por la casa fabricante. La fórmula está diseñada para producir lisis de las membranas celulares y desnaturalización de las proteínas y de las enzimas con capacidad para desnaturalizar los ácidos nucleicos, los cuales quedan atrapados en una matriz tan pronto entran en contacto con el papel, con lo cual se reduce el riesgo de contaminación (15). Los componentes restantes de la muestra son eliminados mediante el lavado con un reactivo, producido por la misma casa fabricante.
Las principales ventajas del uso de estas tarjetas, frente a métodos convencionales de aislamiento de ADN son las siguientes:
1. Facilidad para la colección y transporte de las muestras, ya que en el caso de sangre se requiere una o dos gotas (13).
2. Almacenamiento de gran cantidad de muestras en un menor espacio (9).
3. Conservación de la muestra a temperatura ambiente por largos períodos (2, 10).
4. Alta sensibilidad en la detección de polimorfismos de ADN vía PCR, dado que el ADN que contiene la tarjeta está libre de compuestos como proteínas, DNAsas y sustancias tampón utilizadas en los procesos convencionales de extracción y que pueden afectar la calidad del ADN (13).
5. Rápida purificación de la muestra (16).
Aunque no es posible cuantificar el ADN aislado en la tarjeta de celulosa, se estima que un disco de 1.2 mm puede contener 25 ng de ADN (15).
La técnica AFLP (del inglés Amplified Fragment Length Polymorphisms) combina la aplicación de enzimas de restricción con el poder de la PCR a nivel de todo el genoma del individuo (4). Con esta técnica se utilizan entre 100 y 250 ng de ADN de alta pureza para generar un gran número de bandas sin previo conocimiento del genoma (14).
En el presente estudio, se evaluó el uso de las tarjetas FTA® para la colección, almacenamiento y transporte de las muestras de sangre de Cavia porcellus y además se estandarizó el protocolo para detectar polimorfismos mediante el uso de los marcadores moleculares AFLP´s.
Materiales y métodos
Muestras
Los ejemplares utilizados para este trabajo procedían de la Granja “Botana”, propiedad de la Universidad de Nariño, situada a nueve kilómetros al sur de la ciudad de Pasto, en la República de Colombia a una altura de 2820 m.s.n.m, una temperatura promedio anual de 12.7οC, 967 mm de precipitación media anual y una humedad relativa del 73% (6).
En los análisis se incluyeron ejemplares de tres poblaciones, dos “criollas” y una mejorada genéticamente, conocida como “peruana”, para lo cual se tomaron muestras de sangre de 12 individuos por población.
Colección y almacenamiento de las muestras de sangre en tarjetas FTA®
Las muestras de sangre se obtuvieron mediante punción cardíaca (véase Figura 1). La cantidad extraída varió entre 0.5 y 2.5 ml por animal. Dos gotas de sangre fueron aplicadas directamente a las tarjetas FTA® debidamente marcadas, las cuáles, según lo indicado por la casa fabricante (15), se dejaron secar a temperatura ambiente durante una hora y posteriormente se marcaron, envolvieron en papel aluminio y se enviaron al Laboratorio de Biología Molecular de la Universidad del Valle en la Ciudad de Cali para realizar los procedimientos de extracción de ADN y obtención de los marcadores AFLP´s (véase Figura 2).


Purificación de las muestras para la obtención de ADN
De cada muestra de sangre almacenada en la tarjeta se extrajeron uno, tres y cinco discos FTA® de 1.2 mm, usando un sacabocados, y se colocaron en un tubo Eppendorf.
Para la purificación de los discos de ADN se adicionaron 200 µl de “FTA Purification Reagent” por disco a cada tubo y se incubó por cinco minutos agitando por inversión. Posteriormente, sin remover los discos del tubo, se extrajo y descartó el Reagente; este mismo procedimiento se repitió tres veces (15).
Posteriormente se adicionó en cada tubo, 200 µl de tampón TE (10 mM Tris-HCl, 0.1 mM EDTA, pH 8.0) por disco y nuevamente se incubó por cinco minutos, agitando por inversión. Se descartó el tampón TE sin remover los discos del tubo y se repitió el proceso dos veces. Una vez descartado el tampón TE se dejaron secar los discos a temperatura ambiente durante una hora (15).
Amplificación de fragmentos polimorficos a partir de las muestras de ADN almacenadas en los discos FTA®
La amplificación del ADN se realizó con el kit, AFLP® Analysis System I de Invitrogen, siguiendo el protocolo de Vos et al (14), las recomendaciones de la casa fabricante (7) y los ajustes efectuados en el laboratorio de Biología Molecular de la Universidad del valle, los cuales se describen a continuación:
Digestión. En una solución que contiene 5μl de tampón 5X, 2 μl de EcoRI/MseI y 15.5 μl de agua destilada, se hizo la digestión del ADN almacenado en los discos purificados durante 2 horas a 37 ºC seguido de 15 min a 70 ºC, utilizando un termociclador Perkin Elmer 9700.
Ligación de adaptadores. Terminado el tiempo de digestión se adicionó a cada tubo de PCR 25 µl de la solución de ligación de los adaptadores la cual contiene 24 μl de adaptadores y 1 μl de T4 DNA ligasa, se mezcló suavemente y se incubó a 20ºC por dos horas.
Preamplificación (AFLP+1). Para la preamplificación de las muestras se extrajeron los componentes utilizados en la digestión y ligación de adaptadores, dejando los discos en el tubo. A cada uno de los tubos se les adicionó 40 µl de pre-amp premix, 5 μl tampón 10X plus Mg y 1 μl de Taq polimerasa como lo indica la casa fabricante. El programa de amplificación utilizado para el AFLP+1 se realizó bajo las siguientes condiciones: 20 ciclos, cada uno de 94ºC por 30 seg; 56ºC por 60 seg y 72ºC por 60 seg.
Finalizados los 20 ciclos, las muestras se diluyeron 1:50 en tampón TE, utilizando 1µl de muestra en 49 µl de tampón TE, cantidad suficiente para 10 análisis AFLP+3, y la amplificación de los fragmentos se visualizó en un gel de agarosa al 1.5%.
Para la amplificación selectiva se utilizaron cinco combinaciones de cebadores, tres reportadas por Dragoo et al (3) para la familia Muridae (Rodentia) así: combinación 1, EcoRI-ACG/ MseI-CAT; combinación 2, EcoRI-ACG/ MseI-CAG; combinación 3, EcoRI-ACG/ MseI-CTA y dos reportadas por la casa fabricante (7); combinación 4, EcoRI-AAG/ MseI-CAC; combinación 5, EcoRI-ACA/ MseI-CTG, como combinaciones que son altamente reproducibles en el laboratorio para la estimación de la variabilidad genética en el genoma de roedores y plantas.
Amplificación selectiva (AFLP+3). Dos soluciones premix fueron preparadas para la amplificación selectiva, la primera muestra contenía 0.5 μl de cebador EcoRI y 4.5 μl de cebador MseI y se aplicó a cada par de cebadores. El premix 2 contenía 7.9 μl de agua destilada, 2 μl de tampón 10X plus Mg y 0.1 μl de Taq polimerasa por muestra.
A cada tubo se le adicionaron 5 μl de muestra diliuda, 5 μl del premix 1 y 10 μl del premix 2. Para la amplificación selectiva AFLP+3 se inició con un ciclo a 94 ºC por 30 seg; luego a 65 ºC por 30 seg y 72ºC por 60 seg, seguido de 12 ciclos donde la temperatura de apareamiento de los cebadores descendió 0.7ºC por cada ciclo y 23 ciclos finales a 94ºC por 30 seg, 56 ºC por 30 seg y 72ºC por 60 seg.
La detección de los fragmentos amplificados de longitud variable de Cavia porcellus se realizó en gel denaturante de poliacrilamida al 6% y 3 M de úrea, utilizando una cámara vertical de electroforesis (Sequi-Gen GT Sequencing Cell) de BIO-RAD y tinción con nitrato de plata.
Resultados
Los mejores productos de amplificación, para visualizar y llevar a cabo análisis de polimorfismos con marcadores AFLP, se obtuvieron a partir de muestras con tres discos de FTA® por individuo, (véase Figura 3) lo que sugiere que aproximadamente 75 ng de ADN por individuo, extraído con está metodología, son suficientes para obtener bandas polimórficas de longitud variable en el genoma del C. porcellus.

Discusión
En Cavia porcellus, como ya se mencionó, no existen referencias sobre análisis moleculares realizados en Colombia, por lo tanto para profundizar en esta área se requiere llevar a cabo un trabajo sistemático de investigación, que parta desde la estandarización de los procesos de colección de muestras de sangre, amplificación del ADN y visualización de fragmentos de dicha molécula, que conduzcan a realizar los análisis correspondientes. Este estudio permite concluir que las tarjetas FTA ofrecen varias ventajas para los análisis de muestras de ADN extraído de la sangre del cuy, principalmente en cuanto a facilidad de transporte y almacenamiento de las muestras.
Al utilizar tarjetas FTA, es necesario purificar los discos con el reagente y no se requiere realizar la cuantificación del ADN. Este hecho obligó a determinar el número de discos necesarios para obtener el producto amplificado de la mejor calidad, aspecto que cobra mayor importancia cuando se estudian especies, sin técnicas previamente estandarizadas y que presenten dificultades en cuanto a la toma y conservación de las muestras de sangre.
Los resultados obtenidos permiten recomendar la utilización de tres discos de FTA por individuo, para analizar el ADN del cuy, ya que de este modo se obtuvo un producto amplificado con la calidad necesaria para visualizar fragmentos de ADN. Con un disco sólo fue posible comprobar la amplificación, pero no se obtuvieron resultados de buena calidad, luego de efectuar la amplificación selectiva y los productos finales, obtenidos con cinco y tres discos, no presentaron diferencias en cuanto a la calidad de las bandas, por lo tanto, al utilizar cinco discos se incrementan los costos por la utilización de mayores cantidades de reactivos, especialmente en los procesos de purificación.
Por otra parte, la calidad de los productos amplificados podría atribuirse, en gran medida, a la seguridad que ofrecen las tarjetas FTA, puesto que evitan la contaminación del ADN de las muestras con ADN de otra procedencia o el contacto con componentes que los desnaturalizan.
Otra de las ventajas de las tarjetas FTA, observada con la realización de este estudio, es que se pueden almacenar discos previamente purificados a -20 ºC hasta por un periodo de 30 días, sin que se vea afectada su calidad para realizar los procedimientos de amplificación.
Todo lo anterior permite recomendar el uso de las tarjetas de FTA® para el estudio genético de poblaciones de Cavia porcellus, con el marcador AFLP´s y los procedimientos descritos en el presente artículo. De igual manera, se abre la posibilidad de hacer uso de las tarjetas de FTA® para el estudio de otros marcadores moleculares en la misma especie y en otras en las que resulte la colección de muestras de sangre con el volumen requerido en los análisis tradicionales.
Agradecimientos
A la vicerrectoría de Investigaciones, Posgrados y Relaciones Internacionales de la Universidad de Nariño. Al Departamento de Biología, Sección Genética de la Universidad del Valle.
Summary
Amplified Fragments Length Polymorphisms (AFLP´s) from blood samples stored in FTA® cards for Cavia porcellus.
A methodology that includes the use of FTA® (Whatman Bioscience, Cambridge) to collect and store animals` blood samples and the procedures to extract and to get AFLP markers is presented in this paper. A review of the literature indicates that there are no reports concerning both aspects for the Cavia porcellus case. To reach our goal blood samples of three populations - Two native ones and other genetically improved- were obtained through heart puncture. This blood was stored in the FTA cards in order to extract, purify, amplify and analyze their DNA forms. All of the animals came from “Botana” farm of the Universidad de Nariño, located in Pasto, Colombia. For amplifying the AFLP one, three and five 1.2 mm FTA disks of approximately 75 ng of DNA per disk where used. The tests indicated that the best products to amplify and to visualize the AFLP where those ones obtained from samples of three FTA disks per animal. This suggests that 75 ng of DNA per animal is enough to generate AFLP of high quality in the Cavia porcellus` genome. We recommend the use of FTA cards to carry out genetic analyses in the Cavia porcellus, including the methodology modifications presented in this paper.
Key words: AFLP´s, Cavia porcellus, FTA.
Referencias
1. Chauca L. Producción de cuyes Cavia porcellus. FAO INIA 1997; [20 Septiembre 2005] URL: http://www.fao.org. [ Links ]
2. Dobbs L, Madigan M, Carter A, Earls L. Use of FTA gene guard filter paper for the storage and transportation of tumor cells for molecular testing. Arch Pathol Lab Med 2002; 126: 56-63[ [ Links ]STANDARDIZEDENDPARAG]
3. Dragoo JW, Salazar-Bravo J, Layne LJ, Yates TL. Relationships within the Calomys callosus species group based on amplified fragment length polymorphisms. Bioch Sys and Ecol 2003; 31: 703-713. [ Links ]
4. Ferreira ME, Grattapaglia D. Introducción al uso de marcadores moleculares en el análisis genético. 1 ed. brasilia: embrapa-cenargen; 1998. [ Links ]
5. Florian AA. Mermas de producción por infestaciones de Dermanysus gallinae. informe final proyecto sistemas de producción de cuyes en el Perú, fase I y II. Lima: Inia; 1995. [ Links ]
6. Instituto Nacional de Estudios Ambientales (IDEAM). Reporte de climatología de la estación Botana. Pasto-Colombia: El instituto; 1999. [ Links ]
7. Invitrogen. Instruction manual AFLP Analysis System I, Starter Primer Kit version B 2003; URL http://www.invitrogen.com/content/sfs/manuals/aflpI.pdf [ Links ]
8. Leguia, PG. Mermas de producción debido a enfermedades parasitarias. informe final proyecto sistemas de producción de cuyes en el perú, fase I y II. Lima: inia; 1995. [ Links ]
9. Schifferli C, Villaroel M, Arruga M. Obtención de DNA para el estudio de BLAD en toros de Argentina y España. Arch Zoot 2000; 49:188; 505-508. [ Links ]
10. Sitaraman K, Darfler M, Westfall B. amplification of large DNA from blood stored at room temperature. Focus 1999; 21:1-10. [ Links ]
11. Smith L, Burgoyne L. Collecting, archiving and processing DNA from wildlife samples using FTA® databasing paper. BMC Ecol 2004; 4:4-11. [ Links ]
12. Solarte C, Soto F, Pérez T. Modelo animal multicarácter para la estimación de parámetros genéticos del Cavia porcellus en Colombia. Rev Cuba Cien Agrí. 2002; 36:19-24. [ Links ]
13. Subrungruang I, Mungthin M, Chavalitshewinkoon-petmitr P, Rangsin R, Naaglor T, et al. Evaluation of DNA extraction and PCR methods for detection of Enterocytozoon bienuesi in stool specimens. J Clin Microbiol 2004; 42:3490-3494. [ Links ]
14. Vos P, Hogers R, Bleeker M, Reijans M, Van de Lee T, et al. AFLP: a new technique for DNA fingerprinting. Nucl Acid Res 1995;23:4407-4414. [ Links ]
15. Whatman. FTA® protocols: collect transport, archive and access nucleic acids all at room temperature 2002; http:// www.cosmobio.com.ar/docs/fta%20protocols.pdf [ Links ]
16. Whatman. Room temperature sample collection, storage and purification of nucleic acids 2003; URL http:// www.touchbriefings.com/pdf/16/fdd031_t_whatman.pdf [ Links ]














