Services on Demand
Journal
Article
Indicators
-
 Cited by SciELO
Cited by SciELO -
 Access statistics
Access statistics
Related links
-
 Cited by Google
Cited by Google -
 Similars in
SciELO
Similars in
SciELO -
 Similars in Google
Similars in Google
Share
Biomédica
Print version ISSN 0120-4157On-line version ISSN 2590-7379
Biomédica vol.24 suppl.1 Bogotá June 2004
Inactivación de genes de Mycobacterium tuberculosis y
su potencial utilidad en la prevención y el control de la tuberculosis
Ofelia Chacón 1,2 , Teresa Realpe 1 , Raúl Gerardo Barletta 2 , Jaime Robledo 1,2,3
1 Unidad de Bacteriología y Micobacterias, Corporación para Investigaciones Biológicas, Medellín, Colombia.
2
Department of Veterinary and Biomedical Sciences, University of Nebraska, Lincoln, Nebraska, USA.3
Facultad de Medicina, Universidad Pontificia BolivarianaEl conocimiento derivado del genoma de Mycobacterium tuberculosis, junto con el desarrollo de sofisticados sistemas para la manipulación genética del bacilo, ofrece la mayor promesa para el desarrollo de herramientas nuevas y más eficientes para prevenir y controlar la tuberculosis. Se han desarrollado métodos más eficientes para la inactivación de genes micobacterianos que se han convertido en el pilar de la genómica funcional micobacteriana. La generación de mutantes mediante la inactivación génica, apoyada directa o indirectamente por el desciframiento del genoma micobacteriano, ha permitido la generación de un número significativo de mutantes de M. tuberculosis. En algunos casos, el análisis de estas mutantes ha establecido relaciones entre los productos génicos y sus funciones en la fisiología y la patogenicidad de la micobacteria. En esta revisión se describen los estudios más representativos basados en dichas mutantes.
Palabras claves: Mycobacterium tuberculosis, inactivación de genes, genómica funcional, mutantes atenuadas.
Gene inactivation in Mycobacterium tuberculosis and its use in tuberculosis control and prevention
Availability of the M. tuberculosis genome sequence and the development of sophisticated systems for genetic manipulation of bacilli offer the potential for new and effective tools to prevent and control tuberculosis. Efficient methods to inactivate mycobacterial genes have been developed. These methods have become the cornerstone for the application and development of mycobacterial functional genomics. Specific mutants are generated to establish the role of targetted genes associated with mycobacterial physiology and pathogenesis. Gene inactivation, supported directly or indirectly by the deciphering of the mycobacterial genome, has permitted the generation of large numbers of M. tuberculosis mutants. Analysis of these mutants has (in some cases) established relationships between gene products and their role in mycobacterial physiology and pathogenesis.
Key words: Mycobacterium tuberculosis, gene inactivation, functional genomic, attenuated mutants.
La identificación de Mycobacterium tuberculosis como agente causal de tuberculosis hecha por Robert Koch en 1882 marcó el primer hito en el estudio de esta enfermedad (1). Estos hallazgos, seguidos por el desarrollo y refinamiento de las técnicas de coloración y cultivo realizadas por Paul Erhlich, Franz Ziehl y Friedrich K.A. Nielsen proporcionaron las primeras herramientas para combatir racionalmente la tuberculosis (2). En más de un siglo desde la introducción de estas herramientas, se han desarrollado una vacuna y varios agentes quimioterapéuticos. Aun así, en la actualidad, la tuberculosis sigue siendo la mayor causa de muerte en el mundo debida a un único agente infeccioso (3,4).
Se sabe que casi un tercio de la población mundial está infectada con el bacilo tuberculoso, el cual es la causa de muerte de 2 millones de personas, aproximadamente, cada año. Esta situación ha empeorado recientemente por factores como la aparición de cepas de bacilos multirresistentes a los medicamentos antes efectivos en el tratamiento y por la pandemia VIH/sida. De acuerdo con este panorama, se necesitan nuevas estrategias para prevenir y controlar eficiente-mente esta enfermedad.
Las características propias de M. tuberculosis plantean un verdadero reto para los investigadores. Por ejemplo, M. tuberculosis tiene un tiempo de replicación lento, sus requerimientos para el crecimiento in vitro son exigentes y presenta dificultades para la manipulación genética. Afortunadamente, el conocimiento reciente del genoma de la micobacteria y el desarrollo de la tecnología genómica han proporcionado instrumentos para el desarrollo de nuevas técnicas que permiten la manipulación genética del bacilo (5-8). La inactivación de genes específicos y el estudio de los efectos que dichas inactivaciones tienen en las funciones estructurales y fisiológicas de la micobacteria, incluso sus efectos en la virulencia, así como el estudio de la esencialidad de los genes que codifican posibles blancos para agentes terapéuticos, se han convertido en la piedra angular de la aplicación de la genómica funcional como disciplina para el estudio de la tuberculosis.
Actualmente, la genómica funcional está abriendo un camino totalmente nuevo en el estudio de la tuberculosis y su utilización, probablemente, llevará al diseño racional de estrategias para la identificación de blancos potenciales para la intervención terapéutica, el diagnóstico y la generación de vacunas.
En esta revisión, que no pretende ser exhaustiva por la imposibilidad de cubrir la totalidad del tema, se abordan los hallazgos recientes suministrados por la manipulación genética y la inactivación de algunos genes relacionados con la estructura o el metabolismo de M. tuberculosis. Se describen brevemente las estrategias que se han usado para inactivar diferentes genes y los posibles papeles de cada gene en la fisiología o en el proceso de patogénesis. Esperamos que esta información sea de utilidad en el entendimiento de la genética micobacteriana y que sea aplicable en el diseño y en el desarrollo de nuevas medidas de control y prevención de la tuberculosis.
Para un mejor entendimiento de la información proporcionada, se describirán inicialmente algunas de las metodologías utilizadas para la inactivación génica en micobacterias y, posteriormente, varios estudios en los cuales se han generado mutantes de M. tuberculosis con el uso de algunas de estas metodologías. Esto último se presenta clasificando los genes en: aquéllos relacionados con la biosíntesis de la pared celular - un componente estructural que es cuantitativa y cualitativamente importante para la fisiología micobacteriana - y sus proteínas asociadas; los relacionados con la biosíntesis, el metabolismo o el transporte de metabolitos y nutrientes como aminoácidos, metales y ácidos grasos, y los genes involucrados en procesos complejos que juegan papeles importantes en la adaptación micobacteriana a diferentes factores medio-ambientales como la regulación de la transducción de señales, la transcripción y la resistencia a los mecanismos bactericidas de los macrófagos.
Técnicas utilizadas para la inactivación génica de micobacterias
Las micobacterias, especialmente aquéllas de crecimiento lento como M. tuberculosis, son microorganismos difíciles de manipular genéticamente. Los métodos utilizados inicialmente para generar mutantes de micobacterias se basaron en el uso de diversos agentes mutagénicos como la luz ultravioleta y los agentes químicos (9). Sin embargo, estos métodos no fueron lo suficientemente efectivos como para permitir un desarrollo rápido de la genética funcional micobacteriana. Décadas más tarde, se aplicaron exitosamente otras técnicas como el empleo de vectores adecuados que incluían plásmidos, fagos, fásmidos y transposones que permiten la inactivación de genes por recombinación homóloga y la generación de mutaciones al azar. La inactivación de genes por recombinación homóloga consiste en la introducción de una copia mutada del gen en la micobacteria. Esta copia se recombina luego por mecanismos de recombinación homóloga con el gen sin mutación", el cual se reemplaza en el cromosoma micobacteriano (
figura 1). Mientras que algunos pocos ensayos han tenido éxito introduciendo el gen mutado como un fragmento lineal (10-13), la mayoría de los métodos, como se presenta más adelante, se basan en el uso de plásmidos y fagos.
Por ejemplo, un vector suicida, consistente en un plásmido con un gen que codifica para un producto que resulta en toxicidad o letalidad para la micobacteria, puede ser utilizado para introducir una copia inactivada del gen blanco, asegurándose que la recombinación homóloga resulte en un cromosoma recombinante en el cual el gen letal o tóxico se encuentre ausente. Si se produce una recombinación parcial con integración del plásmido en el cromosoma, la transcripción resultante del gen letal impedirá que se aíslen recombinantes (
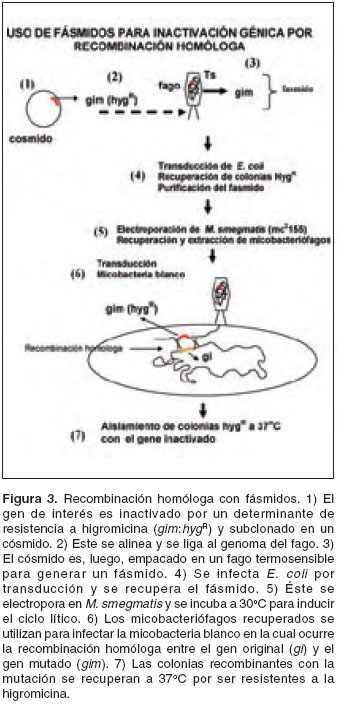
figura 2). Uno de los genes que se han utilizado con este fin es el gen sacB que codifica para levansacarasa. Cuando este gen se transcribe en presencia de sacarosa, resulta en acumulación de levanos lo cual impide el crecimiento de las micobacterias (14). Alternativamente, el gen inactivado puede ser introducido en la micobacteria por un fago. Se pueden empacar plásmidos con el gen mutado en micobacteriófagos para generar fásmidos. Estos fásmidos pueden, a su vez, infectar las micobacterias. La transfección con los fásmidos resulta en la introducción de moléculas de ADN con el gene mutado que sirven como sustrato para la recombinación homóloga (figura 3)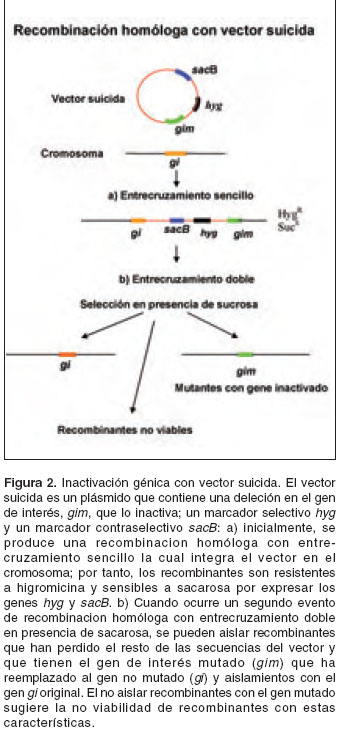
En el caso de mutación por transposones, el gen se inactiva debido a la inserción de un fragmento móvil de ADN (transposón). La inserción de estos fragmentos es al azar y, por tanto, con este sistema no es posible predecir cuál o cuáles genes serán inactivados. Al introducir los transposones por intermedio de plásmidos o de fagos se pueden generar librerías de mutantes que deben, luego, ser analizadas para buscar características específicas de las mutantes de interés (
figura 4).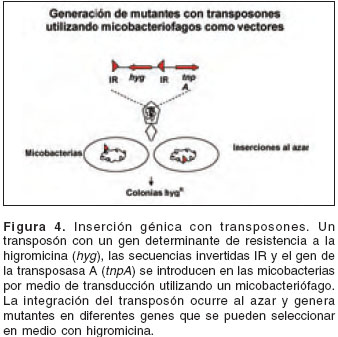
Se han utilizado algunas variaciones para hacer mas rápido y efectivo el análisis de librerías de mutantes, por ejemplo, la mutagénesis del tipo signature tag, en las cual el transposón lleva una pequeña secuencia que permite identificarlo (15,16), o hibridizaciones con el sitio del transposón (TRASH), en las cuales se utilizan microarreglos para detectar los genes que poseen inserciones (17).
Inactivación de genes involucrados en la biosíntesis de la pared celular y de proteínas asociadas
La pared celular y sus proteínas asociadas están situadas estratégicamente en la estructura externa de la micobacteria y juegan un papel importante en la interacción patógeno-hospedero. En estos microorganismos, el efecto bactericida de inhibidores de la síntesis de la pared celular confirma la esencialidad de este componente para la supervivencia de la bacteria. Además, varias proteínas asociadas con la pared celular se han propuesto como factores de virulencia (18-20).
La pared celular de la micobacteria tiene una conformación compleja de glicolípidos y proteínas asociadas con la estructura central micolilarabinogalactanopeptidoglicano (21,22). El arabinogalactano, el mayor componente de la pared celular, está unido a los residuos de ácido. murámico del peptidoglicano (23,24). Esta molécula tiene substituciones de ácidos micólicos, los cuales son ácidos grasos de alto peso molecular. En M. tuberculosis, los ácidos micólicos son ciclopropanos y se pueden clasificar de acuerdo con la presencia y la naturaleza de los sustitutos que contienen oxígeno en la porción distal de la rama del meromicolato, en ácidos alfa micólicos no oxigenados y ácidos oxigenados ceto y metoximicólicos (25-29). La síntesis de los ácidos micólicos involucra varias metiltransferasas necesarias para la formación de los anillos ciclopropanos y las ramas metiladas (25-28,30,31).
Aunque la mayoría de los ácidos micólicos en la pared celular de la micobacteria se encuentran como ésteres ligados al arabinogalactano, algunos también son transferidos por las micolil transferasas a la a-a '-trehalosa para formar glicolípidos extraíbles, incluso a-a '-trehalosa monomicolato (TMM) y a-a '- trehalosa dimicolato (TDM) o factor cordón (18,23). Otros componentes extraíbles que se han identificado en la pared celular de M. tuberculosis, incluyen iocerol dimicocerosato, sulfolípidos y lipoarabinomanam (18,23).
Se han sugerido papeles importantes para los componentes individuales de la pared celular micobacteriana en la patogenicidad y la resistencia a medicamentos del bacilo tuberculoso. Por ejemplo, las alteraciones en las proporciones y en las estructuras de los ácidos micólicos se han relacionado con variaciones en la fluidez y la permeabilidad de la pared celular. Estas variaciones pueden, a su vez, estar relacionadas con la resistencia de las micobacterias a sustancias bactericidas (32). Algunas de las funciones sugeridas para los componentes de la pared celular micobacteriana se han confirmado por análisis genético recientes en M. tuberculosis (16,28-30,33,34).
Genes involucrados en la síntesis y transporte de dimicocerosatos
Los dimicoserosatos (DIM), inclusive el dimicoserosato tiocerol (PDIM), parecen jugar un papel importante en la virulencia de la micobacteria (35). La síntesis de DIM involucra varios genes que incluyen los genes ppsA-E que codifican para una policétido sintasa requerida para la síntesis de phtiocerol (36), el gen mas que codifica para una sintasa del ácido micocerósico (37,38) y el gen fadD28 que codifica para una acil-coA sintasa, la cual parece jugar un papel importante en la transferencia acil del ácido micocerósico a phtiocerol (39).
En M. tuberculosis, estos genes están localizados en una región de 50 kb del cromosoma en el que también se localizan los genes drrA-C que codifican para un polipéptido similar al del ligador del transportador ABC (40), el gen mmpL7 que codifica para una proteína conservada de membrana de micobacterias (8), el gen fadD26 que codifica para una acil-coA sintasa (39,41) y el gen epapA5 que codifica para una policétido sintasa asociada con proteínas de función desconocida (8).
En un aislamiento clínico de M. tuberculosis, cepa Mt103, utilizando mutagénesis con transposones, se generaron mutantes de los genes fadD26, fadD28, mmpL7 y drrC. Estos mutantes se aislaron luego de una selección por su atenuación potencial en pulmones de ratones BALB/c infectados por vía intravenosa (15). Utilizando la misma estrategia se han generado mutantes de la región promotora en la cepa Erdman de M. tuberculosis que incluyen los genes faD26, ppsA-E y los genes fadD28 y mmpL7. Estos últimos se aislaron luego de seleccionarse por su inhabilidad para replicarse en el interior de los pulmones de ratones C57BL/6 infectados por vía intravenosa (16).
El análisis de los lípidos de células enteras extraídos de la mutante que contiene inserciones en la región que incluye los genes fadD26 y ppsA-E, revelaron que tenían cantidades pequeñas o indetectables de PDIM (15,16). Lo anterior sugiere que la expresión de los genes ppsA-E podría estar acoplada a la expresión del gene fadD26 situado inmediatamente corriente arriba (15,36). Futuras investigaciones deberán dirigirse a desentrañar el papel individual de las proteínas PpsA-E y FaD26 en la virulencia de M. tuberculosis.
Estudios adicionales de las mutantes fadD26- de la cepa Mt103 han demostrado que, en ausencia de DIM, se observa una susceptibilidad aumentada a los detergentes como el SDS (sodio dodecil sulfato), aunque no se afecta la susceptibilidad a reactivos intermediarios del nitrógeno (NRI) ni a antibióticos hidrofóbicos o hidrofílicos (40). Estos últimos resultados sugieren que la atenuación observada, probablemente, es el resultado de la susceptibilidad a los mecanismos de muerte en macrófagos diferentes al NRI.
Los análisis de mutantes también han revelado un papel importante de MmpL7 en el transporte de DIM. El estudio de los lípidos de células enteras, extraídos de las mutantes mmpL7- de la cepa Erdman de M. tuberculosis, mostró una acumulación significativa de PDIM maduro (16). Adicionalmente, el análisis de la distribución extracelular del DIM en las mutantes mmpL7- y drrC- del aislamiento clínico Mt103 mostró que sus productos son esenciales para la translocación del DIM (40).
Además de lo anterior, Cox y colaboradores han mostrado en mutantes ppsA-E-, mmpL7- y fadD28-generados en la cepa Erdman de M. tuberculosis, una incapacidad para crecer en pulmones de ratones y una velocidad de crecimiento en bazo y en hígado similar a la cepa original (16). Estudios futuros,que deben incluir las mutantes y sus cepas complementarias correspondientes, podrían indicar si estos genes se requieren para una reacción tisular específica.
En la síntesis de DIM también se ha demostrado la participación de, al menos, uno de los genes similares a los que codifican para una sintasa del ácido micoserósico, denominados msl. Estos genes codifican para una serie de dominios catalíticos requeridos para la síntesis de ácidos grasos saturados.
Dos de estos genes, el msl6 ( pks12) y el msl5 ( pks8 y pks17), fueron inactivados en la cepa H37Rv mediante recombinación homóloga utilizando un sistema de fagos (42,43). La inactivación del gen msl5 no afectó el crecimiento de la cepa en macrófagos alveolares murinos o en pulmones de ratones C57BL6/J infectados por medio de aerosoles (43). Por el contrario, la inactivación del gen msl6 resultó en una drástica disminución en la síntesis de DIM, acompañada de reducción en el crecimiento tanto en macrófagos alveolares murinos como en pulmones de ratones C57BL6/J infectados por vía intranasal (42).
A pesar de los estudios anteriores, aún quedan por determinarse las bases moleculares que puedan explicar la asociación de DIM con virulencia. No es claro todavía si PDIM o DIM funcionan solamente en la superficie celular de la micobacteria, afectando la permeabilidad celular y, posiblemente, la sensibilidad a los mecanismos bactericidas de los macrófagos o si, además, su excreción en el hospedero es importante para la virulencia y la modulación de la respuesta inmune.
Genes involucrados en la síntesis o en la transferencia de los ácidos micólicos y las trehalosas
El papel de los ácidos micólicos en la patogénesis de la tuberculosis se ha confirmado por análisis genéticos recientes en los cuales se han inactivado los genes involucrados en la síntesis y la transferencia de estos ácidos (29,30,33,34). En uno de estos estudios, el gen pcaA ( umaA2) que codifica para una ciclopropano sintasa requerida para la síntesis del anillo proximal ciclopropano de los ácidos micólicos a , fue inactivado por recombinación homóloga en la cepa Erdman de M. tuberculosis (30). Las mutantes pcaA- exhibieron una morfología alterada del factor cordón y un perfil de ácidos micólicos igualmente alterado. Aunque las tres clases mayores de ácidos micólicos estaban presentes, los cetomicolatos eran significantemente más abundantes en la cepa mutante y se presentaron modificaciones en el alfa micolato, como un ligero incremento en la cantidad de ácido micólico transcisciclopropo. El fenotipo alterado de factor cordón de la mutante pcaA- sugiere que el contenido lipídico de la envoltura celular podría relacionarse con las características morfológicas de la bacteria (30).
Estudios in vivo revelaron que estas mutantes se eliminaron más rápidamente en ratones C57B1/6 infectados por vía venosa al compararlas con la cepa original. Los ratones infectados con la mutante también exhibieron un daño pulmonar menos importante. Los resultados anteriores sugieren que la ciclopropano sintasa proximal es necesaria para la supervivencia a largo plazo y la virulencia de M. tuberculosis in vivo (30). Futuras investigaciones podrían revelar si las alteraciones en el contenido de ciclopropano de los lípidos asociados con la pared celular en la mutante pcaA-, incluso TDM, están relacionadas con modulaciones diferentes de la respuesta inmune en el hospedero.
Se generaron por recombinación homóloga cepas Erdman de M. tuberculosis con mutaciones en los genes cmaA2 y mmaA2. Estos genes codifican para una sintasa de ácidos micólicos transciclopropano y una ciclopropano sintasa distal, respectivamente (28,44). Los análisis de la mutante cmaA2- han indicado que el cmaA2 se requiere para la síntesis de los anillos transciclopropano de los ácidos micólicos oxigenados. En el caso de la mutante mmaA2-, se detectaron defectos en la cis-ciclopropanación de los a micolatos (28,44). Estudios futuros podrían involucrar análisis adicionales de estas mutantes tanto in vivo como in vitro.
La inactivación del gen hma ( cmaA, mmaA) que codifica para una enzima que se requiere para la síntesis de ácidos micólicos oxigenados, ha sugerido la forma como este tipo de ácidos micólicos participa en la patogénesis de las micobacterias. Dubnau et al. inactivaron el gen hma en la cepa H37Rv de M. tuberculosis por recombinación homóloga. La comparación de la composición de los lípidos de la cepa original y de la mutante hma- reveló que esta última es incapaz de sintetizar metoximicolatos o cetomicolatos. Estos cambios parecen estar acompañados de modificaciones en la permeabilidad de la pared celular en la cepa mutante, como lo sugieren una captación disminuida de moléculas hidrofílicas e hidrofóbicas, su acumulación y una gran resistencia al peróxido de hidrógeno (H 2 O 2 ) (29).
Estudios in vitro de esta mutante revelaron que aunque el crecimiento en la línea celular THP-1 no se afectó, hubo una disminución significativa en su crecimiento en ratones C57BL/6 infectados mediante aerosoles. La mutante, además, persistió luego de varias semanas de infección, lo cual sugiere que su atenuación para el crecimiento probablemente no se debió a un incremento en la susceptibilidad a los mecanismos bactericidas del hospedero, sino a un efecto en el estado nutricional de la cepa. Estudios futuros en este campo podrían confirmar si la incapacidad de la mutante hma- para adquirir nutrientes específicos puede explicar su comportamiento durante el crecimiento en tejidos de ratón o en el interior de macrófagos humanos (29).
Entre los genes involucrados en la transferencia de ácidos micólicos están los que codifican para el complejo del antígeno 85 (Ag85) (33,45). El complejo está constituido por los antígenos 85A, 85B, 85C, codificados por los genes fbpA, fbpB y fbpC2, respectivamente. Estos antígenos son proteínas ligadoras de fibronectina que catalizan la transferencia de micolatos a trehalosa, un paso necesario para la síntesis de a,a '-trehalosa monomicolato (TMM) y el factor cordón (18). Se ha identificado un cuarto miembro de esta familia, codificada por el gen fbpC1, pero su actividad ligadora micoliltransferasa no ha sido aún demostrada (8,46). Un posible papel del complejo Ag85 en la virulencia de M. tuberculosis podría explicarse por su función en la biosíntesis de la pared celular. Las proteínas del complejo Ag85 también podrían jugar un papel en la virulencia de las micobacterias debido a su habilidad para ligar fibronectina, característica que se ha relacionado con la virulencia en otros microorganismos patógenos (47). Además, debido a sus propiedades ligadoras de fibronectina, se ha propuesto un papel importante para el complejo Ag85 en la entrada de micobacterias patógenas a la célula (48,49).
Armitige y colaboradores obtuvieron mutantes de los genes fbpA y fbpB en la cepa de M. tuberculosis H37Rv por medio de recombinación homóloga (33). Mientras que la inactivación del gen fbpA resultó en crecimiento disminuido en medio de cultivo mínimo en la línea celular humana THP-1 o en la línea celular de macrófagos murinos J774.A1 (33), la inactivación del gen fbpB no afectó el crecimiento de la mutante bajo las mismas condiciones. El análisis de las cepas complementadas confirmaron estos resultados.
Estudios futuros podrían demostrar si las alteraciones observadas en la mutante fbpA- se deben al requerimiento de lípidos o de otros compuestos que se presentan usualmente en los medios enriquecidos o a una susceptibilidad incrementada a lípidos tóxicos que de otra manera serían ligados por la albúmina presente en medios enriquecidos (33). Además, una alteración en el procesamiento de fagosomas o en la activación de los macrófagos por esta mutante también podría resultar en una supervivencia intracelular deteriorada. En relación con lo anterior, estudios previos han mostrado resultados promisorios en ratones inmunizados con vectores de ADN que llevan el gen del Ag 85A (50), lo cual sugiere un posible papel de este antígeno en la respuesta inmunológica a la infección por M. tuberculosis.
La inactivación del gen fbpC2 en un aislamiento clínico de M. tuberculosis, obtenida por mutagénesis con transposones, no afectó significativamente el crecimiento de la cepa en medio mínimo o en macrófagos derivados de médula ósea de ratones BALB/c (34). Estos hallazgos fueron similares a los descritos para la inactivación del gen fbpB en la cepa de M. tuberculosis H37Rv. Como la capacidad de ligamiento a fibronectina es común para FbpA, FbpB y FbpC2, estos resultados sugieren que el efecto de la inactivación del gen fbpA en los patrones de crecimiento podría no estar directamente relacionado con la capacidad de adhesión a la fibronectina.
El análisis de lípidos de la mutante fbpC2- MYC1554 no mostró diferencias significativas en la naturaleza y en las cantidades relativas de micolatos entre la cepa original y la mutante. Sin embargo, la cepa mutante fbpC2- contenía 40% menos micolatos ligados a la pared celular que la cepa original, lo cual sugiere que el Ag 85C podría estar involucrado en la transferencia de micolatos a arabinogalactano. Además, los estudios de permeabilidad celular de esta mutante sugieren que la capa de micolatos puede servir como barrera para la difusión de pequeñas moléculas hidrofílicas e hidrofóbicas. Este fenómeno fue independiente de las subsecuentes vías metabólicas del substrato y no pareció afectar la difusión de isoniacida y otros antibióticos a través de la capa de micolatos (34).
Estudios futuros centrados en el papel específico del complejo del antígeno 85 y los antígenos relacionados codificados por el gen fbpC1 en la virulencia e inmunogenicidad micobacteriana, podrían ser de importancia tanto para el diseño racional de agentes antimicobacterianos dirigidos contra estos antígenos como para la generación de potenciales mutantes inmunogénicas atenuadas.
Los ácidos micolipanoico, micolipénico y micolipodienoico constituyen los mayores componentes de las trehalosas poliaciladas (51). La inactivación del gen msl3 ( pks3 y pks4) en la cepa H37Rv de M. tuberculosis por transducción especializada generó una mutante que no producía ácido micolipanoico ni ácido micolipénico y, además, no producía trehalosas poliaciladas (51). Se necesitan estudios adicionales de esta mutante para demostrar el posible papel del gen msl3 en la virulencia de M. tuberculosis.
Genes involucrados en la síntesis de sulfolípidos
Los sulfolípidos son ácidos grasos metilados y esterificados a trehalosa sulfato (52-54). Se ha sugerido que juegan un papel importante en la patogénesis de las infecciones por micobacterias, posiblemente, a través de la interacción con las células efectoras del sistema inmune del hospedero. Estas interacciones incluyen la inhibición de la activación de los macrófagos (55,56) y la inducción de cambios en la función celular fagocítica, relacionados con procesos inflamatorios exagerados (57).
Se ha descrito la inactivación del gen pks2 ( msl2) en la cepa H37Rv de M. tuberculosis por recombinación homóloga. Este gen codifica para una policétido sintasa involucrada posiblemente en la síntesis de ácidos hidroxiptioseránicos, el mayor componente acil de los sulfolípidos. La caracterización bioquímica de los lípidos celulares reveló que estos mutantes son deficientes en sulfolípidos (58).
Más recientemente, se ha descrito el gen mmpl8, un miembro de los 13 integrantes de la familia mmpl ya mencionados como importantes en el transporte de lípidos. Este gen (mmp18) juega un papel importante en la síntesis de sulfolípido-1 (SL-1) al igual que el gen de la policétido sintasa 2 ( pks2). La generación de mutantes mmpl8- y pks2- por recombinación homóloga con un sistema de fagos demostró un defecto acentuado en la síntesis de SL-1 en ambas mutantes. El análisis de estas cepas en ratones C57/B6 infectados por vía venosa sugieren que SL-1 no es absolutamente indispensable para la replicación de M. tuberculosis durante la infección y que MmpL8 es importante para la persistencia y el crecimiento bacteriano sostenido (59).
Los hallazgos anteriores cuestionan el papel directo de SL-1 en la patogénesis de la fase aguda de la infección, pero sugieren su importancia en las formas crónicas y persistentes lo que señala su potencial como blancos para el diseño de nuevos medicamentos antituberculosos que tengan utilidad en el tratamiento de las formas latentes de la enfermedad.
Genes que codifican para proteínas asociadas con la pared celular
Las proteínas asociadas con la pared celular incluyen las proteínas exportadas que se asocian con la envoltura celular después de ser translocadas a través de la membrana citoplasmática (60). Entre estas proteínas que se asocian con la pared celular de las micobacterias, están el complejo del antígeno 85, descrito anteriormente (18,61), y la proteína Erp (20). La proteína Erp contiene una secuencia señal N-terminal y se sospecha que sea translocada por la vía dependiente de la proteína Sec (60).
Se ha descrito la inactivación del gen erp ( pirG), que codifica para la proteína Erp, en la cepa H37Rv de M. tuberculosis por medio de recombinación homóloga. La mutante que se obtuvo exhibió disminución en su replicación, efectos citopáticos reducidos en los macrófagos derivados de la médula ósea de ratones BALB/c y disminución en la supervivencia de los ratones BALB/c infectados por vía intravenosa (20). Estos resultados, junto con el análisis de microscopía inmunoelectrónica de macrófagos J774, indican que la proteína Erp se produce en los fagosomas de M. tuberculosis y, posiblemente, tiene tráfico intracelular (20). Esto sugiere un importante papel para esta proteína durante el desarrollo del fagosoma y en la multiplicación del patógeno en el interior de la célula hospedera. Los estudios futuros deben enfocarse en entender el papel específico que juega la proteína Erp en los procesos relacionados con el estado intracelular y la patogénesis del microorganismo. En este contexto, estudios recientes han demostrado que la naturaleza del gen erp afecta significativamente el número y el tamaño de las lesiones pulmonares en ratones BALB/c infectados por vía venosa; se observaron lesiones individuales de mayor tamaño y mayor superficie total acumulada de las lesiones en la mutante erp- complementada con el gen erp proveniente de cepas de micobacterias virulentas, comparadas con la mutante complementada con genes provenientes de cepas no virulentas (62).
Inactivación de genes relacionados con la biosíntesis, el metabolismo y el transporte de metabolitos y nutrientes
Genes involucrados en la biosíntesis de aminoácidos. Las micobacterias son capaces de sintetizar todos los aminoácidos necesarios para su crecimiento; se han descrito fenotipos atenuados auxotrópicos de aminoácidos en estos microorganismos (63-65). La biosíntesis de aminoácidos en las micobacterias parece tener las vías usuales descritas en otros microorganismos (66,67). No obstante, existen diferencias que parecen encontrarse en los mecanismos reguladores para estas vías (66,67), tal como lo ha mostrado la vía de la familia del aspartato en M. smegmatis (68). El estudio de mutantes auxotróficas para aminoácidos es una herramienta para entender las características ambientales que confronta la micobacteria que reside en el interior del macrófago y los mecanismos que el microorganismo ha desarrollado para sobrevivir en este tipo de ambiente. Además, las mutantes auxotróficas atenuadas también se pueden considerar como potenciales candidatas a vacunas.
Entre los genes involucrados en la biosíntesis y la captación de aminoácidos en los cuales se ha descrito inactivación en M. tuberculosis, están: lysA, leuD, proC, metB, hisD, trpD, argF y glnA1. Estos genes codifican respectivamente para mesodiaminopimelato decarboxilasa, isopropil-malatoisomerasa, pirroline-5-carboxilato-reductasa, cistatión-gamma sintasa, histidinol dehidrogenasa, antranilato fosforibosiltransferasa, ornitina carbamoiltransferasa y glutamina sintasa (68-72). Las anteriores enzimas están involucradas en el metabolismo o en la biosíntesis de los aminoácidos: lisina, leucina, metionina, histidina, prolina, triptófano, arginina y glutamina, respectivamente (67,69-78).
En la cepa H37Rv de M. tuberculosis se ha descrito la inactivación de los genes lysA, leuD, proC, metB, hisD, trpD y argF mediante recombinación homóloga (68-71). El gen glnA1 ha sido inactivado en M. tuberculosis cepa Erdman por intercambio alélico utilizando un vector termosensible (72).
Aunque la inmunización única de ratones C57BL/ 6 con la mutante de lysA no confirió protección contra un reto con M. tuberculosis virulenta administrada por aerosol, el protocolo con dos dosis de la mutante resultó en niveles de protección similares a los conferidos por la BCG. Además, el auxótrofo lysA- no persistió en ratones inmunocompetentes ni en ratones inmuno-comprometidos, probablemente debido a la incapacidad de esta mutante de sobrevivir en ausencia de lisina. Se ha sugerido el uso potencial de esta mutante auxotrófica como una vacuna de refuerzo independiente de replicación (79).
Los patrones de crecimiento intracelular han mostrado que la mutante leuD- es incapaz de replicarse en el interior de macrófagos derivados de la médula ósea de ratones BALB/cJ, en contraste con la cepa original y con las mismas cepas complementadas (69). Esto sugiere que la leucina del hospedero no es utilizada por la micobacteria intracelularmente. Esta mutante no fue superior a la BCG en términos de protección en ratones BALB/cJ retados con M. tuberculosis (69). Los estudios futuros con este modelo animal podrían dirigirse a definir si este comportamiento puede explicarse por un bajo nivel de persistencia de las mutantes auxotróficas leu-.
En contraste con las características descritas para las mutantes leu-, la inactivación del gene metB- generó una cepa no auxotrófica que exhibió una tasa de supervivencia similar a la de la cepa nativa en macrófagos derivados de la médula ósea de ratones BALB/c. Esta cepa fue virulenta en ratones SCID infectados por vía intravenosa, los cuales han mostrado ser altamente susceptibles a la infección con M. tuberculosis. La mutante metB- tuvo una menor virulencia en ratones inmunocompetentes DBA infectados por vía intravenosa (80). Las futuras investigaciones son necesarias para indicar si la actividad MetB residual estaba presente en la mutante metB- o si el producto de otro gen puede compensar en la mutante la pérdida de la actividad MetB. Los genes candidatos para estas funciones substitutas incluyen el gen metZ el cual se ha identificado en la secuencia genómica de M. tuberculosis (8), que también puede codificar para una cistationina ã -sintasa (70).
En relación con las características observadas en las mutantes proC- y trpD-, como son la reducción significativa en la habilidad para multiplicarse en el interior de los macrófagos derivados de la médula ósea de ratones BALB/c y menor virulencia en ratones SCID y DBA, se ha sugerido un papel importante de estos genes en la virulencia de M. tuberculosis (80). La vacunación en modelos animales utilizando estos mutantes y retados con M. tuberculosis confirió niveles de protección similares (mutante proC-) o más altos ( trpD) que los conferidos por BCG, lo cual indica un potencial interesante de estas cepas atenuadas como candidatas a vacunas (80). Similarmente, el fenotipo atenuado de la mutante glnA1- en macrófagos humanos THP-1 y en cobayos infectados por aerosoles sugiere que este gen juega un papel importante en la virulencia de M. tuberculosis (72).
Genes involucrados en la captación de hierro y en la regulación dependiente de hierro. Las micobacterias, como la mayoría de los organismos vivos, requieren hierro para un apropiado crecimiento y supervivencia (66,81). Evidencias previas sugieren que la captación de hierro y su regulación pueden jugar un importante papel en la patogénesis de las infecciones causadas por micobacterias, tal como se ha descrito en otros patógenos (66,81). En algunos de estos organismos, el hierro juega un papel importante en la regulación de genes y, por ejemplo, la no disponibilidad de este nutriente puede producir represión de genes de virulencia (67,81,82).
El bacilo tuberculoso es capaz de obtener hierro extracelular a través de la micobactina T, un sideróforo hidrofílico que transfiere el hierro a micobactinas solubles en lípidos y asociadas con la célula (83,84). Varias enzimas involucradas en la biosíntesis de micobactinas son codificadas por el grupo mbt, el cual incluye los genes mbtA a mbtH (8, 67,85).
La expresión de éstos y otros genes relacionados con el transporte de hierro parecen estar bajo el control de genes reguladores. Los genes reguladores responsables del hierro en otras bacterias codifican para las proteínas Fur y DtxR, las cuales son capaces de inhibir la expresión bajo concentraciones altas de hierro mediante la unión a secuencias palindrómicas conservadas en los promotores blanco (86-88). Se ha identificado un homólogo del gen dtxR en M. tuberculosis. Este gen denominado ideR codifica para una metaloproteína dependiente de hierro que reconoce una secuencia promotora específica ('caja de hierro') en otros genes regulados por este metal (8, 89).
Una mutante ideR- de M. tuberculosis H37Rv fue generada por recombinación homóloga utilizando un vector suicida. Debido a la esencialidad del gen ideR, esta mutante solamente se pudo aislar en la presencia de una segunda copia funcional del mismo gen. La mutante generada exhibió mayor sensibilidad al estrés oxidativo comparada con las cepas original y complementada. El análisis posterior de la mutante permitió la identificación de varios genes regulados por hierro y por IdeR, inclusive endogenes codificadores y proteínas involucradas en la síntesis de sideróforos y en el almacenamiento de hierro (89).
El gen mbtB codifica una enzima que, aparentemente, cataliza algunos de los primeros pasos en la biosíntesis de la micobactina T, incluso la condensación del ácido salicílico con serina y la formación de la estructura hidroxifeniltiazolina de este sideróforo (90). Este gen se inactivó en la cepa de M. tuberculosis H37Rv a través de recombinación homóloga (91). El análisis estructural de la mutante demostró crecimiento en presencia del ácido salicílico [ 7-14C] y reveló que esta cepa no parecía incorporar el marcador en material extraíble de células enteras. Una porción del marcador se encontró incorporado en un metabolito liberado en el medio de cultivo, pero no se le demostró capacidad de ligar hierro. Los ensayos funcionales revelaron, además, que la mutante no exhibió la capacidad de quelar hierro. Estos resultados, junto con la observación de crecimiento deficiente de esta mutante en medio pobre en hierro, sugieren un importante papel para las moléculas queladoras de hierro en el crecimiento de M. tuberculosis (91).
El papel de los sideróforos en la replicación de la micobacteria en el interior de macrófagos humanos está adicionalmente soportado por el crecimiento defectuoso de la mutante mbtB- en células THP-1 comparado con la cepa nativa. El crecimiento defectuoso no se modificó por la incubación previa de los microorganismos en medio enriquecido con hierro (91). Las investigaciones futuras podrían enfocarse en el detalle estructural y en la caracterización funcional de las micobactinas de M. tuberculosis, la identificación de los papeles específicos que juegan otros genes mbt y los genes relacionados con mbt en la biosíntesis y la virulencia de las micobacterias. Además, debe abordarse el estudio y la identificación del papel que juegan los genes reguladores en la expresión de genes dependientes del hierro en M. tuberculosis.
El gen mramp, un homólogo micobacteriano del gen que codifica para la proteína de macrófagos 1, asociada con la resistencia natural (Nramp1), parece jugar un papel importante en la captación de varios cationes, incluso Fe 2+ y Mn 2+ . Una mutante mramp- de M. tuberculosis MT103 generada por recombinación homóloga exhibió crecimiento alterado bajo condiciones limitantes de hierro. El crecimiento y la supervivencia de la mutante en líneas celulares de macrófagos murinos y su supervivencia en ratones BALB/c infectados por vía venosa no se alteraron, lo cual indica que Mramp no juega un papel realmente importante en la virulencia de M. tuberculosis en el modelo murino (92).
Genes involucrados en el transporte de magnesio. La habilidad para transportar cationes como el magnesio parece ser un mecanismo de adaptación bacteriano que le permite a los patógenos intracelulares sobrevivir dentro del fagosoma (11,93). Se ha sugerido que en M. tuberculosis este mecanismo puede estar involucrado en la supervivencia del micro-organismo dentro de un fagosoma ligeramente acídico y limitado en Mg 2+ (11).
El gen mgtC, que codifica para una proteína C transportadora de magnesio, fue identificado en M. tuberculosis basado en la identidad de su producto con el del gen mgtC de Salmonella enterica (8,11). Este gene se inactivó en la cepa Erdman de M. tuberculosis por recombinación homóloga con un fragmento lineal que contenía el gen inactivado (11).
El análisis de los patrones de crecimiento de la mutante en diferentes medios indicó que el gen mgtC se requiere para el crecimiento de M. tuberculosis bajo condiciones de baja concentración de Mg + . Este efecto fue más evidente bajo condiciones levemente acídicas. La mutante mgtC- también exhibió crecimiento alterado en macrófagos derivados de monocitos humanos y menor supervivencia en ratones BALB/c infectados por vía venosa, lo cual indica que el gen mgtC juega un papel importante en el crecimiento de M. tuberculosis en macrófagos humanos y en su virulencia en ratones (11).
Genes involucrados en el metabolismo de ácidos grasos. Los ácidos grasos son la mayor fuente de carbono y energía que pueden catabolizarse o incorporarse en complejos lipídicos y se ha sugerido que juegan un importante papel en el metabolismo de M. tuberculosis y de otras bacterias (94,95). El catabolismo de los ácidos grasos incluye el ciclo de la â -oxidación, la cual produce varias moléculas de acetil-CoA y la vía acortada del glioxilato, la cual está involucrada en la utilización de las moléculas de acetil-CoA generadas por la vía de la â -oxidación (95).
La expresión de la isocitrato liasa (Icl), una enzima derivada del glioxilato, está aumentada en micobacterias durante la infección de macrófagos y durante la adaptación para sobrevivir bajo condiciones de anaerobiosis (96-99). El gen que codifica para esta enzima, icl, ha sido inactivado en la cepa Erdman de M. tuberculosis por recombinación homóloga (94).
La mutante icl- obtenida por recombinación homóloga mostró persistencia y virulencia disminuida en ratones BALB/c infectados por vía intravenosa, comparada con las cepas nativa y complementada. La virulencia de esta mutante en ratones IFN-g -/- infectados por vía intravenosa, sugiere la posible relación entre el estado inmune del hospedero y el estado metabólico de M. tuberculosis in vivo (94).
La evaluación de la expresión Icl-Gfp (isocitratoliasa- green fluorescent protein) en cepas transformadas con un plásmido que contiene la fusión icl-gfp, en macrófagos murinos de médula ósea inactivados y activados con INF... g con LPS, ha mostrado que la expresión de Icl permaneció elevada luego de 24 horas posinfección sólo en los macrófagos infectados. Además, la supervivencia de la mutante icl- fue significativamente más reducida en macrófagos activados, comparados con las cepas nativa y complementada. En el resto de los macrófagos, la supervivencia de la mutante fue ligeramente más reducida comparada con las mismas cepas. Estos resultados sugieren que la Icl juega un papel muy importante en la supervivencia bacteriana en el interior de los macrófagos activados (94). Las investigaciones futuras podrían indicar si es posible desarrollar inhibidores de Icl con actividad preferencial contra micobacterias en estado de persistencia.
El ácido pantoténico se requiere para la síntesis de la coenzima A y de la proteína transportadora de acilos (PTA), los cuales juegan un papel importante en el metabolismo de los ácidos grasos, en el ciclo del ácido tricarboxílico y en otras reacciones, inclusive la biosíntesis de policétidos (100). Una mutante doble de los genes panC y panD, que están involucrados en la síntesis de novo del pantotenato, fue generada en M. tuberculosis H37Rv por recombinación homóloga utilizando un sistema de transducción especializada. La mutante demostró ser altamente atenuada en ratones inmunocomprometidos SCID e inmunocompetentes BALB/c infectados por vía venosa. Además, confirió un nivel de protección comparable con el de la BCG en ratones C57BL/ 6j inmunizados por vía subcutánea (101).
Inactivación de genes relacionados con la transducción de señales y la transcripción
Sistemas de dos componentes. Los sistemas de regulación de dos componentes son comunes en microorganismos y controlan la transducción de señales en respuesta al medioambiente y a los estímulos internos en la bacteria. Estos sistemas se caracterizan por un dominio histidinoproteín-cinasa, el cual es capaz de catalizar su propia fosforilación y funciona como el sensor de la señal y por un dominio regulador de la respuesta que es capaz de controlar la transcripción de los genes blanco (102). Se han descrito algunos sistemas de dos componentes esenciales, inclusive genes que están involucrados en la permeabilidad de la membrana (103) y en el control del ciclo celular (104-106). Se han identificado once pares de sensores de histidino-cinasas y secuencias reguladoras de respuestas en M. tuberculosis (8), incluso los genes mtrA- mtrB (107).
La inactivación del gen mtrA que codifica para un activador transcripcional putativo, se ha descrito en la cepa H37Rv de M. tuberculosis por medio de recombinación homóloga. La esencialidad del gen mtrA se probó por el hecho de que su reemplazo solamente se obtuvo después de transformar con un plásmido que contenía el mismo gen (107). Este gen se expresó durante el crecimiento de M. tuberculosis en medio 7H9 y en macrófagos de origen humano y murino (107). Las investigaciones futuras, inclusive el análisis del gen mtrB y de su expresión relativa, podrían revelar funciones específicas controladas por este sistema de dos componentes en los mecanismos de adaptación y virulencia de M. tuberculosis.
Otro sistema de dos componentes descrito en M. tuberculosis es el denominado prrA-prrB (97). Se obtuvo una mutante en el gen prrA en M. tuberculosis Mt103 mediante mutagénesis por transposones. La mutante exhibió una velocidad de crecimiento disminuida durante los primeros días de infección en macrófagos murinos derivados de médula ósea de ratones BALB/c. La velocidad de crecimiento de la mutante se equiparó con la cepa original en estadios más tardíos de la infección, lo cual sugiere un posible papel de este sistema en las fases tempranas de crecimiento intracelular de M. tuberculosis (108).
El sistema de dos componentes PhoP/PhoR se ha asociado con el metabolismo del fosfato en M. tuberculosis (8). Este sistema es similar al sistema PhoP/PhoQ, el cual controla la transcripción de varios genes relacionados con virulencia en diversos patógenos (109). Se generó una mutante en el gen phoP en M. tuberculosis Mt103 por recombinación homóloga utilizando un plásmido con el gen phoP inactivado. Se observaron alteraciones en la multiplicación de la mutante en macrófagos derivados de médula ósea, a pesar de que la supervivencia total de los microorganismos dentro de los macrófagos no se vio afectada. Esta mutante resultó atenuada en ratones BALB/c infectados por vía venosa. Los resultados sugieren un posible papel para PhoP en el crecimiento intracelular de M. tuberculosis (109).
Factores sigma. Los factores sigma primarios y alternativos de ARN polimerasa confieren a esta enzima la habilidad de unirse a secuencias promotoras específicas e iniciar la transcripción (88). La expresión genética condicional en respuesta a las condiciones medioambientales, incluso la expresión de genes que codifican para factores de virulencia, se lleva a cabo comúnmente a través del reemplazo de los factores sigma primarios por factores sigma alternativos con cambios resultantes en la especificidad del promotor de la ARN polimerasa (88,110-115). Ya se han identificado trece secuencias de factores sigma putativos en M. tuberculosis, incluso los genes del factor sigma sigA y sigB, diez factores sigma con función extracitoplasmática y un tipo de factor sigma relacionado con condiciones de estrés y esporulación, conocido como sigF (8).
El gen whiB3 codifica una proteína (WhiB3) que interactúa específicamente con el factor sigma principal RpoV. Una mutante de whiB3 se generó en la cepa H37Rv por recombinación homóloga con un sistema de transducción especializada. No se observaron defectos en la replicación de esta cepa en ratones C57Bl/6 o BALB/c infectados por vía venosa, o en cobayos infectados por vía subcutánea. Sin embargo, la supervivencia fue mayor en los ratones infectados con la mutante comparada con la de aquellos infectados con la cepa original. Estos resultados sugieren un papel para el gen whiB3 en la virulencia de M. tuberculosis (116).
Los miembros de la familia extracitoplasmática de factores sigma alternativos (ECF) parecen jugar un papel importante en la regulación de la expresion genética relacionada con las respuestas al estrés oxidativo y térmico. Se ha llegado a sugerir que estos factores pueden jugar un papel en la expresión de antígenos de superficie y en la secreción de sustancias inmunomoduladoras micobacterianas (97,117,118). En cuanto al factor SigF, se ha demostrado que el gen sigF es regulado positivamente en la fase de crecimiento estacionario, en condiciones de depleción de nitrógeno y choque frío y durante el crecimiento en macrófagos (97,119,120). La exposición a medicamentos antimicobacterianos, inclusive etambutol, rifampicina, estreptomicina y cicloserina, también conducen a la regulación positiva de este gen (120).
Una mutante sigH de M. tuberculosis H37Rv fue generada por recombinación homóloga mediante un sistema de transducción especializada. Como se esperaba, la mutante fue más susceptible al estrés oxidativo que la cepa original y la complementada (121). Igualmente, una mutante para este gen de la cepa de M. tuberculosis H37Rv generada por recombinación homóloga con un vector suicida, fue más susceptible al estrés térmico y oxidativo comparado con las cepas nativa y complementada, pero no exhibió alteraciones en su crecimiento dentro de macrófagos murinos. En este caso, el análisis de los cambios en la expresión génica global después de la exposición a diamida permitió la identificación de 39 genes dependientes de sigH, incluso sigB, sigE y el mismo sigH (117). El gen sigH fue también inactivado en M. tuberculosis CDC1551 por recombinación homóloga con un plásmido que llevaba el gen inactivado, lo cual demuestra persistencia en ratones C57BL/6 infectados por aerosoles. Esta mutante, además, indujo lesiones immunopatológicas reducidas en estos ratones, al igual que en ratones CH3 infectados por vía venosa. Estos datos sugieren que, aunque el gen sigH de M. tuberculosis no es indispensable para el crecimiento y la supervivencia de la bacteria en el hospedero, sí es indispensable en la generación de lesiones inmunopatológicas causadas por M. tuberculosis. La mortalidad reducida inducida por la mutante sigH- sugiere un papel de este gen en la letalidad de M. tuberculosis en ratones (118).
Una mutante en el gen sigE fue generada en M. tuberculosis H37Rv por recombinación homóloga con un vector suicida que contenía el gen inactivado. La mutante fue más susceptible al choque térmico y a SDS que las cepas nativa y complementada. Esta mutante también exhibió un crecimiento defectuoso en macrófagos humanos y murinos, así como una mayor sensibilidad a macrófagos murinos activados con lipopoli-sacárido e interferón gamma. El análisis de esta mutante mostró que sigE está involucrado en la respuesta al estrés y, además, permitió la identificación de varios genes, incluso sigB, que son inducidos de una forma dependiente de sigE después del estrés inducido por SDS (122).
El gen sigF ha sido inactivado en la cepa CDC1551 de M. tuberculosis mediante recombinación homóloga. La mutante obtenida alcanzó alta densidad en medios ricos durante la fase estacionaria comparada con la cepa nativa. Además, no presentó retraso en la misma fase estacionaria de crecimiento cuando se inoculó en medio fresco según los procedimientos estándar. Las pruebas de sensibilidad de la mutante y de las cepas complementadas indicaron que la mutación en sigF resultó en una alta susceptibilidad a medicamentos tipo rifamicinas, inclusive la rifampicina (110).
Un modelo de infección in vitro indicó que la supervivencia a corto plazo y la proliferación de la mutante sigF- en monocitos de sangre periférica humana fueron comparables con los que exhibió la cepa nativa. No se encontraron diferencias significativas entre la susceptibilidad de la mutante sigF- y la cepa nativa en la muerte mediada por linfocitos in vitro (110). Sin embargo, los estudios en modelos de infección en ratones BALB/c infectados por vía intravenosa indicaron que la inactivación del gen sigF resultaba en una virulencia reducida y los animales exhibieron un perfil de citocinas tipo Th2 después de ser infectados (110). Estos resultados sugieren que el SigF juega un importante papel en este modelo de infección animal. Las futuras investigaciones podrían indicar si este efecto también es evidente en el perfil de citocinas tipo Th1 en el modelo murino.
Inactivación de genes relacionados con resistencia a mecanismos bactericidas de los macrófagos
Uno de los aspectos claves en las infecciones causadas por micobacterias es la interacción con los macrófagos, las células en las que el microorganismo se multiplica en el hospedero. La actividad bactericida propia de estas células es capaz de destruir más del 90% de los bacilos; sin embargo, los que logran sobrevivir son suficientes para iniciar su replicación intracelular (123). Por tanto, la habilidad de las micobacterias patógenas para sobrevivir dentro de los macrófagos es un determinante crucial en la patogénesis de estas infecciones.
Para sobrevivir dentro de las células fagocíticas, las micobacterias deben evitar los mecanismos bactericidas de estas células, como las enzimas lisosómicas y la actividad de los reactivos intermediarios del oxígeno y del nitrógeno (124,125). Para este fin, las micobacterias patógenas usan diversas estrategias. Por ejemplo, M. tuberculosis y las especies relacionadas sintetizan sustancias que inhiben la acidificación del fagosoma (126) y la fusión del fagolisosoma (127). Otros productos génicos que pueden contribuir a la detoxificación de los reactivos intermediarios del oxígeno o del nitrógeno son las enzimas catalasa/peroxidasa (KatG), alkil-hidroperoxidasa (AhpC) y superóxido dismutasa (SOD) (13,128,129).
La enzima KatG, codificada por el gen katG, es una catalasa/peroxidasa que parece proteger a M. tuberculosis de la actividad bactericida de los reactivos intermediarios del oxígeno (13,129,130). El gen katG parece estar cotranscrito en una misma región reguladora con el gen furA, que codifica para la proteína FurA, la cual es reguladora de la captación de hierro (131). Las mutaciones en katG que codifican para una catalasa/ peroxidasa incapaz de convertir la isoniacida (INH) a su forma activa, confieren resistencia a este fármaco (130,132). Se han realizado diversos estudios para definir el efecto que las mutaciones en este gen tienen en la fisiología y la patogénesis de M. tuberculosis (130,131). El análisis de varias cepas isogénicas indicaron que la mutación que ocurre más frecuentemente, asociada con la resistencia a INH, no afecta la virulencia de la cepa en ratones BALB/c infectados por vía intravenosa (130).
La enzima alkil hidroperoxidasa (AhpC) es capaz de detoxificar peróxidos orgánicos y, probable-mente, también peróxido de hidrógeno (133-135). La mayoría de los estudios demuestra bajos niveles de AhpC en M. tuberculosis (128,136-138). Esta enzima se ha inactivado en M. tuberculosis H37Rv por recombinación homóloga utilizando un plásmido con el gen inactivado. La mutante obtenida fue tan virulenta como la cepa original en ratones BALB/c infectados por vía intravenosa (138). Estos resultados sugieren que este gen no juega un papel importante en el inicio de la infección. Sin embargo, el hallazgo de niveles elevados de expresión de ahpC en cultivos estáticos de M. tuberculosis (138) obligan a definir en el futuro el papel de esta enzima en niveles posteriores de infección.
Dos genes en M. tuberculosis codifican las enzimas superóxido dismutasas, sodA y sodC. Estas proteínas parecen jugar un papel importante en la protección del microorganismo contra la acción de los intermediarios reactivos del oxígeno. El análisis de la mutante secA2- de H37Rv generada por recombinación homóloga usando un vector suicida contraselectivo, demostró que SodA es una proteína dependiente de la proteína SecA2 para su secreción (139).
Con respecto a SodC, una mutante sodC- de M. tuberculosis cepa Erdman, obtenida por recombinación homóloga utilizando un fragmento lineal con el gen sodC inactivado, presentó un in-cremento significativo en la sensibilidad a superóxido de origen externo. Esta mutante fue también más susceptible a los mecanismos bactericidas de macrófagos peritoneales murinos activados con interferón gamma (INF-g ) y generadores de productos de la explosion respiratoria (13).
Existe también una gran cantidad de genes micobacterianos con función controladora ( housekeeping genes) cuya expresión es necesaria para que estos microorganismos sobrevivan a la acción bactericida de las células fagocíticas. Por ejemplo, las mutantes de M. tuberculosis auxotróficas para leucina (69), micobactina (91) o purinas (45) poseen fenotipos atenuados caracterizados por supervivencia disminuida en macrófagos. Los estudios relacionados con algunos de estos genes se han descrito en párrafos anteriores.
En el
cuadro 1 que aparece a continuación, se presenta la bibliografía relacionada con la inactivación de genes de M. tuberculosis con el fin de facilitarle a los lectores la consulta de la misma.
Consideraciones finales
Desde la secuenciación por primera vez del genoma de M. tuberculosis H37Rv en 1998, ha aparecido una gran cantidad de información relacionada con las funciones adscritas a los genes del microorganismo como lo confirma la actual revisión. Esto todavía es una porción muy pequeña del total del genoma compuesto por 4'411.529 pares de bases que conforman aproximadamente 4.000 genes (8). Aún así, solamente, aproximadamente, al 52% de los genes del genoma de H37Rv se les ha adscrito funciones precisas o putativas de acuerdo con la homología con genes de otros microorganismos, mientras que el 48% restante son genes hipotéticos conservados o genes con función desconocida (140).
Las futuras investigaciones probablemente continuarán enfocándose en la inactivación de genes para identificar blancos útiles en el diseño de vacunas, agentes terapéuticos y pruebas diagnósticas. Los estudios comparativos de expresión génica en diferentes cepas, estadios y modelos de infección pueden jugar un papel importante en la identificación de dichos genes.
Especial énfasis se le ha dado a la atenuación racional de cepas con el fin de generar candidatas para vacunas. A este respecto, sería de utilidad para futuras investigaciones recordar que dichas cepas deberán poseer inactivaciones en dos genes, por lo menos, idealmente involucrados en diferentes vías metabólicas del bacilo. Estas cepas deben exhibir un grado adecuado de atenuación, de tal modo que sean lo suficientemente inmunogénicas y así generen una respuesta inmunológica protectora que sea útil en el caso eventual de una infección. Idealmente, también la inactivación de genes deberá generar cepas que sean compatibles con pruebas diagnósticas y que permitan una rápida y eficaz diferenciación entre individuos vacunados e infectados. Además, las cepas atenuadas deberán ser productos finales carentes de marcadores, principalmente marcadores que confieran resistencia a agentes quimioterapéuticos, para evitar la transferencia de estos genes a los aislamientos clínicos (141).
Estudios adicionales podrían eventualmente emplear la inactivación de genes para identificar posibles candidatos con potencial en otros tipos de vacunas, incluidas vacunas de subunidades naturales o sintéticas o vacunas de ADN, o para identificar blancos potenciales para pruebas diagnósticas y nuevos agentes quimioterapéuticos. Finalmente, vale la pena anotar que aunado al conocimiento del genoma mico-bacteriano, la reciente secuenciación del genoma humano abre aun nuevas puertas para el entendimiento de la patogenicidad en tuberculosis.
Las futuras investigaciones basadas en el estudio genético de las interacciones hospedero-bacteria, a nivel individual y de población, indudablemente jugarán un papel primordial en el desentrañamiento de los misterios que encierra dicha interacción y proporcionarán nuevos conocimientos útiles para el diagnóstico, la prevención y el tratamiento de la tuberculosis
Agradecimientos
El presente manuscrito fue realizado gracias a un convenio marco Universidad de Nebraska-CIB y al proyecto cofinanciado por Colciencias código 2213-04-11899.
Correspondencia:
Jaime Robledo, Unidad de Bacteriología y Micobacterias,
Corporación para Investigaciones Biológicas, Carrera 72ª No.78B-141, Medellín, Colombia.
Teléfono: 441 0855; fax: 441 5514
Recibido: 15/08/03; aceptado: 31/01/04
Referencias
1. Koch RD. Aetiologie der Tuberculos. Ber Klin Wochenschr 1882; 19: 221. Reprinted as a translation by Pinner B, Pinner M. Am Rev Tuberc 1932;25:285-323. [ Links ]
2. Daniel TM, Bates JH, Downes KA. History of tuberculosis. En: Bloom B, editor. Tuberculosis: pathogenesis, protection and control. Washington D.C.: American Society for Microbiology Press; 1994. p. 13-24. [ Links ]
3. Bloom BR, Murray CJL. Tuberculosis: commentary on a reemergent killer. Science 1992; 257: 1055-64. [ Links ]
4. Dye CS, Scheele S, Dolin P, Pathania V, Raviglione MC. Global burden of tuberculosis: estimated incidence, prevalence, and mortality by country. JAMA 1999; 282: 677-86. [ Links ]
5. Jacobs WR Jr., Tuckman M, Bloom BR. Introduction of foreign DNA into mycobacteria using a shuttle phasmid. Nature 1987; 327: 532-5. [ Links ]
6. Bardarov S, Kriakov J, Carriere C, Yu S, Vagabonded C, Macadam RA, et al. Conditionally replicating mycobacteriophages: a system for transposon delivery to Mycobacterium tuberculosis. Proc Natl Acad Sci USA 1997; 94: 10961-6. [ Links ]
7. Pelicic V, Jackson M, Reyrat JM, Jacobs WR Jr., Gicqel B, Guilhot C. Efficient allelic exchange and transposon mutagenesis in Mycobacterium tuberculosis. Proc Natl Acad Sci USA 1997; 94: 10955-60. [ Links ]
8. Cole ST, Brosch R, Parkhill J, et al. Desciphering the biology of Mycobacterium tuberculosis from the complete genome sequence. Nature 1998; 393: 537-44. [ Links ]
9. Konickova-Radochova M, Konicek J, Malek L. The study of mutagenesis in Mycobacterium phlei. Folia Microbiol 1970; 15: 88-102. [ Links ]
10. Balasubramanian V, Pavelka MS Jr, Bardarov SS, Martin J, Weisbrod TR, McAdam RA, et al. Allelic exchange in Mycobacterium tuberculosis with long linear recombination substrates. J Bacteriol 1996; 178: 273-9. [ Links ]
11. Buchmeier N, Blanc-Putard A, Ehrt S, Piddington D, Riley L, Goisman EA. A parallel intraphagosomal survival strategy shared by Mycobacterium tuberculosis and Salmonella enterica. Mol Microbiol 2000; 35: 1375-82. [ Links ]
12. Chacon O, Feng Z, Harris NB, Caceres NE, Adams LG, Barletta RG. Mycobacterium smegmatis D-alanine racemase mutants are not dependent on D-alanine for growth. Antimicrob Agents Chemother 2002; 46: 47-54. [ Links ]
13. Piddington DL, Fang FC, Laessig T, Cooper AM, Orme IM, Buchmeier N. Cu, Zn superoxide dismutase of Mycobacterium tuberculosis contributes to survival in activated macrophages that are generating an oxidative burst. Infect Immun 2001; 69: 4980-7. [ Links ]
14. Pelicic V, Reyrat JM, . Expression of the Bacillus subtilis sacB gene confers sucrose sensitivity on mycobacteria. J Bacteriol 1996; 178: 1197-9. [ Links ]
15. Camacho LR, Ensergueix D, Perez E, Gicquel B, Guilhot C. Identification of a virulence gene cluster of Mycobacterium tuberculosis by signature-tagged transposon mutagenesis. Mol Microbiol 1999; 34: 257-67. [ Links ]
16. Cox JS, Chen B, McNell M, Jacobs WR Jr. Complex lipid determines tissue-specific replication of Mycobacterium tuberculosis in mice. Nature 1999; 402: 79-83. [ Links ]
17. Sassetti CM, Boyd DH, Rubin EJ. Genes required for mycobacterial growth defined by high density mutagenesis. Mol Microbiol 2003; 48: 77-84. [ Links ]
18. Trias J, Benz R. Permeability of the cell wall of Mycobacterium smegmatis. Mol Microbiol 1994; 14: 283-90. [ Links ]
19. Belisle JT, Vissa VD, Slevert T, Takayama K, Brennan PJ, Besra GS. Role of the major antigen of Mycobacterium tuberculosis in cell wall biogenesis. Science 1997; 276: 1420-2. [ Links ]
20. Berthet FX, Rasmussen PB, Rosenkrands I, Andersen P, Gicquel B. Attenuation of virulence by disruption of the Mycobacterium tuberculosis erp gene. Science 1998; 282: 759-62. [ Links ]
21. McNeil M, Daffe M, Brennan PJ. Location of the mycolyl ester substituents in the cell walls of mycobacteria. J Biol Chem 1991; 266: 13217-23. [ Links ]
22. Daffe M, Draper P. The envelope layers of mycobacteria with reference to their pathogenicity. Advances in Microbial Physiology 1998; 39: 131-203. [ Links ]
23. Brennan PJ, Nikaido H. The envelope of mycobateria. Annu Rev Biochem 1995; 64: 29-63. [ Links ]
24. Lederer E. Cell walls of mycobacteria and related organisms; chemistry and immunostimulant proper-ties. Molecular and Cellular Biochemistry 1975; 7: 87-104. [ Links ]
25. Yuan Y, Barry CE 3rd. A common mechanism for the biosynthesis of methoxy and cyclopropyl mycolic acids in Mycobacterium tuberculosis. Proc Natl Acad Sci USA 1996; 93: 12828-33. [ Links ]
26. Yuan Y, Lee RE, Besra GS, Belisle JT, Barry CE 3rd. Identification of a gene involved in the biosynthesis of cyclopropanated mycolic acids in Mycobacterium tuberculosis. Proc Natl Acad Sci USA 1995; 92: 6630-4. [ Links ]
27. George KM, Yuan Y, Sherman DR, Barry CE 3rd. The biosynthesis of cyclopropanated mycolic acids in Mycobacterium tuberculosis. Identification and functional analysis of CMAS-2 J Biol Chem 1995; 270: 27292-8 [ Links ]
28. Glickman MS, Cahill SM, Jacobs WR Jr. The Mycobacterium tuberculosis cmaA2 gene encodes a mycolic acid trans-cyclopropane synthetase. J Biol Chem 2001; 276: 2228-33. [ Links ]
29. Dubnau E, Chan J, Raynaud C, Mohan VP, Lanéelle MA, Yu K, et al. Oxygenated mycolic acids are necessary for virulence of Mycobacterium tuberculosis in mice. Molec Micro 2000; 36: 630-7. [ Links ]
30. Glickman MS, Cox JS, Jacobs WR Jr. A novel mycolic acid cyclopropane synthetase is required for cording, persistence, and virulence of Mycobacterium tuberculosis. Molecular Cell 2000; 5: 717-27. [ Links ]
31. Dubnau E, Laneelle MA, Soares S, Benichou A, Vaz T, Prome D, et al. Mycobacterium bovis BCG genes involved in the biosynthesis of cyclopropyl keto-and hydroxy-mycolic acids. Mol Microbiol 1997; 23: 313-22. [ Links ]
32. Liu J, Barry 3rd CE, Besra GS, Nikaido H. Mycolic acid structure determines the fluidity of the mycobacterium cell wall. J Biol Chem 1996; 271: 29545- 51. [ Links ]
33. Armitige LY, Jagannath C, Wanger AR, Norris SJ. Disruption of the genes encoding antigen 85A and antigen 85B of Mycobacterium tuberculosis H37Rv: effect on growth in culture and in macrophages. Infect Immun 2000; 68: 767-78. [ Links ]
34. Jackson M, Raynaud C, Lanéelle MA, Guilhot C, Laurent-Winter C, Ensergueix D, et al. Inactivation of the antigen 85C gene profoundly affects the mycolate content and alters the permeability of the Mycobacterium tuberculosis cell envelope. Molec Micro 1999; 31: 1573-87. [ Links ]
35. Goren MB, Broki O, Schaefer WB. Lipids of putative relevance to virulence in Mycobacterium tuberculosis: phthiocerol dimycocerosate and the attenuation indicator lipid. Infect Immun 1974; 9: 150-8. [ Links ]
36. Azad AK, Sirakova TD, Fernandez ND, Kolattukudy PE. Gene knockout reveal a novel gene cluster for the synthesis of a class of cell wall lipids unique to pathogenic mycobacteria. J Biol Chem 1997; 272: 16741-5. [ Links ]
37. Azad AK, Sirakova TD, Rogers LM, Kolattuduki PE. Targeted replacement of the mycocerosic acid synthase gene in Mycobacterium bovis BCG produces a mutant that lacks mycosides. Proc Natl Acad Sci USA 1996; 93: 4787-92. [ Links ]
38. Mathur M, Kolattuduki PE. Molecular cloning and sequencing of the gene for mycocerosic acid synthase, a novel fatty acid elongating multifunctional enzyme, from Mycobacterium tuberculosis var. bovis bacillus Calmette-Guerin. J Biol Chem 1992; 267: 19388-95. [ Links ]
39. Fitzmaurice AM, Kolattukudy PE. An acyl-CoA synthase (acoas) gene adjacent to the mycocerosic acid synthase (mas) locus is necessary for mycocerosyl lipid synthesis in Mycobacterium tuberculosis var. bovis BCG. J Biol Chem 1998; 273: 8033-9. [ Links ]
40. Camacho LR, Constant P, Raynaud C, Laneélle MA, Triccas JA, Guicquel B, et al. Analysis of the phthiocerol dimycocerosate locus of Mycobacterium tuberculosis: evidence that this lipid is involved in cell wall permeability barrier. J Biol Chem 2001; 276: 19845-54. [ Links ]
41. Fitzmaurice AM, Kolattukudy PE. Open reading frame 3, which is adjacent to the mycocerosic acid synthase gene, is expressed as an acyl coenzyme A synthase in Mycobacterium bovis BCG. J Bacteriol 1997; 179: 2608-15. [ Links ]
42. Sirakova T, Dubey VS, Kim HJ, Synamon MH, Kolattukudy PE. The largest open reading frame (pks12) in the Mycobacterium tuberculosis genome is envolved in pathogenesis and dimicocerosil phthiocerol synthesis. Infect Immun 2003; 71: 3794-801. [ Links ]
43. Dubey VS, Sirakova TD, Cynamon MH, Kolattukudy PE. Biochemical function of msl5 (pks8 plus pks17) in Mycobacterium tuberculosis H37Rv: biosynthesis of monomethyl branched unsaturated fatty acids. Infec Immun 2003; 185: 4620-5. [ Links ]
44. Glickman MS. The mmaA2 gen of Mycobacterium tuberculosis encodes the distal cyclopropane synthase of the alpha-mycolic acid. J Biol Chem 2003; 278: 7844-9. [ Links ]
45. Jackson M, Phalen SW, Lagranderie M, Ensergueix D, Chavarot P, Marchal G, et al. Persistence and protective efficacy of a Mycobacterium tuberculosis auxotroph vaccine. Infect Immun 1999; 67: 2867-73. [ Links ]
46. Kremer L, Baulard AR, Besra GS. Genetics of mycolic acid biosynthesis. En: Hatfull GF, Jacobs WR Jr, editors. Molecular genetics of Mycobacteria. Washington, D.C.: American Society for Microbiology Press; 2000. p. 173-90. [ Links ]
47. Patti JM, Allen BL, McGavin MJ, Hook M. MSCRAMM-mediated adherence of microorganisms to host tissues. Ann Rev Microbiol 1994; 48: 585-617. [ Links ]
48. Peake P, Gooley A, Britton WJ. Mechanism of interaction of the 85B secreted protein of Mycobacterium bovis with fibronectin. Infec Immun 1993; 61: 4828-34. [ Links ]
49. Espitia C, Laclette JP, Mondragon-Palomino M, Amador A, Campuzano J, Martens A, et al. The PE-PGRS glycine-rich proteins of Mycobacterium tuberculosis: a new family of fibronectin-binding proteins? Microbiology 1999; 145: 3487-95. [ Links ]
50. Huygen K, Content J, Denis O, Montgomery L, Yauman AM, Deck RR, et al. Immunogenicity and protective efficacy of a tuberculosis DNA vaccine. Nat Med 1996; 2: 893-8. [ Links ]
51. Dubey VS, Sirakova TD, Kolattukudy PE. Disruption of msl3 abolishes the synthesis of mycolipanoic and mycolipenic acids required for polyacyltrehalose synthesis in Mycobacterium tuberculosis H37Rv and causes cell aggregation. Mol Microbiol 2002; 45: 1451-9. [ Links ]
52. Goren M, Brokl BO, Das BC. Sulfatides of Mycobacterium tuberculosis: the structure of the principal sulfatide (SL-I). Biochemistry 1976; 15: 2728-35. [ Links ]
53. Alugupalli S, Lanelle MA, Larson L, Daffe M. Chemical characterization of the ester-linked 3-hidroxy fatty acyl-containing lipids in Mycobacterium tuberculosis. J Bacteriol 1995; 177: 4566-70. [ Links ]
54. Kolattukudy PE, Fernandez ND, Azad AK, Fitzmaurice AM, Sirakova TD. Biochemistry and molecular genetics of cell-wall lipid biosynthesis in mycobacteria. Mol Microbiol 1997; 24: 263-70. [ Links ]
55. Pabst MJ, Gross JM, Brozna JP, Goren MB. Inhibition of macrophage priming by sulfatide from Mycobacterium tuberculosis. J Immunol 1988; 140: 634-40. [ Links ]
56. Brozna JP, Horan M, Rademacher JM, Pabst KM, Pabst MJ. Monocyte responses to sulfatide from Mycobacterium tuberculosis: inhibition of priming for enhanced release of superoxide, associated with increased secretion of interleukin-1 and tumor necrosis factor alpha, and altered protein phosphorylation. Infect Immun 1991; 59: 2542-8. [ Links ]
57. Zhang L, Goren MB, Holzer TJ, Andersen BR. Effect of Mycobacterium tuberculosis derived sulfolipid I on human phagocytic cells. Infect Immun 1988; 56: 2876-83. [ Links ]
58. Sirakova TD, Thirumala AK, Dubey VS, Sprecher H, Kolattukudy PE. The Mycobacterium tuberculosis pks2 gene encodes the synthase for the hepta- and octamethyl branched fatty acids required for sulfolipid synthesis. J Biol Chem 2001; 276: 16833-9. [ Links ]
59. Converse SE, Mougous JD, Leavell MD, Leary JA, Bertozzi CR, Cosx JS. Mmpl8 is required for sulpholipid-1 biosynthesis and Mycobacterium tuberculosis virulence. PNAS 2003; 100: 6121-6. [ Links ]
60. Braunstein M, Belisle JT. Genetics of proteins secretion. En: Hatfull GF, Jacobs WR Jr., editors. Molecular genetics of Mycobacteria. Washington, D.C.: American Society for Microbiology Press; 2000. p. 203-20. [ Links ]
61. Abou-Zeid C, Ratliff TL, Wiker HG, Harboe M, Bennedsen J, Rook GA. Characterization of fibronectin-binding antigens released by Mycobacterium tuberculosis and Mycobacterium bovis BCG. Infect Immun 1988; 56: 3046-51. [ Links ]
62. de Mendonca-Lima L, Bordat Y, Pivert E, Recchi C, Neyrolles O, Maitournam A, et al. The allele encoding the mycobacterial Erp protein affects lung disease in mice. Cell Microbiol 2003; 5: 65-73. [ Links ]
63. McAdam RA, Weisbrod TR, Martin J, Scuderi JD, Brown AM, Cirillo JD, Bloom BR, Jacobs WR. In vivo growth characteristics of leucine and methionine auxotrophic mutants of Mycobacterium bovis BCG generated by transposon mutagenesis. Infect Immun 1995; 63: 1004-12. [ Links ]
64. Bange FC, Brown AM, Jacobs WR Jr. Leucine auxotrophy restricts growth of Mycobacterium bovis BCG in macrophages. Infect Immun 1996; 64: 1794-9. [ Links ]
65. Guleria I, Teitelbaum R, McAdam RA, Kalpana G, Jacobs WR Jr, Bloom BR. Auxotrophic vaccines for tuberculosis. Nat Med 1996; 2: 334-7. [ Links ]
66. Ratledge C. Nutrition, growth and metabolism. En: Ratledge C, Stanford J, editors. The biology of mycobacteria. London: United Kingdom Academic Press; 1982. p. 185-271. [ Links ]
67. Pavelka MS Jr. Genetics of Mycobacterium metabolism. En: Hatful GF, Jacobs WR Jr., editors. Molecular genetics of mycobacteria. Washington, D.C.: American Society for Microbiology Press; 2000. p.221-34. [ Links ]
68. Pavelka MS Jr, Jacobs WR Jr. Comparison of the construction of unmarked deletion mutations in Mycobacterium smegmatis, Mycobacterium bovis bacillus Calmette-Guérin and Mycobacterium tuberculosis H37Rv by allelic exchange. J Bacteriol 1999; 181: 4780-9. [ Links ]
69. Hondalus MK, Bardarov S, Russell R, Chan J, Jacobs WR Jr, Bloom BR. Attenuation and protection induced by a leucine auxotroph of Mycobacterium tuberculosis. Infect Immun 2000; 68: 2888-98. [ Links ]
70. Parish T, Gordhan BG, McAdam RA, Duncan K, Mizrahi V, Stoker NG. Production of mutants in amino acid biosynthetic genes of Mycobacterium tuberculosis by homologous recombination. Microbiology 1999; 145: 3497-503. [ Links ]
71. Gordhan BG, Smith DA, Alderton H, McAdam RA, Bancroft GJ, Mizrahi V. Construction and characterization of an auxotrophic mutant of Mycobacterium tuberculosis defective in l-arginine biosyntesis. Infect Immun 2002; 70: 3080-4. [ Links ]
72. Tullius M, Harth G, Horwitz MA. Glutamine synthase GlnA1 is essential for growth of Mycobacterium tuberculosis in human THP-1 macrophages and Guinea pigs. Infec Immun 2003; 71: 3927-36. [ Links ]
73. Patte JC. Biosynthesis of threonine and lysine. En: Neidhardt FC, editor. Escherichia coli and Salmonella cellular and molecular biology. Washington, D.C.: American Society for Microbiology Press; 1996. p. 528-41. [ Links ]
74. Umbarger HE. Biosynthesis of the branched chain aminoacids. En: Neidhardt FC, editor. Escherichia coli and Salmonella cellular and molecular biology. Washington, D.C.: American Society for Microbiology Press; 1996. p. 442-57. [ Links ]
75. Greene RC. Biosynthesis of methionine. En: Neidhardt FC, editor. Escherichia coli and Salmonella cellular and molecular biology. Washington, D.C.: American Society for Microbiology Press; 1996. p. 542-60 [ Links ]
76. Winkler ME. Biosynthesis of histidine. En: Neidhardt FC, editor. Escherichia coli and Salmonella cellular and molecular biology. Washington, D.C.: American Society for Microbiology Press; 1996. p. 485-505. [ Links ]
77. Leisinger T. Biosynthesis of proline. En: Neidhardt FC, editor. Escherichia coli and Salmonella cellular and molecular biology. Washington, D.C.: American Society for Microbiology Press; 1996. p. 434-41. [ Links ]
78. Pittard AJ. Biosynthesis of the aromatic aminoacids. En: Neidhardt FC, editor. Escherichia coli and Salmonella cellular and molecular biology. Washington, D.C.: American Society for Microbiology Press; 1996. p. 458-84. [ Links ]
79. Pavelka MS Jr, Chen B, Kelley CL, Collins FM, Jacobs WR Jr. Vaccine efficacy of a lysine auxotroph of Mycobacterium tuberculosis. Infect Immun 2003; 71: 4190-2. [ Links ]
80. Smith DA, Parish T, Stoker NG, Bancroft GJ. Characterization of auxotrophic mutants of Mycobacterium tuberculosis and their potential as vaccine candidates. Infect Immun 2001; 69: 1142-50. [ Links ]
81. Litwin CM, Calderwood SB. Role of iron in regulation of virulence genes. Clin Microb Rev 1993; 6: 137-49. [ Links ]
82. Manabe YC, Saviola BJ, Sun L, Murphy JR, Bishai WR. Attenuation of virulence in Mycobacterium tuberculosis expressing a constitutively active iron repressor. Proc Natl Acad Sci USA 1999; 96: 12844-8. [ Links ]
83. Gobin J, Hortwitz MA. Exochelins of Mycobaterium tuberculosis remove iron from human iron-binding proteins and donate iron to mycobactins in the M. tuberculosis cell wall. J Exp Med 1996; 183: 1527-32. [ Links ]
84. Golden CA, Kochan I, Spriggs DR. Role of mycobactin in the growth and virulence of tubercle bacilli. Infect Immun 1974; 9: 34-40. [ Links ]
85. Quadri LE, Sello J, Keating TA, Weinreb PH, Walsh CT. Identification of a Mycobacterium tuberculosis gene cluster encoding the biosynthetic enzymes for as-sembly of the virulence-conferring siderophore mycobactin. Chem Biol 1998; 5: 631-45. [ Links ]
86. Earhart CF. Uptake and metabolism of iron and molybdenum. En: Neidhardt FC, editor. Escherichia coli and Salmonella cellular and molecular biology. Washington, D.C.: American Society for Microbiology Press; 1996. p. 1075-90. [ Links ]
87. Tao X, Schiering N, Zeng HY, Ringe D, Murphy JR. Iron, DtxR, and the regulation of diphteria toxin expression. Mol Microbiol 1994; 14: 191-7. [ Links ]
88. Gómez M, Smith I. Determinants of mycobacterial gene expression. En: Hatfull GF, Jacobs WR Jr, editors. Molecular genetics of Mycobacteria. Washington, D.C.: American Society for Microbiology Press; 2000. p. 111-29. [ Links ]
89. Rodríguez GM, Voskuil MI, Gold B, Schollnik GK, Smith I. IdeR, an essential gene in Mycobacterium tuberculosis: role of IdeR in iron-dependent gene expression, iron metabolism, and oxidative stress response. Infec Immun 2002; 70: 3371-81. [ Links ]
90. De Voss JJ, Rutter K, Schroeder BG, Barry CE III. Iron acquisition and metabolism in mycobacteria. J Bacteriol 1999; 181: 4443-51. [ Links ]
91. De Voss JJ, Rutter K, Schroeder BG, Su H, Zhu Y, Barry CE 3rd. The salicylate derived mycobactin siderophores of Mycobacterium tuberculosis are essential for growth in macrophages. Proc Natl Acad Sci USA 2000; 97: 1252-7. [ Links ]
92. Boechat N, Lagier-Roger B, Petit S, Bordat Y, Rauzier J, Hance AJ et al. Disruption of the gene homologous to mammalian Nramp1 in Mycobacterium tuberculosis does not affect virulence in mice. Infec Immun 2002; 70: 4124-31. [ Links ]
93. Blanc-Potard AB, Groisman EA. The Salmonella selC locus contains a pathogenicity island mediating intra macrophage survival. EMBO J 1997; 16: 5376-85. [ Links ]
94. McKinney JD, Zu Bentrup KH, Muñoz-Elias EJ, Miczak A, Chen B, Chan W-T, et al. Persistence of Mycobacterium tuberculosis in macrophages and mice requires the glyoxylate shunt enzyme isocitrate lyase. Nature 2000; 406: 735-8. [ Links ]
95. Nunn WD. Two-carbon compounds and fatty acids as carbon sources. En: Neidhart FC, editor. Escherichia coli and Salmonella: cellular and molecular biology. Washington, D.C.: American Society for Microbiology Press; 1987. p. 285-301. [ Links ]
96. Zu Bentrup KH, Miczak A, Swenson DL, Russell DG. Characterization of activity and expression of isocitrate lyase in Mycobacterium avium and Mycobacterium tuberculosis. J Bacteriol 1999; 181: 7161-7. [ Links ]
97. Graham JE, Clark Curtiss JE. Identification of Mycobacterium tuberculosis RNAs synthesized in response to phagocytosis by human macrophages by selective capture of transcribed sequences (SCOTS). Proc Natl Acad Sci USA 1999; 96: 11554-9. [ Links ]
98. Wayne LG, Lin KY. Glyoxilate metabolism and adaptation of Mycobacterium tuberculosis to survival un-der anaerobic conditions. Infect Immun 1982; 37: 1042-9. [ Links ]
99. Sturgill-Koszycki S, Haddix PL, Russell DG. The interaction of the Mycobacterium and the macrophage analyzed by two-dimensional polyacrylamide gel electrophoresis. Electrophoresis 1997; 18: 2558-65. [ Links ]
100. Jackowski S. Biosynthesis of pantothenic acids and coenzyme A. En: Neidhart FC, editor. Escherichia coli and Salmonella typhimurium: cellular and molecular biology. Washington, D.C.: American Society for Microbiology Press; 1996. p. 687-94. [ Links ]
101. Sambandamurthy VK, Wang X, Chen B, Russell RG, Derrick S, Collins FM, et al. A pantothenate auxotroph of Mycobacterium tuberculosis is highly attenuated and protects mice against tuberculosis. Nat Med 2002; 8: 1171-4. [ Links ]
102. Ninfa AJ. Regulation of gene transcription by extracellular stimuli. En: Neidhardt FC, editor. Escherichia coli and Salmonella cellular and molecular biology. Washington, D.C.: American Society for Microbiology Press; 1996. p. 1246-62. [ Links ]
103. Martin PK, Li T, Sun D, Biek DP, Schmid M. Role in cell permeability of an essential two-component system in Staphylococcus aureus. J Bacteriol 1999; 181: 3666-73. [ Links ]
104. Quon KC, Marczynski GT, Saphiro L. Cell cycle control by an essential bacterial two-component signal transduction protein. Cell 1996; 84: 83-93. [ Links ]
105. Wu J, Ohta N, Newton A. An essential multi-component signal transduction pathway required for cell cycle regulation in Caulobacter. Proc Natl Acad Sci USA 1998; 95: 1443-8. [ Links ]
106. Jacobs C, Domian IJ, Madock JR, Shapiro L. Cell cyle-dependent polar localization of an essential bacterial histidine kinase that controls DNA replication and cell division. Cell 1999; 97: 111-120. [ Links ]
107. Zahrt TC, Deretic V. An essential two-component signal transduction system in Mycobacterium tuberculosis. J Bacteriol 2000; 182: 3832-8. [ Links ]
108. Graham JE, Clark-Curtiss JE. Identification of Mycobacterium tuberculosis RNAs synthesized in response to phagocytosis by human macrophages by selective capture of transcribed sequences (SCOTS). Proc Natl Acad Sci USA 1999; 96: 11554-9. [ Links ]
108. Ewann F, Jackson M, Pethe K, Cooper A, Mielcarek N, Ensergueix D, Gicquel B, Locht C, Supply P. Transient requirement of the PrrA-PrrB two component system for early intracellular multiplication of Mycobacterium tuberculosis. Infec Immun 2002; 70: 2256-63. [ Links ]
109. Pérez E, Samper S, Bordas Y, Guilhot C, Gicquel B & Martín C. An essential role for phoP in Mycobacterium tuberculosis virulence. Mol Micro 2001; 41: 179-87. [ Links ]
110. Chen P, Ruiz RE, Li Q, Silver RF, Bishai WR. Construction and characterization of a Mycobacterium tuberculosis mutant lacking the alternate sigma factor gene, sigF. Infect Immun 2000; 68: 5575-80. [ Links ]
111. Deretic V, Schurr MJ, Boucher JC, Martin DW. Conversion of Pseudomonas aeruginosa to mucoidy in cystic fibrosis: environmental stress and regulation of bacterial virulence by alternative sigma factors. J Bacteriol 1994; 176: 2773-80. [ Links ]
112. Fang FC, Libbly SJ, Buchmeier NA, Loewen PC, Switala J, Harwood J, Guiney DG. The alternatives factor KatF (RpoS) regulates Salmonella virulence. Proc Natl Acad Sci USA 1992; 89: 11978-82. [ Links ]
113. Collins D, Kawakami R, de Lisle G, Pascopella L, Bloom B, Jacobs WR Jr. Mutation of the principals factor causes loss of virulence in a strain of the Mycobacterium tuberculosis complex. Proc Natl Acad Sci USA 1995; 92: 8036-40. [ Links ]
114. Finlay BB, Falkow S. Common themes in microbial pathogenicity revisited. Microbiol Mol Biol Rev 1997; 61: 136-69. [ Links ]
115. Miller JF, Mekalanos JJ, Falkow S. Coordinate regulation and sensory transduction in the control of bacterial virulence. Science 1989; 243: 916-22. [ Links ]
116. Steyn AJC, Collins DM, Hondalus MK, Jacobs WR Jr, Kawakami RP, Bloom BR. Mycobacterium tuberculosis WhiB3 interacts with RpoV to affect host survival but is dispensable for in vivo growth. Proc Natl Acad Sci 2002; 99: 3147-52. [ Links ]
117. Manganelli R, Voskuil MI, Schoolnik GK, Dubnau E, Gomez M, Smith I. Role of the extracytoplasmic-function sigma factor sigma(H) in Mycobacterium tuberculosis global gene expression. Mol Microbiol 2002; 45: 365-74. [ Links ]
118. Kaushal D, Schroeder BG, Tyagi S, Yoshimatsu T, Scott C, Ko C, et al. Reduced immunopathology and mortality despite tissue persistence in a Mycobacterium tuberculosis mutant lacking alternative factor, SigH. Proc Natl Acad Sci 2002; 99: 8330-5. [ Links ]
119. DeMaio J, Zhang Y, Ko C, Young D, Bishai WR. A stationary phase stress-response sigma factor from Mycobacterium tuberculosis. Proc Natl Acad Sci USA 1996; 93: 2790-4. [ Links ]
120. Michele T, Ko C, Bishai WR. Antibiotic exposure induces expression of the Mycobacterium tuberculosis sigF gene: implications for chemotherapy against mycobacterium persistors. Antimicrob Agents Chemother 1999; 43: 218-25. [ Links ]
121. Raman S, Song T, Puyang X, Bardarov S, Jacobs WR Jr, Husson RN. The alternative sigma factor SigH regulates major components of oxidative and heat stress responses in Mycobacterium tuberculosis. J Bacteriol 2001; 183: 6119-25. [ Links ]
122. Manganelli R, Voskuil MI, Schoolnik GK, Smith I. The Mycobacterium tuberculosis ECF sigma factor sigmaE: role in global gene expression and survival in macrophages. Mol Microbiol 2001; 41: 423-37. [ Links ]
123. Fenton MJ, Vermeulen MW. Immunopathology of tuberculosis: roles of macrophages and monocytes. Infect Immun 1996; 64: 683-90. [ Links ]
124. Lowrie DB, Andrwe PW. Macrophage anti-micobacteria mechanisms. BR Med Bull 1988; 44: 624-34. [ Links ]
125. Nathan C, Shiloh MU. Reactive oxygen and nitrogen intermediates in the relationship between mammalian host and microbial pathogens. Proc Natl Acad Sci USA 2000; 97: 8841-8. [ Links ]
126. Sturgill-Koszycki S, Schlesinger PH, Chakraborty P, Haddix PL, Collins HL, Fohk AK, et al. Lack of acidification in Mycobacterium phagosomes produced by exclution of the vesicular proton-ATPase. Science 1994; 263: 678-81. [ Links ]
127. Armstrong JA, Hart PD. Phagosome-lysosome interactions in cultured macrophage infected with virulent tubercle bacilli. Reversal of the usual nonfusion pattern and observation on bacterial survival. J Exp Med 1975; 142: 1-16. [ Links ]
128. Chen L, Xie QW, Nathan C. Alkyl hydroperoxide reductase subunit C (AhpC) protects bacterial and human cells against reactive nitrogen intermediates. Mol Cell 1998; 1: 795-805. [ Links ]
129. Manca C, Paul S, Barry CE 3rd, Freedman VH, Kaplan G. Mycobacterium tuberculosis catalase and peroxidase activities and resistance to oxidative killing in human monocytes in vitro. Infect Immun 1999; 67: 74-9. [ Links ]
130. Pym AS, Saint-Joanis B, Cole ST. Effect of katG mutations on the virulence of Mycobacterium tuberculosis and the implication for transmission in humans. Infect Immun 2002; 70: 4955-60. [ Links ]
131. Pym AS, Domenech P , Honore N, Song J, Deretic V, Cole ST. Regulation of catalase-peroxidase (KatG) expression, isoniazid sensitivity and virulence by furA of Mycobacterium tuberculosis. Mol Microbiol 2001; 40: 879-89. [ Links ]
132. Johnsson K, Froland WA, Schultz PG. Over-ex-pression, purification, and characterization of the catalase-peroxidase KatG from Mycobacterium tuberculosis. J Biol Chem 1997; 272: 2834-40. [ Links ]
133. Jacobson FS, Morgan RW, Christman MF, Ames BN. An alkyl hydroperoxide reductase from Salmonella typhimurium involved in the defense of DNA against oxidative damage. Purification and properties. J Biol Chem 1989; 264: 1488-96. [ Links ]
134. Sherman DR, Mdluli K, Hicky MJ, Arain TN, Morris SL, Barry CE 3rd, et al. Compensatory ahpCgene expression in isoniazid resistance Mycobacterium tuberculosis. Science 1996; 272: 1641-3. [ Links ]
135. Niimura Y, Poole LB, Massey V. Amphibacillus xylanus NADH oxidase and Salmonella typhimurium alkyl-hydroperoxide reductase flavoprotein components show extremely high scavenging activity for both alkyl hydroperoxide and hydrogen peroxide in the presence of S. typhimurium alkyl-hydroperoxide reductase 22-kDa protein component. J Biol Chem 1995; 270: 25645-50. [ Links ]
136. Dhandayuthapani S, Mudd M, Deretic V. Interactions of OxyR with the promoter region of the oxyR and ahpC genes from Mycobacterium leprae and Mycobacterium tuberculosis. J Bacteriol 1997; 179: 2401-9. [ Links ]
137. Zhang Y, Dhandayuthapani S, Deretic V. Molecular basis for the exquisite sensitivity of Mycobacterium tuberculosis to isoniazid. Proc Natl Acad Sci USA 1996; 93: 13212-6. [ Links ]
138. Springer B, Master S, Sander P, Zahrt T, McFalone M, Song J, et al. Silencing of oxidative stress response in Mycobacterium tuberculosis: expression patterns of ahpC in virulent and avirulent strains and effect of ahpC inactivation. Infect Immun 2001; 69: 5967-73. [ Links ]
139. Braunstein M, Espinosa BJ, Chan J, Belisle JT, Jacobs WR. SecA2 functions in the secretion of superoxide dismutase A and in the virulence of Mycobacterium tuberculosis. Mol Microbiol 2003; 48: 453-64. [ Links ]
140. Cole ST. Comparative and functional genomics of the Mycobacterium tuberculosis complex. Microbiology 2002; 148: 2919-28. [ Links ]
141. Barletta RG, Donis RO, Chacon O, Shams H, Cirillo JD. Vaccines against intracellular pathogens. Subcell Biochem 2000; 33: 559-99. [ Links ]














