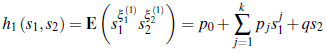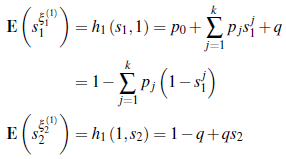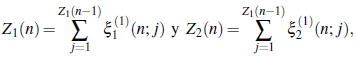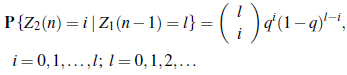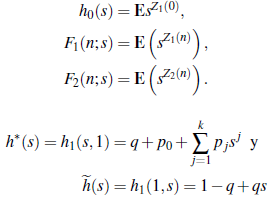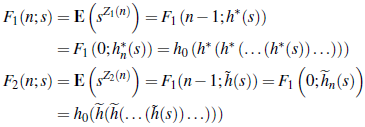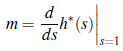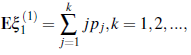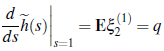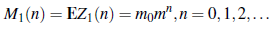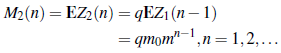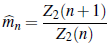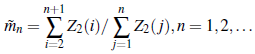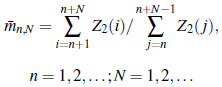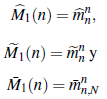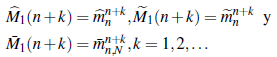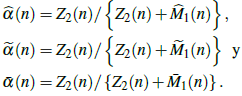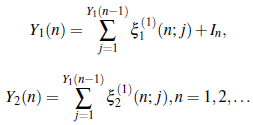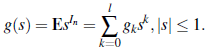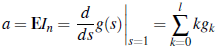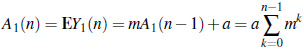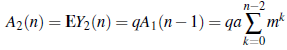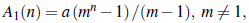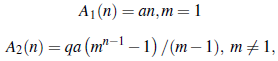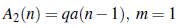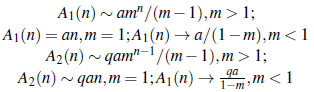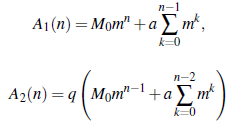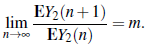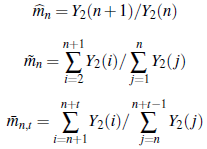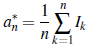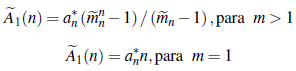1 Introducción
Uno de los temas de salud con mayor tendencia en la actualidad y que ha generado gran impacto a nivel mundial, es el brote de uno de los virus más prolíferos en la historia de la humanidad, el SARS-COV-2, virus cuya tasa de mortandad no es muy alta en comparación con los de otras pandemias que ha enfrentado el ser humano (como la peste negra o la gripe española), sin embargo, la tasa de contagio con la que se transmite este virus de un individuo a otro es muy rápida, y como consecuencia de ello se tienen innumerables casos registrados en casi todos los países alrededor del mundo dejando un alto número de personas fallecidas. En Colombia, el primer caso de contagio se presentó en la ciudad de Bogotá el día 6 de marzo de 2020 con la confirmación de una mujer infectada. Este caso y el de otros individuos reportados como infectados en diferentes partes del país, trajo como consecuencia una rápida propagación del virus en el territorio colombiano. Al día 25 de agosto del 2021, se han reportado más de 1,438,543 casos acumulados de contagios y cerca de 27,356 personas fallecidas a causa del Covid.
Entre los múltiples modelos epidemiológicos, los procesos de ramificación, en particular los modelos de Galton-Watson, han demostrado ser una herramienta sencilla para modelar problemas de esta naturaleza. Si bien, en un principio, el modelo fue propuesto por los matemáticos ingleses Sir Francis Galton (1822-1911) y Henry William Watson (1827-1903), para resolver el problema de la extinción de apellidos aristocráticos, su uso se ha extendido para abordar problemas en otras áreas del conocimiento, tales como, Epidemiología, Biología, Medicina, Química, Economía, Física, Demografía entre otras ciencias [1, 8]. En este trabajo, se hará uso de un procesos de Galton-Watson bitipo para determinar el número esperado diario de individuos contagiados, por un individuo infectado con Covid-19, pero que no son registrados en la base de datos de la Secretaría de Salud de Bogotá, por ser asintomáticos.
En este modelo se asume que el proceso de infección se desarrolla dentro de una población cerrada, esto es, el proceso inicia con un número de infectados dentro de la misma población sin tener en cuenta a individuos que arriban de otras ciudades e incluso de otros países (inmigrantes) y que inciden en la propagación del virus. Por esta razón, en la última sección, se considerará una extensión del modelo anterior teniendo en cuenta la población inmigrante.
2 Modelo de ramificación para los individuos contagiados
Dentro de los individuos infecciosos podemos clasificar a dos tipos de personas; los individuos sintomáticos, que corresponden a aquellas personas infectadas y que han sido confirmadas por una institución prestadora de salud, hay que resaltar que es a partir de estos individuos que se pretende hacer el estudio, es decir, son los datos con los que se hace el proceso de análisis para los individuos no registrados, también cabe mencionar que para estos individuos que poseen síntomas, se asume que el proceso evolutivo de la enfermedad termina en ellos, ya que al ser casos positivos confirmados, estas personas deben ser aislados del resto de la población en el proceso de cuarentena. Por otro lado, están los individuos asintomáticos que son aquellas personas que tienen el virus, pero no saben de su condición ya que no poseen ninguna clase de sintomatología, estos últimos individuos a su vez dan lugar a una nueva serie de contagios que bien pueden ser individuos sintomáticos o individuos asintomáticos.
De acuerdo a la teoria de procesos de ramificación, el objetivo principal de este artículo consiste en construir un modelo que describa el desarrollo del virus Covid-19 en la ciudad de Bogotá con base a los dos tipos de individuos contagiados que se registran día tras día. Un enfoque de proceso de ramificación se considera una aproximación clásica para las epidemias [5], además, aunque el número de personas infectadas es pequeño, se cree que las personas se comportan de forma independiente, por lo que los procesos de ramificación pueden modelar suficientemente las primeras etapas de una epidemia [5, 9]. Con el fin de determinar el número promedio m de individuos infectados, contagiados al día por un individuo infectado pero no registrado, se implementará la metodología desarrollada por Yanev et al (2020) en [11] y [10], teniendo como referencia el número de infectados registrados. Se hará uso de algunos métodos de estimación para determinar el valor de m y se presentan las correspondientes gráficas para cada uno de estos estimadores con el fin de obtener predicciones para analizar el posible comportamiento del valor medio de estas personas contaminadas. Puesto que solamente se tiene disponible el registro de los individuos que han sido reportados como contagiados por las correspondientes EPS, se propone el siguiente proceso de Galton-Watson bi-tipo para describir la situación.
Sea (Z n)n el proceso de Galton-Watson bi-tipo con Z n = (Zi(n),Z 2(n)} donde Z\(n) y Z 2(n) corresponden, respectivamente, al número de tipo T 1 , esto es, individuos contagiados, asintomáticos y no registrados, y al número de individuos contagiados y registrados, denominados individuos tipo T2, presentes en el tiempo n. Se observa que Z 1 (n) es el número total de individuos de tipo T 1 , en el día n, infectados por los individuos contagiados presentes en el día (n - 1) y que Z 2 (n) es el número total de individuos con Covid-19 registrados, presentes en el día n. Se asume que el proceso empieza en Z 1 (0) individuos contagiados. Sean
ξ11(n; j): Número de individuos contagiados tipo T 1 en el día n, infectados por el j-ésimo individuo en el día (n - 1) donde j = 1,2,3..., Z1(n - 1). ξ21(n; j): Número de individuos contagiados registrados con Covid-19 del tipo T2 en el día n, infectados por el j-ésimo individuo contagiado del (n - 1)-ésimo día con j = 1,2,3...,Z 1(n - 1).
El vector de descendencia ξ1 de los individuos de tipo T1 está dado por = ξ 1=( ξ11), ξ21), donde las componentes indican el número total de descendientes de tipo T 1 y de tipo T 2, de los individuos contagiados iniciales. La función generadora de probabilidades conjunta de descendencia de ξ11 y de ξ21 está dada por [11]:
q = 1 - Σ k j=0 Pj , h1(1,1) = 1, con |s1| ≤ 1, |s2| ≤ 1 donde P0 es la probabilidad de que los individuos del tipo T 1 salgan del proceso de reproducción, es decir, individuos que se mejoran o salen del lugar de propagación (por ejemplo, emigran a otro país), P j es la probabilidad de producir j nuevos contaminados del tipo T 1 y q es la probabilidad de que un individuo del tipo T 1 sea confirmado enfermo o muerto. Puesto que los individuos de tipo T 2 no se reproducen ya que son aislados o puestos en cuarentena, entonces la función generadora de probabilidades correspondiente está dada por: h 2 (S 1, S 2 ) ≡ 1.
Las correspondientes funciones generadoras de probabilidades de ξ11 y de ξ21 están dadas por:
Se asume que Z1(0) > 0 y Z2(0) = 0 entonces para n = 1 , 2,…
donde los vectores
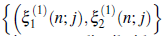 son independientes e idénticamente distribuidos (iid) como (ξ11 ξ21)
son independientes e idénticamente distribuidos (iid) como (ξ11 ξ21)
Puesto que P {ξ21(n; j) = 0} = 1 - q y P {ξ21(n; j) = 0} = q, entonces el proceso Z2(n) tiene distribución binomial de parámetros Z1(n- 1) y q. Esto es,
Es decir que, q puede ser interpretado como la proporción de individuos contagiados registrados dentro del grupo de individuos contagiados en el día (n- 1). Para determinar el número promedio de individuos contagiados pero no registrados en la muestra se hará uso de las funciones generadoras de probabilidades de Z1(n) y Z2(n), siguiendo el procedimiento habitual de acuerdo a [11].
Sean
Sean hn(s1, s2) la función generadora de probabilidades conjunta de ξ11(n; j) y ξ21 (n; j), h*
n: = h
n(s, 1) y
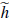 n = hn(1,s). No es difícil probar que
n = hn(1,s). No es difícil probar que
Se tiene entonces que el número promedio de individuos contagiados por un i.c. no reportado es igual a
y que el número promedio de individuos contagiados, no reportados, por un i.c. no reportado es igual a
Se observa que
es el valor medio de los individuos contagiados por un i.c. registrado.
De las ecuaciones dadas anteriormente se obtiene que:
y
donde, m0 = 1 y E(Z2(0)) = 0.
2.1 Estimación de los parámetros
Se observa que:
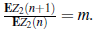 Entonces (ver [3]),
Entonces (ver [3]),
es un estimador empírico del parámetro m, el cual es además un estimador fuertemente consistente [7].
El estimador de máxima verosimilitud, llamado estimador de Harris [7], está dado por:
Por último, se considerará el estimador de Crump-Hove [10] el cuál es dado por:
Se tiene entonces que al estimar el valor de m, se puede predecir el valor esperado de los individuos no reportados en la población de personas contagiadas con Covid-19.
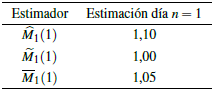
Por lo tanto, con Z1(0) = 1, M1(n) = EZ1 (n) puede ser aproximado respectivamente por
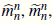
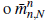 Obteniendo de esta manera los siguientes tres estimadores de M1 (n) :
Obteniendo de esta manera los siguientes tres estimadores de M1 (n) :
Por lo tanto, si se tienen las observaciones (Z2(1),Z2(2),...,Z2(n)) en los primeros n días, se puede predecir el valor medio de los individuos contagiados en los próximos j días por medio de las siguientes relaciones;
Proporción de individuos contagiados registrados (T 2) en la población.
De acuerdo a [10], la proporción estimada de individuos infectados y registrados en el n-ésimo día está dada por:
2.2 Implementación computacional del modelo
Para la parte computacional del modelo, se tomó como base de datos, los informes registrados por la página del portal de datos abiertos del Estado Colombiano [4] de la ciudad de Bogotá, el cual es el único punto de acceso digital del ciudadano a todos los trámites, servicios de información pública y ejercicios de participación, colaboración y control social que ofrecen las entidades del Estado. Los datos tomados son información pública dispuesta para los ciudadanos en formatos que permiten su uso y reutilización, bajo licencia abierta y sin restricciones legales, para su aprovechamiento de acuerdo a la Ley 1712 de 2014 sobre Transparencia y Acceso a la Información Pública Nacional en Colombia. Finalmente, para el procesamiento y análisis de estos registros, se implementó el software Python versión 3.7 a la base de datos suministrada.
El procedimiento de estimación se puede resumir en los siguientes pasos [11]:
• La estimación para cada una de las observaciones Z2(1 ),...,Z2(n) de los nuevos individuos contagiados, Z1 (n), por un individuo infectado es estimado por los estimadores Lotka-Nagaev, Harris y Crump-Hove.
• Para realizar los pronósticos, los valores (fechas) más recientes son utilizados.
• Los valores esperados del número de individuos contagiados no registrados por un i.c. (parámetro de contagio) es calculado para cada uno de los tres estimadores.
• El número de individuos contagiados no registrados Z1(n) es desconocido así como también el valor inicial m0 = EZ1 (n), en vez de estimar m0, se estima M1 (n).
• La proporción α(s + j) con s = 1, 2,..., n, de las personas registradas contagiadas dentro de la población, en el (s + j)-ésimo día es estimada.
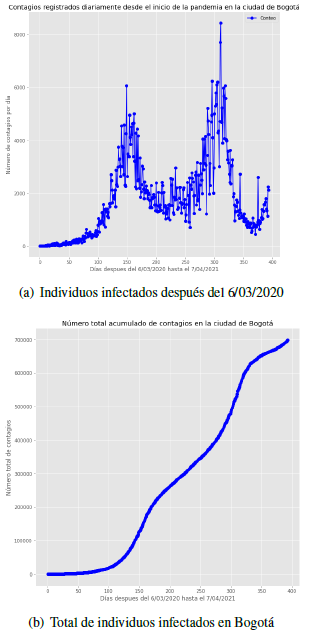
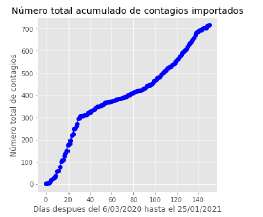
En las figuras 1(a) y 1(b) se muestra la gráfica de los casos positivos registrados diariamente para Covid-19 en la ciudad de Bogotá y el total acumulado respectivamente comenzando desde el 6 de marzo de 2020 (primer contagio registrado) hasta el 7 de abril del 2021 (registro más reciente para este estudio).
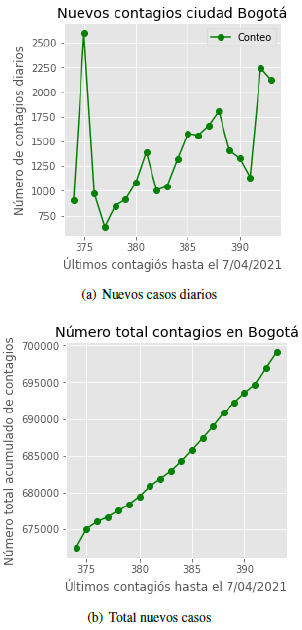
En las figuras 2(a) y 2(b) se presentan, respectivamente, los gráficos de los nuevos casos registrados diariamente y el total acumulado durante los últimos 20 días antes del 7 de abril del 2021 (fecha en la cual se hace el análisis para este trabajo) en la ciudad de Bogotá.
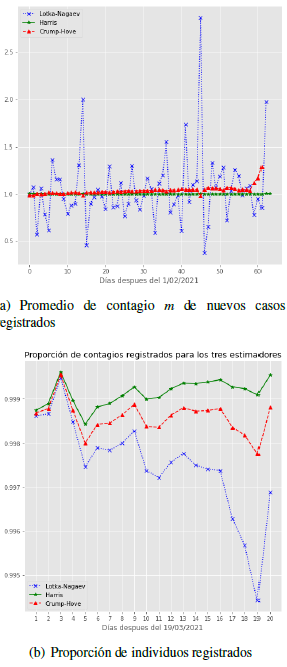
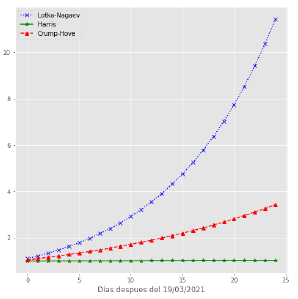
El parámetro de contagio m para los nuevos contagios es estimado para cada uno de los tres estimadores propuestos anteriormente, el comportamiento de este valor se puede apreciar en la figura 3(a). Este valor indica el número esperado de nuevos contagios causados por un individuo contagiado. Estos valores corresponden a los últimos 60 días que sucedieron después del 1 de febrero del año 2021. Los estimadores Harris y Crump-Hove muestran un comportamiento relativamente más estable durante el periodo analizado, es decir que dichos valores no varían mucho a medida que pasan los días.
La figura 3(b) muestra los correspondientes valores para la proporción de casos registrados Z2(n) con Covid-19 dentro del total de la población contagiada (individuos del tipo T1 o T2) para cada estimador, este valor es muy cercano a 1, por lo tanto esto quiere decir que cerca del 99% de los contagios registrados corresponden a personas que se les hizo la prueba, dieron positivo para Covid-19 y corresponden a individuos del tipo T2 (personas sintomáticas). Es importante resaltar que el valor medio de reproducción m permite clasificar el proceso, [8], en tres casos: supercrítico (m > 1), crítico (m = 1) y subcrítico (m < 1). En el caso supercrítico, el crecimiento de la media poblacional es exponencial, en el caso crítico el valor medio de crecimiento es constante y en el caso subcrítico hay un decremento exponencial, este valor tiende a cero a medida que n tiende a infinito.
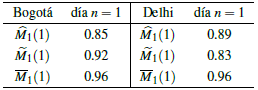
El cuadro 1 representa el valor de la estimación del parámetro de reproducción del virus por un i.c. en el día Z2(1), dato correspondiente al 19 de marzo del 2021. Este valor se obtuvo con base en cada estimador y es uno de los 20 valores del proceso Z2(1),Z2(2),...,Z2(20), siendo este el primero de los 20 casos más recientes en este estudio.
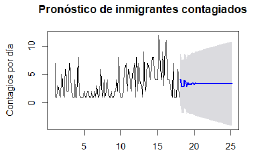
Es importante resaltar que el valor de estas estimaciones determinará el comportamiento asintótico promedio de contagios de la enfermedad para los individuos no registrados (individuos asintomáticos del tipo T1) en los tres casos ya mencionados. Como era de esperarse, en la figura 4 se observa un crecimiento exponencial (caso supercrítico) para el estimador de Lotka-Nagaev, de forma similar se tiene un valor de m (ligeramente supercrítico) para el número esperado de contagios por un i.c. en el estimador de Crump-Hove, el cual tiene un comportamiento asintótico similar. Finalmente, el valor del parámetro de contagio para el estimador de Harris fue mayor a uno, sin embargo, este valor fue muy cercano a uno, lo cual se puede clasificar como un comportamiento crítico y por ende se espera que el crecimiento de contagios por un i.c. crezca de forma constante.
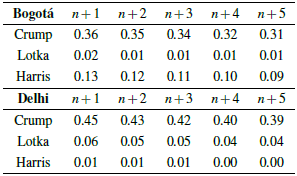
El valor esperado del número de contagios por un i.c. para los individuos no registrados se aprecia en la figura 4 junto con un pronóstico para 5 días del 8 al 12 de abril (cuadro 2) después del día 20 (último día) para cada uno de los tres estimadores propuestos.
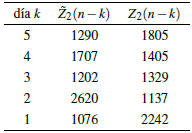
Para medir el comportamiento de este modelo, podríamos comparar el número total de predicciones de individuos contagiados
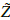 2(n - k) (usando el estimador de Lotka-Nagaev) con el valor real obtenido en las observaciones Z2(n - k) para los últimos 5 días antes del último día mas reciente (7 de abril del 2021), dicha comparación la podemos observar en el cuadro 3.
2(n - k) (usando el estimador de Lotka-Nagaev) con el valor real obtenido en las observaciones Z2(n - k) para los últimos 5 días antes del último día mas reciente (7 de abril del 2021), dicha comparación la podemos observar en el cuadro 3.
Cabe mencionar que la estimación del parámetro m puede ser considerada como una primera etapa hacia un modelo epidemiológico mucho más complejo, por lo tanto, con base a los resultados obtenidos, hasta el momento, se procede a estudiar el caso en el cual se adiciona a este modelo el proceso de inmigración (número de inmigrantes que entran en el proceso evolutivo de la enfermedad), ya que si se suma este proceso al modelo anteriormente analizado, el número de nuevos contagios y el valor medio de reproducción cambian significativamente.
Antes que nada, hay que resaltar algunas diferencias entre los modelos con y sin inmigración, en primer lugar, para ambos casos, cuando el parámetro de contagio m es mayor a uno (caso supercrítico), el crecimiento es exponencial independientemente de si el modelo contempla o no la llegada de inmigrantes, sin embargo, en el caso crítico (m = 1) y en el caso subcrítico (m < 1) el comportamiento asintótico entre estos dos modelos es diferente ([10]), en el caso crítico, el parámetro de contagio m en el modelo con inmigración crece linealmente mientras que, en el modelo sin inmigración el valor medio es constante. En el caso subcrítico, el valor medio de contagio del proceso con inmigración converge a un valor constante positivo pero en el proceso sin inmigración el valor m tiende a cero. De forma general este caso corresponde a un proceso de ramificación con inmigración que se estudiará con más detalle en la siguiente sección.
3 Modelo con Inmigración
En el sección anterior se estudió el caso donde el proceso de transmisión de la enfermedad se transmite dentro de los individuos que conforman una misma población, sin embargo, es importante precisar que en el proceso de infección, los individuos o personas que arriban de otros países también generan una cantidad considerable de contagios, por tal razón es importante tener en cuenta a los individuos inmigrantes que proceden de otros lugares y que también hacen parte del proceso evolutivo de la enfermedad.
Para ello, retomando gran parte de los resultados de la sección anterior, el objetivo será analizar el caso en el cuál se incluye en el modelo, el proceso de inmigración I n el cual determina el número de inmigrantes infectados.
Sean Y 1(n) y Y 2 (n) los procesos de ramificación definidos por:
donde los vectores,
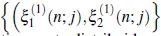 son independientes e idénticamente distribuidos como
son independientes e idénticamente distribuidos como
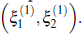
Las funciones generadoras de probabilidad son definidas como en el modelo sin inmigración dado anteriormente y son independientes de {I n}. La función generadora de probabilidad asociada al componente de inmigración es definida como:
g
k
corresponde a la probabilidad de que un individuo inmigrante genere k contagios. Se puede asumir que Yi(0) > 0 es independiente de
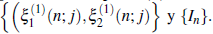
Nótese también que Y 2(0) = 0. Otro posible supuesto es que, Y 1 (0) = Y 2(0) = 0, lo cual implica que, de hecho, el proceso empieza con los primeros inmigrantes reales.
Interpretación de los términos:
• Yi(n): es el número total de individuos del tipo T 1 en el n-ésimo día infectados por los individuos en el (n - 1)-ésimo día más los nuevos inmigrantes reales.
• Y2(n): es el número total de individuos infectados oficialmente registrados (T 2) en el n-ésimo día.
A partir de la función generadora de probabilidad del número de inmigrantes In, se obtiene el valor esperado de inmigración:
Análogamente al modelo sin inmigración, se obtiene que el valor esperado de los procesos Y 1 (n) y Y 2(n) para n = 1, 2,..., están dados por:
y
donde se asume que A1 (0) = E Y1 (0) = 0 y donde los parámetros m y q son definidos como en la sección anterior. Por lo tanto, haciendo el desarrollo de la serie geométrica para cada uno de los casos de m se tiene que:
y
y
Por lo tanto, si n tiende a infinito, estas expresiones, respectivamente, se pueden aproximar por [11]:
Si A1(0) = E Y1(0) = M 0 > 0, se obtiene ([11]):
De nuevo, hay que tener en cuenta que solo se dispone información del proceso de contagio para los casos Y 2(1), Y 2(2),..., Y 2(n), que corresponden a los casos registrados oficiales, por lo tanto, con base a esta información se pueden obtener las correspondientes estimaciones.
3.1 Estimación de los parámetros
De manera similar al caso sin inmigración y asumiendo que m ≥ 1 se puede obtener [3]:
Por lo tanto, para n suficientemente grande podemos considerar (análogamente al caso sin inmigración [10]) los estimadores siguientes:
Estos estimadores corresponden a los ya mencionados en la sección anterior, esto es; el estimador de Lotka-Nagaev, el estimador de Harris y Crump-Hove respectivamente. Si m > 1 podemos determinar el valor medio de los individuos contagiados no registrados dentro de la población. Si asumimos que el proceso inicia con los primeros inmigrantes, entonces A1 (n) = E Y1(n) puede ser aproximado utilizando los estimadores propuestos anteriormente.
3.2 Parámetro de inmigración α
Uno de los principales problemas, en este modelo, es la estimación del parámetro a de inmigración. Si por ejemplo, tenemos información de casos de contagios procedentes de individuos inmigrantes I n los primeros n días, se puede proponer el siguiente estimador para α [10]:
por lo tanto;
De manera análoga se procede con los estimadores
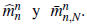 Desafortunadamente, en el caso de Bogotá, no hay información, a partir del 25 de enero del 2021, del número de inmigrantes infectados que ingresaron a la ciudad. Para antes de esa fecha se dispone solamente de información parcial. Por esta razón, fue necesario implementar una metodología de series de tiempo para poder extrapolar la estimación de este parámetro a* hacia la fecha actual en la cual se hizo el estudio (7 de abril del 2021).
Desafortunadamente, en el caso de Bogotá, no hay información, a partir del 25 de enero del 2021, del número de inmigrantes infectados que ingresaron a la ciudad. Para antes de esa fecha se dispone solamente de información parcial. Por esta razón, fue necesario implementar una metodología de series de tiempo para poder extrapolar la estimación de este parámetro a* hacia la fecha actual en la cual se hizo el estudio (7 de abril del 2021).
3.3 Implementación computacional para el modelo con inmigrantes
La implementación del modelo con inmigración se puede resumir en los siguientes pasos:
• La estimación del parámetro de inmigración α n no pudo ser obtenida a través de los datos recopilados ya que habían demasiados datos faltantes, no se tenía información para las últimas 53 observaciones de inmigrantes contagiados, por lo tanto el valor de esa estimación tuvo que ser aproximado y extrapolado a la fecha actual a través de una metodología de series de tiempo, particularmente un modelo ARIMA y un modelo de suavizamiento exponencial de Holt-Winters [2].
• Los datos utilizados para la estimación corresponden al mismo conjunto de datos en el modelo anterior sin inmigración [4].
• El valor promedio de individuos contagiados en este modelo con inmigración es calculado a partir de: A1(n) = M0 m n +
de acuerdo a [10].
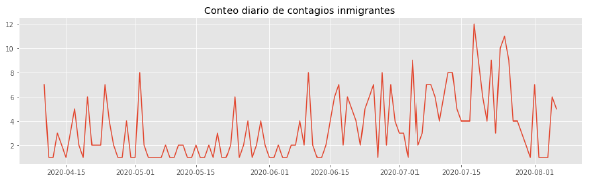
La gráfica de inmigrantes contagiados por día en la ciudad de Bogotá y el total acumulado, puede ser apreciado en las figuras 5 y 6.
Uno de los grandes inconvenientes para este modelo es que los datos correspondientes a individuos inmigrantes solo se encontraban disponibles hasta alrededor del día 151 (gran cantidad de datos faltantes dentro de esta base de datos) y el conjunto de datos para toda la población infectada tiene registros por encima del día 400 desde el inicio de la pandemia en Bogotá, por lo tanto se tiene que extrapolar esa estimación para un horizonte de pronóstico hasta la fecha actual.
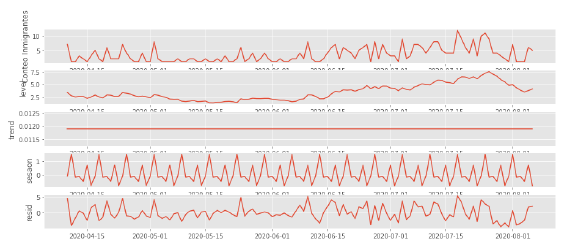
La descomposición de la serie de inmigrantes contagiados en cada una de sus componentes de tendencia, nivel, ruido y estacionalidad se pueden apreciar en la siguiente gráfica (figura 7).
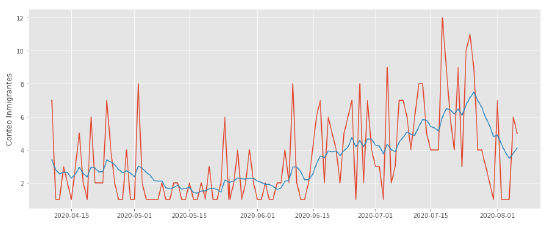
Claramente, como la serie de tiempo tiene un comportamiento que no es estacionario se opta por realizar, en primer lugar, un ajuste por suavizamiento exponencial. El ajuste de acuerdo a la salida del código Python se puede apreciar en la figura 8. El pronóstico arrojado por esta metodología fue un valor aproximado de αn * = 4. 40.
Por otro lado, se realizó un ajuste mediante el enfoque ARIMA a través de la función auto.arima() de la librería forecast en R-Studio cuya gráfica se muestra en la figura 9. De acuerdo a esta función, el mejor modelo con base al criterio de información AIC, fue un modelo ARIMA(0,1,1). El pronóstico para este caso, fue de aproximadamente an = 3.520, por lo tanto este será el valor a extrapolar y con este valor se hará la comparación para el modelo sin inmigración, es importante recordar que este valor representa el valor esperado del proceso estocástico (I k ) k asociado a las personas inmigrantes contagiadas.
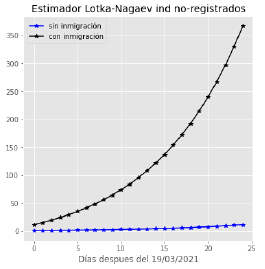
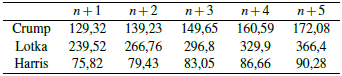
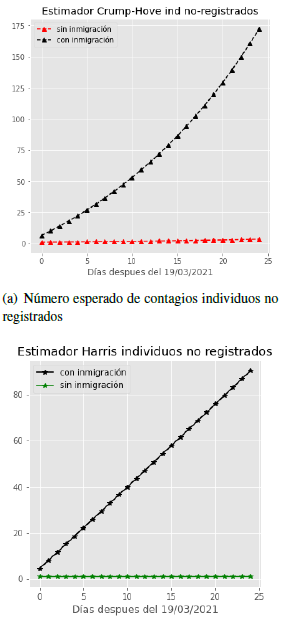
A continuación, se presentan las comparaciones del valor promedio de contagio por un i.c. para los modelos con y sin inmigración, para cada uno de los estimadores anteriormente mencionados (figuras 10, 11(a) y 11(b)). Adicional a ello se hace un pronóstico de este valor para 5 días, los cuales corresponden a las últimas cinco observaciones en cada una de estas gráficas a partir del día 20.
En ambos casos, figuras 10, 11(a), se tiene un crecimiento exponencial supercrítico debido a que el parámetro de contagio fue mayor a uno. Los valores pronosticados del modelo con inmigrantes se muestra en el cuadro 4:
En las figuras 10, 11(a) y 11(b) el comportamiento asintótico de los estimadores, para cada caso, corresponde a lo establecido por la teoría, en primer lugar, los estimadores Lotka- Nagaev y Crump-Hove, al ser valores supercríticos m > 1, el comportamiento asintótico del parámetro de contagio tiene un crecimiento exponencial para ambos casos (con y sin inmigración), sin embargo, para el estimador de Harris, cuyo valor estimado fue muy cercano a uno m = 1, el comportamiento asintótico para el modelo con inmigración es creciente de forma lineal.
4 Comparación del modelo en la ciudad de Delhi para la situación actual
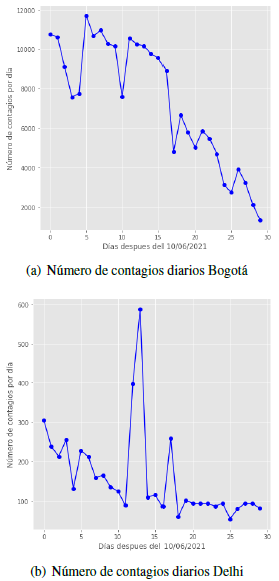
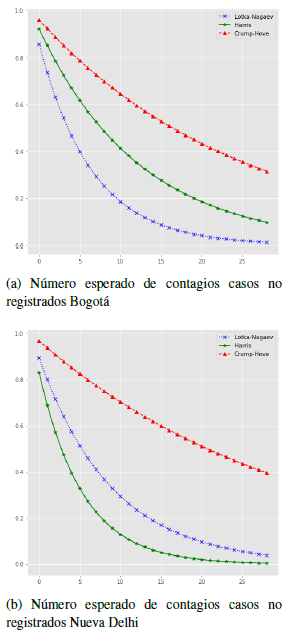
En esta sección, se hará una comparación para la situación actual de contagios desde la fecha del 10 de junio del 2021 entre las ciudades de Bogotá y Delhi de la República de la India. Las figuras 13(a) y 13(b) muestran un comportamiento exponencial decreciente del parámetro de contagio para estas dos ciudades en cada uno de los tres estimadores. Esto coincide con una baja de contagios que se ha venido presentando en ambas ciudades recientemente (figuras 12(a) y 12(b)). En ambos casos, vemos que se presenta un nivel subcrítico ya que para ambas ciudades el parámetro de contagio fue inferior a uno, m < 1. Este comportamiento, de acuerdo a la teoría de procesos de ramificación, corresponde a una eventual extinción del proceso evolutivo de la enfermedad a medida de que pasan los días, en otras palabras, se espera que el proceso de contagio se extinga o tienda a ser cero a medida que transcurran los días en estas dos ciudades.
El valor promedio de individuos no registrados m contagiados por un i.c. es estimado para cada caso. Los valores obtenidos se presentan en la tabla 5 en cada una de las ciudades.
En la tabla 6 se presenta un pronóstico de m para 5 días después del 09/07/2021 en ambas ciudades. Recordemos que este valor indica, en promedio, cuántos contagios de individuos no registrados se espera tener por un solo individuo infectado con Covid-19. Estos valores corresponden a las últimas 5 observaciones de la figura 13.
5 Conclusiones y trabajo futuro
A grandes rasgos, el mayor esfuerzo de este trabajo se centra en dar una estimación al parámetro de reproducción m, esto debido a que la estimación de este valor permite determinar la criticidad del proceso asociado a los individuos asintomáticos de la población. En el proceso sin inmigración, la criticidad del proceso puede ser subcrítica (m < 1) crítica (m = 1) o supercrítica (m > 1), de acuerdo a lo obtenido, el parámetro de reproducción para los estimadores de de Lotka-Nagaev y Crump-Hove fue mayor a uno en ambos casos, supercrítico para el estimador de Lotka-Nagaev y ligeramente supercrítico para el estimador de Crump-Hove, por lo tanto era de esperarse que el comportamiento asintótico del número esperado de contagios por un i.c. fuera creciente de forma exponencial. Por otro lado, el valor de reproducción, obtenido por el estimador de Harris, fue muy cercano a uno, lo cual se considera un caso crítico y como consecuencia se esperaba que el crecimiento asintótico del parámetro de contagio m, a medida que n tiende a infinito, fuera constante. De manera similar, en el caso con inmigración, la estimación de este parámetro también determina el comportamiento asintótico del proceso para cada uno de los estimadores y los resultados obtenidos corresponden a lo establecido por la teoría, en primer lugar, para los estimadores Lotka- Nagaev y Crump-Hove, al ser valores supercríticos, el comportamiento asintótico se esperaba que fuera exponencial para ambos casos (con y sin inmigración). Por el contrario, en la estimación de Harris, al ser un valor muy cercano a uno, el comportamiento asintótico para el modelo con inmigración fue creciente de forma lineal.
A pesar de las ventajas que pueda ofrecer el presente modelo debido a su sencillez, la validez de los resultados dependen de los datos oficiales de la región donde se desee hacer el estudio. En este caso, para la estimación del parámetro de reproducción inmigrante α n , hubo que hacer una extrapolación del valor para la fecha actual en la cual se hizo el estudio, esto a raíz de que no se tenían registros de inmigrantes contagiados a partir del 25 de enero del 2021 y el estudio se hizo para los 20 días más recientes antes del 7 de abril del 2021. A la fecha actual de este análisis, el valor m de reproducción en la ciudad de Bogotá es ligeramente mayor a uno, lo cual corresponde a un crecimiento exponencial y la estimación de este valor puede ser considerada como un primer paso a la elaboración de un modelo epidemiológico mucho mas complejo [11], por ejemplo, un modelo que además de los individuos del tipo T1 y T2 tenga en cuenta otros individuos y variables, entre ellos, el periodo de incubación de la enfermedad, los individuos recuperados, hospitalizados, vacunados, entre otros.
Una de las principales métricas para evaluar la eficacia del modelo corresponde a la estimación del número de contagios por día (por ejemplo vía Harris) comparado con el valor que se obtuvo en la muestra, sin embargo, se podría mejorar este criterio de calidad, analizando la distribución asociada a los individuos del proceso Z1 (n) la cual es desconocida, por ejemplo vía simulaciones, y comparar los valores calculados para el parámetro de contagio obtenidos por medio del proceso conocido Z2(n) para evaluar la eficiencia del modelo presentado frente al modelo desconocido. Otra desventaja presentada en este modelo fue la estimación del parámetro de reproducción asociado a los individuos inmigrantes en vista de que no se tenían registros para muchos de estos casos y a la no estacionariedad de la serie de contagios, se optó por dos metodologías del análisis de series de tiempo, suavizamiento exponencial y un modelo ARIMA, pudiendo optar por otra alternativa (por ejemplo modelos de redes neuronales y aprendizaje profundo para pronósticos de largo plazo, los cuales han demostrado buenos ajustes [6]) sin embargo, la estimación de este parámetro es algo que se podría abordar con más detalle ya que la estimación de este valor es determinante para evaluar la diferencia entre el modelo con y sin inmigración. Como trabajo futuro, esta metodología es útil para estudiar el modelo de ramificación con edad dependiente y factores de comorbilidad aún siguiendo el mismo esquema de procesos de ramificación de Galton-Watson.